Caso I (46XY, dup 15q11.2-q13.1)
3.1. Analisi citogenetica del probando
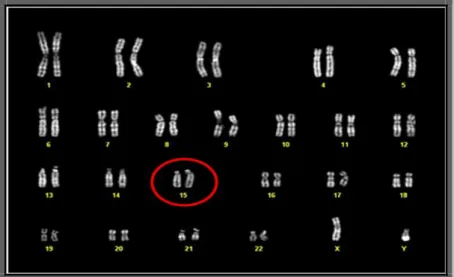
Il caso da noi preso in esame è stato inizialmente sottoposto ad una analisi citogenetica classica con bande G (risoluzione di 320 bande). I risultati ottenuti non hanno mostrato alcuna alterazione cromosomica. La citogenetica classica non consente infatti di evidenziare alterazioni strutturali nell’ordine delle 5-7 Mb, e (nella pratica laboratoristica) la presenza di piccole duplicazioni spesso risulta più difficile da vedere rispetto alle piccole delezioni.
3.2. CGH array
Il DNA del paziente è stato sottoposto al CGH array.
L’analisi è stata effettuata utilizzando vetrini Agilent 4x44, ovvero vetrini su cui sono stati spottati 44000 sonde oligonucleotidiche della lunghezza di circa 60 bp, che coprono l’intero genoma e hanno fra loro una distanza media di circa 30-40kb. Vengono considerate potenzialmente anomale quelle regioni in cui si rilevano almeno 3 sonde consecutive con log del rapporto di fluorescenza intorno a -1 (delezioni) o +0.5 (duplicazione).
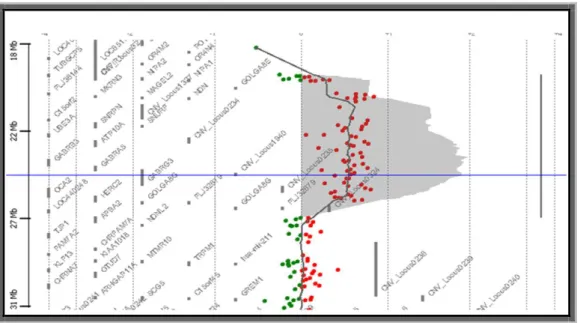
Nella figura riportiamo il grafico CGH array relativo al cromosoma n.15. In alto e’ riportato un ideogramma del cromosoma 15 e le relative bande citogenetiche, mentre nella parte inferiore i valori di fluorescenza relative agli oligo lungo il cromosoma. Come si può notare, nella porzione prossimale del braccio lungo il profilo mostra un evidente scostamento dal valore 0 (normalità) verso un valore intorno a +0.5, indice della presenza di una duplicazione.
Figura 3.2 Ideogramma del cromosoma 15 e profilo del CGH array.
Il programma di analisi fornisce anche la posizione degli oligo che mostrano un profilo anomalo, e in particolare nel nostro caso ha evidenziato una duplicazione che si estende dall’ oligo in posizione 20335887 bp (mappa di riferimento Hg18) all’ oligo in posizione 26199055 bp, per un’estensione di circa 5.8 Mb. Questa regione corrisponde alle bande citogenetiche 15q11.2-15q13.1 e , sempre con riferimento alla mappa Hg18, comprende i seguenti geni : TUBGCP5, CYFIP1,
NIPA2, NIPA1, MKRN3, NDN, SNRPN, UBE3A, ATP10A, GABRB3, GABRB5, GABRG3, OCA2 e HERC2.
Figura 3.3 La zona duplicata del cromosoma 15
L’analisi del CGH –array è stata estesa anche ai genitori, e si è mostrato che la stessa duplicazione è presente nella madre del probando.
3.3. Methylation-specific MLPA (MS-MLPA)
Abbiamo effettuato sul DNA del probando la tecnica MS-MLPA la quale è in grado, in un singolo esperimento, di fornire informazioni relative ad una determinata regione sul numero di copie di alleli e sul profilo di metilazione.
La procedura prevede che ogni campione di DNA sia diviso in due aliquote, la prima viene sottoposta unicamente alla procedura di ligation delle sonde mentre la seconda viene sottoposta dopo la procedura di ligation anche alla digestione enzimatica con HhaI, che riconosce unicamente il DNA non-metilato. Ovviamente per la seconda aliquota l’amplificazione e la successiva identificazione con elettroforesi capillare viene ottenuta solamente se le isole CpG dei siti HhaI sono metilate.
Il DNA del nostro soggetto è stato analizzato in contemporanea con un soggetto controllo ‘normale’; inoltre, l’affidabilità dei risultati è stata valutata anche analizzando i valori forniti dalle 18 sonde di riferimento interno del kit (sonde reference nella tabella). Nella tabella 3.1 riportiamo le sonde utilizzate nell’esperimento. Per ciascuna sonda, riportiamo l’area del picco ottenuta dal controllo (2° colonna), l’area relativa del picco relativo rispetto al totale (3° colonna), l’area del picco ottenuta nel nostro soggetto (4° colonna), l’area relativa (5° colonna), il rapporto fra le aree relative del soggetto rispetto al controllo (6° colonna); l’area del picco nel soggetto digerito con enzima HhaI (7 colonna), l’ area relativa (8 colonna) e il rapporto fra le aree relative (colonna8/colonna 5) (9 colonna).
Per quanto riguarda il numero di copie, si considerano indicativi di duplicazione i valori in colonna 6 quando >1.30; di delezione quando < 0.70.
Nel nostro caso, risulta che i valori sono fortemente indicativi di presenza di duplicazione per le sonde evidenziate in rosso nella tabella. Da notare come invece le sonde reference hanno sempre un valore vicino a 1.
Per quanto riguarda invece i risultati del pattern di metilazione (ottenuti dopo digestione) (colonna 9), ci attendiamo in caso di normalità (una copia materna, una paterna) un rapporto intorno a 0.5; nel caso di disomia uniparentale materna un rapporto di circa 1; nel caso di disomia uniparentale paterna un rapporto intorno allo zero; nel caso di duplicazione paterna un rapporto di circa 0.33 e nel caso di duplicazione materna un rapporto all’incirca di 0.66. Nel nostro caso le sonde duplicate hanno un valore intorno a 0.64, suggerendo la duplicazione materna.
CONTROLLI PROBANDO SONDE AREA PICCO AREA RELATIVA AREA PICCO AREA RELATIVA AREA RELATIVA PROBANDO/AREA RELATIVA CONTROLLO reference 16878 0,06 24336 0,05 0,92 CYFIP1 18401 0,06 25010 0,05 0,87 SNRPN 17408 0,06 38170 0,08 1,40 reference 10945 0,04 16299 0,03 0,95 TUBGCP5 15311 0,05 22075 0,05 0,92 UBE3AEX9 16322 0,05 30121 0,06 1,18 SNRPNEX1 16185 0,05 37073 0,08 1,47 MKRN3EX1 11023 0,04 25284 0,05 1,47 MAGEL 11670 0,04 21289 0,05 1,17 UBE3AEX1 13939 0,05 29390 0,06 1,35 SNRPN 19659 0,07 48851 0,10 1,59 MAGELEX1 15823 0,05 39515 0,08 1,60 APBA2 13786 0,05 20699 0,04 0,96 MKRN3EX1 16065 0,05 35176 0,07 1,40 GABRB3EX9 11740 0,04 24648 0,05 1,34 reference 19335 0,06 31910 0,07 1,06 reference 23634 0,08 34740 0,07 0,94 SNRPNEXU1B 23670 0,08 49316 0,10 1,33 SNRPNEXU3 14054 0,05 30137 0,06 1,37 reference 12168 0,04 17640 0,04 0,93 reference 27694 0,09 41720 0,09 0,96 reference 18679 0,06 29844 0,06 1,02 SNRPNEX3 17465 0,06 40491 0,09 1,48 reference 15359 0,05 23718 0,05 0,99 reference 18233 0,06 28253 0,06 0,99 NDNEX1 11915 0,04 13007 0,03 0,70 reference 21559 0,07 35607 0,08 1,06 reference 21033 0,07 31958 0,07 0,97 reference 12352 0,04 13924 0,03 0,72 UBE3AEX1 11629 0,04 27517 0,06 1,51 ATP10AEX1 16123 0,05 37468 0,08 1,49 reference 19065 0,06 28893 0,06 0,97 ATP10AEX2 10313 0,03 25210 0,05 1,56 reference 16122 0,05 29846 0,06 1,18 UBE3AEX1 10069 0,03 32137 0,07 2,04 GABRB3EX1 16784 0,06 40607 0,09 1,55 NDNEX1 16064 0,05 35069 0,07 1,40 reference 14253 0,05 21490 0,05 0,96 OCAEX22 8901 0,03 21543 0,05 1,55 OCAEX1 15432 0,05 36475 0,08 1,51 reference 15236 0,05 25056 0,05 1,05 reference 9194 0,03 19160 0,04 1,33 reference 9693 0,03 16590 0,04 1,10 TOTALE 301432 470984
DIGESTIONE
SONDE AREA PICCO AREA RELATIVA RAPPORTO AREE RELATIVE
reference 19716 0,06 1,13 CYFIP1 21142 0,06 1,17 SNRPN 20199 0,06 0,73 reference 0 0,00 0,00 TUBGCP5 16905 0,05 1,06 UBE3AEX9 26937 0,08 1,24 SNRPNEX1 18880 0,06 0,71 MKRN3EX1 19060 0,06 1,05 MAGEL 17772 0,05 1,16 UBE3AEX1 0 0,00 0,00 SNRPN 27625 0,08 0,79 MAGELEX1 31554 0,09 1,11 APBA2 19045 0,06 1,28 MKRN3EX1 24297 0,07 0,96 GABRB3EX9 20704 0,06 1,17 reference 22324 0,07 0,97 reference 28770 0,08 1,15 SNRPNEXU1B 21510 0,06 0,61 SNRPNEXU3 23383 0,07 1,08 reference 14471 0,04 1,14 reference 34725 0,10 1,16 reference 23361 0,07 1,09 SNRPNEX3 35484 0,10 1,22 reference 19621 0,06 1,15 reference 22708 0,07 1,12 NDNEX1 6474 0,02 0,69 reference 28355 0,08 1,11 reference 24553 0,07 1,07 reference 0 0,00 0,00 UBE3AEX1 22095 0,07 1,12 ATP10AEX1 0 0,00 0,00 reference 22358 0,07 1,07 ATP10AEX2 18700 0,06 1,03 reference 25536 0,08 1,19 UBE3AEX1 0 0,00 0,00 GABRB3EX1 0 0,00 0,00 NDNEX1 12583 0,04 0,50 reference 17537 0,05 1,13 OCAEX22 18547 0,05 1,20 OCAEX1 0 0,00 0,00 reference 20230 0,06 1,12 reference 0 0,00 0,00 reference 14909 0,04 1,25
Dall’analisi con MS-MLPA possiamo concludere che il nostro campione risulta aver una duplicazione delle sonde relative ai loci SNRPN,MKRN3EX1, UBE3AEX1,
MAGELEX1, GABRB3EX9, GABRB3EX1, SNRPNEXU1B, SNRPNEXU3,
SNRPNEXU3, ATP10AEX1, ATP10AEX2, OXAEX22 E OXAEX1, in accordo con
quanto verificato con array CGH.
Dal profilo con MS-MLPA la duplicazione parrebbe di origine materna.
3.4. Analisi con i microsatelliti
Per confermare l’estensione della duplicazione e verificarne l’origine parentale, abbiamo effettuato una analisi molecolare della regione 15q11-q13 mediante tipizzazione di marcatori altamente polimorfici (STRs).
Abbiamo effettuato una PCR (Polymerase Chain Reaction) utilizzando primers specifici per i microsatelliti da noi scelti. I prodotti della PCR sono stati successivamente caricati in un sequenziatore automatico e quindi analizzati con il programma GeneScan che ci fornisce informazioni sulla posizione dei picchi e sulla loro relativa ampiezza. I risultati ottenuti sono illustrati nella tabella 3.2.
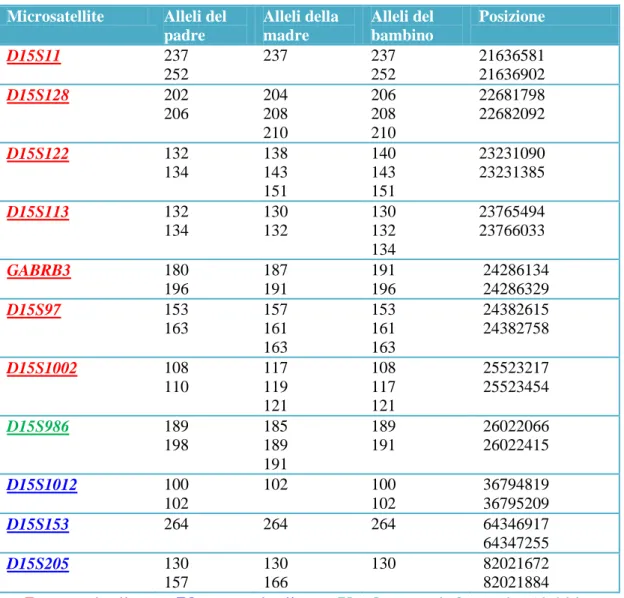
Tabella 3.2. Risultati dell’analisi con i microsatelliti del DNA effettuata sul caso
Microsatellite Alleli del padre Alleli della madre Alleli del bambino Posizione D15S11 237 252 237 237 252 21636581 21636902 D15S128 202 206 204 208 210 206 208 210 22681798 22682092 D15S122 132 134 138 143 151 140 143 151 23231090 23231385 D15S113 132 134 130 132 130 132 134 23765494 23766033 GABRB3 180 196 187 191 191 196 24286134 24286329 D15S97 153 163 157 161 163 153 161 163 24382615 24382758 D15S1002 108 110 117 119 121 108 117 121 25523217 25523454 D15S986 189 198 185 189 191 189 191 26022066 26022415 D15S1012 100 102 102 100 102 36794819 36795209 D15S153 264 264 264 64346917 64347255 D15S205 130 157 130 166 130 82021672 82021884 Rosso= duplicato Blu= non duplicato Verde= non informativo/dubbio
Per ciascun microsatellite è stata verificata la segregazione allelica da genitore a figlio.
La duplicazione dell’allele materno è stata riscontrata per i microsatelliti D15S97, D15S122, D15S113, GABRβ3, D15S11, D15S128 e D15S1002.
Il marcatore D15S986 risulta essere non informativo.
I microsatelliti D15S986, D15S1012, D15S153, D15S205 al di fuori della regione critica presentano solo un allele paterno e uno materno e sono quindi indicativi dell’assenza di duplicazione.
Caso II (46XX,inv dup(15) (p13
cen::cen p13)
3. Analisi citogenetica del probando
Anche per il paziente II è stata eseguita una analisi di citogenetica classica con bande G (risoluzione di 320 bande).
I risultati ottenuti hanno mostrato la presenza di un marker sovrannumerario in 100 metafasi analizzati.
3. FISH (Ibridazione in Situ a fluorescenza)
E’ stata utilizzata la sonda centromerica del cromosoma 15 che ha mostrato 2 segnali a livello del marker suggerendo che è un derivativo isodicentrico del 15. Mentre il segnale era presente sui cromosomi 15 normali mancava sul marker evidenziando l’assenza della regione critica. E’ stata eseguita anche la Fish con le sonde del PWS/AS ed è risultata negativa.
3. CGH array
Per il CGH array è stato utilizzato un 44K oligo-array (Agilent Tecnologies, Santa Clara, California) che ha escluso la presenza di duplicazioni nel braccio lungo del cromosoma 15; nessuna duplicazione o delezione era presente in altri cromosomi analizzati con una risoluzione di circa 100Kb.
CASO III (47,XX, +der(15)t(7;15)(q36;q11.2)
Il caso tre è giunta alla nostra attenzione in seguito all’analisi del liquido amniotico della sorella La sorella della probanda aveva fatto l’amniocentesi per età e presentava una traslocazione tra il cromosoma 15 e il cromosoma 7. Di seguito è stato fatto il cariotipo ai genitori e la madre presentava la stessa traslocazione. Di conseguenza è stata analizzata la sorella laquale però presentava un marker sovrannumerario.
3. Analisi citogenetica del probando
3. FISH (Ibridazione in Situ a fluorescenza)
3. CGH array
A livello del braccio lungo il profilo mostra un evidente scostamento dal valore 0
(normalita’) verso un valore intorno a +0.5, indice della presenza di una duplicazione che si estende dall’oligo 156.123.854 all’oligo 158.781.397 bp per un estensione di circa 2MB.
Presenza di una microduplicazione del cromosoma 15 che si estende dall’oligo 20.335.887 all’oligo 25.979.180 per un estensione di circa 5MB.
Ci aspettiamo che abbia un fenotipo patologico in quanto comprende la regione PWSAR. Avendo un pezzo del cromosoma 7 è un caso particolare rispetto agli altri che presentano solo un marker del cromosoma 15.