19
2.
PROBLEMATICHE
RELATIVE
ALL’USO
DI
RNA
I
2.1.
Problematiche relative ad effetti collaterali dannosi e
loro eliminazione
2.1.1. Effetti al di fuori del bersaglio dipendenti dalla sequenza di base
Come precedentemente sottolineato, il filamento guida del siRNA potrebbe funzionare come un miRNA quando contiene una sequenza di
base che si combina con le regioni 3′UTR dell’mRNA, ma, allo stesso tempo, potrebbe condurre ad OTE dannosi attraverso la repressione della traduzione o la degradazione di mRNA.
È stato dimostrato che tali OTE non si verificano se il filamento guida si accoppia con l’”open reading frame” (ORF) dei trascritti di
mRNA; dunque evitando che i siRNA di base si combinino con 3′UTR dell’mRNA, e questo può essere ottenuto utilizzando algoritmi “on-line”
che ricercano 3′UTR, potenzialmente si potrebbero ridurre, ma non necessariamente eliminare, gli OTE dannosi.
20
È chiaro che devono essere effettuati studi preclinici estremamente accurati della vitalità e della funzione cellulare. Si può per il momento affermare che ad oggi non sono stati riportati casi di OTE dannosi dalla sequenza di base nei “trials” preclinici o clinici precoci di siRNA.
2.1.2. Evitare la risposta dell’interferone
Un aspetto cruciale, come accennato in precedenza, sull’impiego terapeutico, e quindi sicuro, di RNAi è rappresentato dall’attivazione dell’immunità cellulare innata, derivante dall’introduzione di agenti estranei: è importante dunque che venga evitata (7).
Recentemente, sono stati compiuti importanti progressi nella comprensione del meccanismo di risposta dell’interferone ad agenti patogeni estranei ed agli acidi nucleici, consentendo di ottenere significative informazioni proprio per la progettazione di siRNA che eludano l’attivazione di questa via.
Gli interferoni di tipo I sono attivati a livello delle cellule dendritiche plasmocitoidi quando sono presenti nei siRNA esogeni motivi della specifica sequenza immunostimolatoria.
Precedenti studi avevano suggerito che solo RNA a doppio filamento più lunghi di 30 coppie di basi potevano scatenare la risposta
21
dell’interferone, generata attraverso l’attivazione della chinasi PKR e la cessazione dell’espressione genica nel suo complesso.
Tuttavia, numerosi studi hanno ormai messo in evidenza che i recettori tipo Toll (TLR), espressi negli endosomi, sono capaci di riconoscere gli RNA sia a singolo che a doppio filamento, inducendo proprio una risposta dell’interferone e scatenando la traslocazione dei fattori di trascrizione, fattore nucleare-κB (NF-κB), fattore regolatore dell’interferone (IRF) e fattore 2 attivante la trascrizione (ATF2), all’interno del nucleo. È stato dimostrato che TLR3, che riconosce l’RNA a doppio filamento, e TLR7 e TLR8, che riconoscono invece quello a singolo filamento, sono in grado di mediare le risposte dell’interferone sia
in vivo che in colture di cellule eucariotiche tramite il riconoscimento dei
siRNA incorporati per via endosomica.
Questi TLR fungono dunque da sensori per il riconoscimento di specifici motivi delle sequenze immunostimolatorie: tali sequenze, come 5′-GUCCUUCAA-3′ e 5′-UGUGU-3′, dovrebbero pertanto essere eliminate nella progettazione dei siRNA.
Da un punto di vista pratico ed applicativo, i siRNA sintetizzati chimicamente risultano preferibili rispetto ai siRNA trascritti ad opera della RNA polimerasi del batteriofago T7, che lasciano i trifosfati in 5’ che inducono la produzione dell’interferone di tipo I. Le estremità piatte di
22
processazione di Dicer risultano anch’esse immunostimolatorie e sono riconosciute dalla RIG-I elicasi codificata dal gene I sensibile all’acido retinico. La risposta del RIG-I (nota come DDX58) citoplasmatico è distinta dalla risposta dell’interferone mediata da TLR, che richiede il rilascio di siRNA tramite la via endosomiale (Figura 4).
Figura 4. Effetti immunostimolatori dell’RNA interferente.
Gli RNA a doppio filamento più lunghi di 30 coppie di basi attivano protein chinasi R (PKR) e inducono la risposta dell’interferone, così come le estremità appiattite, i piccoli RNA interferenti (siRNAs), che sono rivelati da RIG-I. I siRNA che sono incorporati negli endosomi possono attivare recettori Toll (TLRs), questo dipende dalla presenza di motivi immunostimolatori così da indurre la risposta dell’interferone di tipo I attivando i fattori di trascrizione come NFkB a livello nucleare, il fattore 2 attivante la trascrizione (ATF 2) e il fattore di regolazione dell’interferone (IRF).
23
Sono state individuate diverse specie cellulari in cui è possibile evitare l’attivazione dell’immunità cellulare innata e le risposte dell’interferone mediante l’impiego di shRNA espressi.
Le cellule dendritiche plasmocitoidi derivate dalla cellula progenitrice CDR34+ mostrano una risposta immunitaria quando transfettate con siRNA immunostimolatori attraverso il rilascio mediato da lipidi, che richiede l’incorporazione endosomiale; al contrario sequenze identiche non sono in grado di indurre l’IFN (interferone) di tipo I quando espresse come shRNA da parte del promotore della polimerasi III nell’ambito di un vettore lentivirale integrato.
Dunque, evitare la risposta immune, attraverso l’uso di shRNA espressi, potrebbe limitare la processazione ad opera della via endogena dell’RNAi, e conseguentemente eliminare le interazioni con TLR ed il riconoscimento attuato da RIG-I (2).
2.1.3. Prevenzione della saturazione delle vie di silenziamento endogene
Sebbene siano stati riportati un considerevole numero di studi relativi al silenziamento specifico di geni bersaglio tramite shRNA espressi senza effetti nocivi a livello della vitalità cellulare, recenti studi hanno
24
mostrato che un’abbondante espressione di shRNA può risultare tossica per le cellule.
Uno studio recente ha messo in evidenza come l’espressione di shRNA ad elevate concentrazioni, mediante il promotore U6 in vettori associati ad adenovirus (AAV), determini la saturazione dell’esportina 5 e la successiva inibizione dell’esportazione nucleare dei pre-miRNA endogeni; in uno studio su animali, proprio questo fenomeno rappresentò il fattore letale per 23 topi su 49. Lo stesso studio ha dimostrato, invece, che la somministrazione di dosi più basse di vettori AAV che esprimono shRNA non solo non risulta letale, ma, in particolare, fornisce un efficace e prolungato intervento terapeutico nei confronti del virus dell’epatite B (HBV).
Altri studi effettuati su linfociti primari umani hanno dimostrato che i shRNA guidati dal promotore U6 producevano citotossicità, mentre l’utilizzo di un debole promotore H1 per l’espressione degli stessi shRNA sembrava abrogare l’effetto tossico.
In conclusione, un ruolo significativo nello studio delle applicazioni in campo terapeutico è attribuito all’ottimizzazione dei livelli di espressione di shRNA.
25
2.2.
Problematiche relative alla veicolazione in vivo
2.2.1. Veicolazione con agenti non virali di agenti che inducono RNAi
La veicolazione cellulo-specifica di siRNA in vivo rappresenta il principale aspetto da prendere in considerazione nello sviluppo di un efficace agente terapeutico basato su RNAi.
Numerosi sono gli ostacoli che i siRNA possono incontrare nel tentativo di entrare in contatto diretto con la cellula. Ad esempio i siRNA duplex, essendo polimeri caricati negativamente, risultano incapaci di penetrare facilmente le membrane cellulari idrofobiche senza l’assistenza di carriers come i liposomi o nanoparticelle.
Inoltre, enzimi quali le RNAasi sieriche, intervengono degradando rapidamente siRNA non protetti e non modificati.
Dunque, fondamentali risultano le modificazioni chimiche con funzione stabilizzante, cioè tutte quelle alterazioni che possono essere impiegate per aumentarne l’emivita e la loro funzionalità in vivo.
Metodi di veicolazione non virali includono la veicolazione sistemica non selettiva e quella cellulo-specifica che dovrebbe ridurre gli effetti collaterali, arrestandone il rilascio non richiesto in quelle cellule non scelte come bersaglio.
26
2.2.1.1. Modificazioni chimiche volte ad aumentare la stabilità di siRNA
Alla luce di quanto appena esposto, risultano particolarmente utili
modificazioni chimiche a livello della posizione 2′ del ribosio, comprese le 2′-O-metilpurine e le 2′-fluoropirimidine, in quanto portano ad un aumento della stabilità dei siRNA; infatti tali alterazioni strutturali sono in grado di generare resistenza nei confronti dell’attività delle RNAasi a livello sierico (Figura 3a). A dimostrazione di ciò, uno studio recente, ha individuato una protezione maggiore nei confronti dell’infezione in vivo con HBV da parte dei siRNA 2′-O-metil-modificati, se confrontati con gli analoghi non modificati di siRNA. Si tratta, tuttavia, di un processo delicato; infatti il posizionamento di queste modificazioni può risultare anche negativo,
come nel caso di alterazioni dell’estremità 5′ del filamento guida che possono addirittura influenzare negativamente la potenza del silenziamento.
Recenti studi hanno in realtà dimostrato che le metilazioni
dell’ossigeno in posizione 2′, scaglionate tra i due filamenti di siRNA, sembrano fornire un bilancio ottimale tra il mantenimento della potenza del RNAi e la protezione contro la degradazione. Infine, ma non meno
27
significativo, la metilazione in posizione 2′ produce un ulteriore beneficio; l’uso di siRNA modificati evita l’induzione dell’interferone.
2.2.1.2. Veicolazione sistemica non selettiva di siRNA
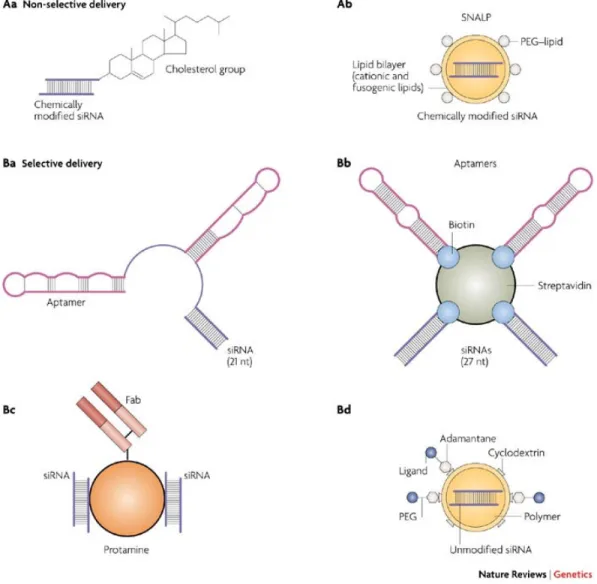
Un efficace metodo di veicolazione sistemica non selettiva di siRNA riguarda l’iniezione intravenosa di siRNA chimicamente modificati o coniugati con il colesterolo o immagazzinati all’interno di particelle liposomiali protettive (figura 5).
28
Figura 5. La parte sinistra mostra varie strategie di veicolazione in vivo di farmaci basati su siRNA sia veicolazione specifica che non specifica. La parte destra mostra un diagramma schematico di espressione di shRNA partendo da DNA utilizzando promotori della Pol III o Pol II. Queste unità di espressione di shRNA possono essere veicolate attraverso metodi virali o non virali al tessuto bersaglio, come illustrato, dal trasferimento genico utilizzando la tecnologia del vettore lentivirale.
(da Lars Aagaard and John J.Rossi. Science Direct-Advanced Drug Delivery Reviews. (2007)
29
Questa forma non selettiva di veicolazione sistemica risulta particolarmente adatta per alcuni tipi di tessuti come il fegato ed il digiuno, ma non lo è per la veicolazione/rilascio in altri specifici tipi cellulari.
I gruppi di colesterolo legati chimicamente all’ossidrile in 3′ del filamento di siRNA “passenger” facilitano l’”uptake” cellulare del siRNA attraverso il processo di endocitosi mediata recettorialmente (Figura 5-6a).
30
Figura 6. Veicolazione di piccoli RNA interferenti.
a) Approcci non selettivi. Gruppi di colesterolo possono essere legati a piccoli RNA interferenti (siRNAs) modificati chimicamente per la loro veicolazione sistemica (Aa). I siRNA possono anche essere veicolati sistematicamente da particelle formate da lipidi e acidi nucleici (SNALPs) (Ab).
b) Approcci selettivi. Chimere formate da aptameri e siRNA fanno sì che i siRNA possono essere veicolati a specifici tipi cellulari che hanno recettori sulla superficie, per gli aptameri e siRNAs (Ba e Bb). Un approccio basato interamente su RNA può essere usato per accoppiare aptameri e siRNAs (Ba), oppure un approccio biotina-streptadiviva può essere usato per raggiunge l’accoppiamento (Bb). La catena pesante dei frammenti anticorpali (Fabs) e i siRNAs possono essere legati con protamine per veicolare siRNAs a specifici recettori sulla superficie cellulare (Bc). Nanoparticelle che hanno sulla loro superficie specifici ligandi possono essere usati per direzionare i siRNA a particolari tipi cellulari (Bd).
31
Una ricerca recente ha illustrato come questo approccio sia stato impiegato con successo per il rilascio di siRNA all’interno del fegato o del digiuno in seguito a somministrazione sistemica nei topi. Lo studio si basava sull’uso di siRNA diretti contro la apolipoproteina B (ApoB) per modificare il metabolismo del colesterolo. Fu riscontrata una riduzione superiore al 50% nell’mRNA di ApoB a livello epatico ed una riduzione del 70% a livello intestinale; inoltre fu osservato un abbassamento dei livelli di colesterolo complessivo. Questi risultati hanno dunque dimostrato che siRNA rilasciati sistemicamente possono essere veicolati ai tessuti mediante la coniugazione con specifiche molecole che promuovono l’”uptake” cellulare.
Per ottenere il rilascio sistemico di siRNA sono stati impiegati siRNA chimicamente stabilizzati e incapsulati in doppi strati lipidici specializzati, noti come particelle stabili acido nucleico-lipide (SNALP). Il doppio strato delle SNALP è costituito da lipidi cationici e neutri, che sono rivestiti in superficie da uno strato di polietilenglicole idrofilico (PEG) (Figura 6a). Un importante studio relativo al rilascio sistemico di siRNA usando SNALP ha evidenziato un’efficace risposta terapeutica dell’RNAi in primati non umani. In scimmie Cinomolgus, una singola dose di siRNA veicolato da SNALP ha abbassato i livelli di ApoB, delle lipoproteine a bassa densità e del colesterolo per 11 giorni o più, con meno del 10% dei livelli normali del mRNA di ApoB a livello epatico. L’effetto
32
del silenziamento è risultato inoltre più potente di quello osservato in studi su topo precedentemente descritti e non è stata osservata alcuna tossicità.
Sebbene debba essere ancora chiarito se possano derivare OTE da un rilascio non specifico di siRNA ad altri tessuti e/o tipi cellulari in conseguenza di ripetute somministrazioni, il metodo di rilascio/veicolazione di SNALP è stato considerato sicuramente efficace in
vivo per “targets” terapeutici espressi a livello epatico.
2.2.1.3. Veicolazione sistemica selettiva di siRNA
Un aspetto chiave da tenere in considerazione ai fini di un impiego terapeutico per gli RNAi riguarda il dosaggio dei siRNA che deve essere somministrato per ottenere l’efficacia desiderata in vivo. Infatti, è da evidenziare come uno svantaggio del rilascio sistemico non selettivo sia rappresentato dalla notevole quantità di siRNA richiesta per ottenere il silenziamento di un gene in vivo. Il rilascio sistemico con “target” selettivo dei siRNA verso recettori specifici localizzati sulla superficie cellulare risulta conveniente a bassi dosaggi, che oltretutto riducono i potenziali OTE nei tessuti non bersaglio.
Da un punto di vista applicativo, siRNA terapeutici sono stati accoppiati con frammenti di anticorpi ed aptameri oppure impacchettati in
33
nanoparticelle rivestite con ligandi che hanno come bersaglio i recettori. Queste strategie di rilascio cellulo-specifiche facilitano l’incorporazione dei siRNA tramite l’endocitosi (figura 5).
Frammenti anticorpali Fabs, specifici per la glicoproteina (gp120) del mantello di HIV, sono stati impiegati per ottenere un rilascio selettivo di siRNA in cellule infettate con il virus dell’HIV sia in coltura che in vivo (Figura 6b).
Le molecole Fabs sono state coniugate con la protamina caricata positivamente, che è in grado di interagire elettrostaticamente con molecole di siRNA caricate negativamente. Per monitorare la specificità nei confronti del bersaglio costituito dalle cellule infettate è stata utilizzata una marcatoria fluorescente. I complessi siRNA-Fab che hanno come obiettivo l’mRNA dell’HIV codificante per la proteina antigenica gruppo specifica (Gag) sono stati incorporati con successo in linfociti T in coltura infettati con HIV-1, determinando una riduzione superiore al 70% nei livelli di p24Gag.
Anche il recettore per il fattore di crescita ErbB2 è stato efficacemente reso bersaglio di coniugati Fab-siRNA in linee cellulari di tumore mammario, evidenziando così l’utilità di questo approccio per il rilascio cellulo-specifico del siRNA.
È possibile progettare ligandi strutturati di RNA noti come aptameri che possono legarsi con specifici recettori della superficie cellulare e
34
coniugarli covalentemente a siRNA per il rilascio cellulo-specifico in vivo. Le molecole di siRNA vengono rilasciate dall’aptamero durante il processo di ingresso all’interno della cellula.
In un altro studio, aptameri che si legano specificamente ad un antigene di membrana specifico della prostata (PSMA), che è espresso sulla superficie delle cellule neoplastiche prostatiche, sono stati coniugati a siRNA ed hanno facilitato la riduzione della crescita tumorale in un modello murino dopo iniezioni intratumorali effettuate ogni 2 giorni per un periodo di 20 giorni. Ancora, impiegando un approccio basato sugli aptameri, una recente ricerca, che aveva come obiettivo le cellule che esprimono PSNA, ha utilizzato siRNA ed aptameri biotinilati, legati insieme in modo non covalente mediante la proteina streptadiviva (Figura 6b). iRNA 27 mers marcati con biotina sono stati incorporati in questi complessi di aptamero al fine di indurre una risposta dell’RNAi più potente, incorporando il passaggio del Dicer nella fase di caricamento ed attivazione del RISC.
Le nanoparticelle rappresentano un ulteriore potente approccio per il rilascio sistemico selettivo di molecole che inducono RNAi. Questi veicoli sono stati studiati e progettati per trasportare grandi carichi protetti di siRNA alle cellule bersaglio, in maniera estremamente specifica. In particolare, l’indirizzamento al bersaglio è garantito da un rivestimento sulla superficie delle nanoparticelle costituito da ligandi cellulo-specifici.
35
In un interessante studio, che aveva come bersaglio in vivo il sarcoma di Ewing, è stato sperimentato questo sistema impiegando nanoparticelle rivestite con ligandi della transferrina. Queste nanoparticelle, costituite da policationi contenenti ciclodestrine (CDP), erano stati progettati per incorporare siRNA caricati negativamente. Inoltre, polimeri di PEG erano stati condensati alla superficie esterna di queste nanoparticelle attraverso gruppi terminali di adamantano per ottenere una maggiore stabilità e per prevenire una possibile aggregazione, fenomeno che senza dubbio ne limiterebbe l’efficacia in vivo. Le catene adamantano-PEG erano dunque legate covalentemente ai ligandi della transferrina ed inoltre le catene adamantano-PEG e il CDP lineare erano state progettate per garantire l’autoassemblaggio in nanoparticelle di dimensioni uniformi (circa 50 nm di diametro), per un rilascio sistemico selettivo (Figura 6b). La ricerca ha permesso di osservare un’inibizione della formazione della neoplasia in un modello murino xenografico in seguito a svariate iniezioni di nanoparticelle che trasportavano siRNA diretto contro il gene di fusione
36
2.2.2. Veicolazione mediante virus di agenti che inducono RNAi
Il problema principale delle malattie croniche è che esse richiedono un trattamento a lungo termine con RNAi, il quale deve essere veicolato da vettori di espressione virale. Questo coinvolge un approccio di terapia genica per incorporare shRNA transgenici all’interno di cellule per l’integrazione genomica o per l’espressione episomiale. Lo sviluppo dei vettori virali è stato pertanto ipotizzato come strategia alternativa per alcune patologie (figura 5).
Diversi vettori per la terapia genica sono stati sviluppati nelle due decadi precedenti. I shRNA sono stati incorporati in vettori plasmidici allo scopo di guidare la loro espressione in vivo. Sebbene ciò sia stato realizzato anche con cellule di mammifero, in cellule neuronali e cellule emopoietiche, sono state incontrate non poche difficoltà. Infatti, il rilascio
in vivo di questi plasmidi e l’integrazione dei loro transgeni non sempre
hanno fornito risultati positivi né per l’analisi transgenica né per le terapie basate su shRNA-plasmide (3).
Le problematiche concernenti la sicurezza dell’uso dei vettori virali si applicano anche alle terapie basate sul siRNA. Problematiche vitali sono ancora la direzionalità dei vettori virali a specifici tipi cellulari e la minimizzazione della loro tossicità.
37
L’impiego di vettori virali costituisce una metodica facile da utilizzare e che permette di transfettare in modo efficiente, tanto che lo sviluppo di tali vettori, negli ultimi anni, ha permesso di utilizzarne ampiamente alcuni (lentivirus e retrovirus). In particolare, come dimostrano alcuni studi recenti, i vettori virali, attraverso un’integrazione genomica, possono essere impiegati per veicolare in modo stabile gli RNAi (3).
Vettori Lentivirali
I lentivirus possiedono numerose caratteristiche che li rendono interessanti come sistemi di rilascio in vivo. Rappresentano un tipo di
retrovirus in grado di transfettare sia cellule in divisione che non,
consentendo una stabile espressione di shRNA attraverso l’integrazione del transgene all’interno del genoma della cellula ospite (Figura 7). Questo, grazie alla capacità del proprio involucro di attraversare la membrana nucleare intatta delle cellule verso cui sono diretti.
38
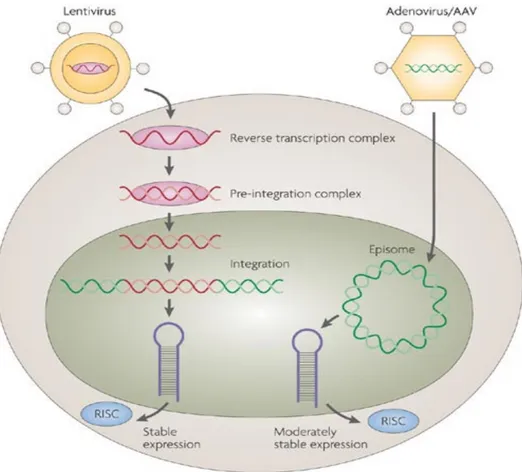
Figura 7. Veicolazione virale degli RNA a forcina corta.
Vettori lentivirali vengono usati per la veicolazione terapeutica di transgeni che permettono l’espressione di shRNA a forcina corta che si integrano nel genoma per una espressione stabile dell’shRNA. I vettori adenovirali e i virus adeno-associati non integrano i loro transgeni nel genoma, ma esprimono gli shRNAs episomamente producendo livelli stabili ma moderati di espressione di shRNA.
(da Daniel H.Kim and John J.Rossi. Nature Reviews. (2007) Vol. 8,173-184).
39
La loro capacità di infettare cellule non in divisione, è fondamentale per un loro possibile impiego contro “targets” come neuroni, macrofagi, cellule del sistema emopoietico, cellule muscolari ed epatiche sulle quali precedenti strategie terapeutiche sono risultate fallimentari.
L’impiego di lentivirus come vettori evita il trattamento ex vivo ed inoltre le cellule vengono riconosciute semplicemente dalle proteine recettoriali di membrana. Inoltre, ad oggi, non sono state riportate come conseguenza, alcun tipo di risposta immunitaria. La specificità cellulare è sicuramente limitata e influenzata dal tipo di lentivirus utilizzato, ma l’aspetto interessante è che si tratta di un fine meccanismo di specificità. Ad esempio, attraverso l’uso di vettori derivanti dal virus dell’HIV è possibile infettare le cellule “T-helper” ed altri macrofagi.
Indubbiamente il maggior vantaggio del sistema di rilascio basato su
lentivirus sta nel fatto che i transgeni espressi dal virus non vengono
silenziati durante lo sviluppo e pertanto possono essere usati per generare animali transgenici attraverso l’infezione di cellule staminali embrionali (ES) o embrioni. Basandosi su questo approccio, alcuni ricercatori hanno usato un sistema lentivirale per il rilascio di shRNA in cellule di mammifero, in cellule staminali e zigoti in modelli murini (3).
È proprio per tutti questi vantaggi che i lentivirus sono stati ampiamente utilizzati per esprimere molti shRNA. Comunemente derivanti dal virus dell’HIV o da quello della immunodeficienza felina, i
40
vettori lentivirali sono spesso assemblati con glicoproteine del virus della stomatite vescicolare (VSV-G) con lo scopo di aumentare il “range” di tipi cellulari che possono essere transfettati. Un’applicazione di RNAi che potrà potenzialmente essere utilizzata in un futuro “trial” clinico riguarda la veicolazione ex vivo mediante vettore lentivirale del sistema di espressione di shRNA in cellule emopoietiche di pazienti affetti da HIV; le cellule transfettate saranno poi reinfuse in questi pazienti per ottenere un beneficio terapeutico in vivo (2).
Vettori Retrovirali
I retrovirus sono una classe di virus con involucro il cui genoma contiene un singolo filamento di RNA di 10 Kb. In seguito all’infezione questo genoma viene trascritto in due filamenti di DNA ed integrato nel genoma dell’ospite. I vettori retrovirali, per il rilascio di shRNA, sono comunemente derivanti dal virus della leucemia murina (Moloney, Mo-MLV) che è in grado di infettare sia i topi che le cellule umane.
La principale limitazione è la richiesta per l’integrazione e l’espressione dei geni virali di cellule “targets” in divisione. Questo restringe il campo di impiego a cellule proliferanti in vivo o ex vivo, affinché la transfezione sia efficace. Se da un lato però questo aspetto
41
rappresenta uno svantaggio, dall’altro ne evidenzia invece la potenziale applicazione in terapie tumorali in vivo (3).
È stato messo in evidenza da diversi studi che i retrovirus vengono facilmente inattivati dagli interferoni, dalla proteina C del complemento e dall’anticorpo anti-α galattosio. Si tratta di considerazioni che hanno senza dubbio rallentato lo sviluppo di tali vettori. Studi futuri dovranno concentrarsi, dunque, sullo sviluppo di vettori retrovirali complemento-resistenti per aprire nuove strade a questa categoria di sistemi di rilascio.
Vettori Adenovirali
I vettori possono infine derivare anche da adenovirus o AAV e sono impiegati per l’espressione transiente di shRNA in strategie terapeutiche contro i tumori ed altre patologie in cui non è desiderata una permanenza a lungo termine del RNAi. Questi vettori non si integrano e rimangono episomiali, integrandosi solo con bassa frequenza, e transfettano sia cellule in divisione che non con i transgenici degli shRNA terapeutici (Figura 6). Un possibile inconveniente dei vettori adenovirali e AAV riguarda il fatto che ripetute somministrazioni possono innescare forti risposte immunitarie, limitandone l’efficacia in certi campi terapeutici.