1
INDICE
1. INTRODUZIONE
2
1.1 Il genere Klebsiella 11
1.2 Eziologia, patogenesi ed epidemiologia
1.2.1 Quadri clinici 15
1.2.2 Differenziazione delle specie genere Klebsiella 16
1.3 Isolamento ed identificazione di Klebsiella spp . 17
1.3.1 Identificazione con metodica MALDI TOF 22
1.3.2 Processo di laboratori 26
1.3.3 Identificazione delle carbapenemasi 29
1.4 Resistenza agli antibiotici 33
1.4.1 Meccanismi di resistenza agli antibiotici 38
1.4.2 Antibiotici, resistenza ai β-lattamici e β lattamasi 44
1.4.2.1 ESBL 46
1.4.2.2 Meccanismo di resistenza 47
1.4.2.3 TEM, SHV, OXA e AmpC 48
1.4.3 Tipizzazione molecolare dei geni β-lattamasi 49
1.4.4 Multi resistenza di Klebsiella pneumoniae 50
1.4.5 KPC e Carbapenemasi 52
1.5 Screening per l’identificazione dei pazienti colonizzati da Enterobatteri
produttori di carbapenemasi 55
1.5.1 Test di screening per individuare i soggetti colonizzati 58 1.6 Prevenzione e controllo della trasmissione di enterobatteri
produttori di carbapenemasi nelle strutture sanitarie e sociosanitari 60
1.6.1 Misure di carattere generale 60
1.6.2 Misure di carattere organizzativo 62
1.6.3 Misure di carattere assistenziale 63
1.7 Segnalazione dei singoli casi e degli eventi epidemici: il sistema
regionale alert di segnalazione rapida 66
2. SCOPO DELLATESI
69
3. MATERIALI E METODI
723.1. Il setting ospedaliero oggetto della ricerca 72
3.2. Procedura di analisi dei campioni clinici per la ricerca
di Klebsiella pneumoniae 72
3.3. Strumenti utilizzati: il sistema automatizzato VITEK 2 75
4. RISULTATI E DISCUSSIONE
82
4.1. Risultati dell’identificazione microbiologica 82
4.2. Risultati antibiogramma 87
5. CONCLUSION
I89
2
1. INTRODUZIONE
La prevenzione delle infezioni correlate all’assistenza pone le sue fondamenta sulla intuizione di Ignaz Semmelweis, che nel 1840 dimostrò l’importanza dell’igiene delle mani nella trasmissione delle infezioni negli ospedali e sul lavoro di Joseph Lister che, introducendo i principi dell’antisepsi intorno al 1860, permise una drastica riduzione delle drammatiche conseguenze delle infezioni post-operatorie.
Nel 1940 l’introduzione sul mercato degli antibiotici contribuì a diffondere l’idea che gli antibiotici potessero eradicare qualsiasi infezione e microrganismo. Negli anni ‘50 questa falsa illusione s’infranse, quando gli ospedali statunitensi e inglesi furono colpiti da una pandemia di infezioni da stafilococchi meticillino resistenti (Robinson DA, 2005); le infezioni ospedaliere hanno continuato a rappresentare la più frequente "complicanza" ospedaliera e il loro trend, in assenza di programmi di controllo, è in continuo aumento.
Le infezioni Ospedaliere (IO) o, con definizione più recente, Correlate alle Pratiche Assistenziali (ICPA) sono infezioni che insorgono durante il ricovero in ospedale e in altre strutture assistenziali o, in altri casi dopo che il paziente è stato dimesso, e che non erano manifeste clinicamente, né in incubazione al momento dell'ammissione. Il concetto di ICPA si estende anche alle infezioni contratte dai lavoratori come risultato delle loro attività professionali all’interno del sistema sanitario. In tale ottica, quindi, le ICPA rientrano in quella categoria di eventi avversi dagestire e controllare nell’ambito del rischio clinico.
Tutte le infezioni già presenti al momento del ricovero (con quadro clinico manifesto o in incubazione) vengono invece considerate acquisite in comunità (infezioni comunitarie), ad eccezione di quelle correlabili ad un precedente ricovero ospedaliero.
A causa dell’evoluzione dei modelli organizzativi dell’assistenza, che sta progressivamente spostandosi verso le prestazioni ambulatoriali e verso il territorio, molte infezioni oggi non si manifestano più durante il ricovero, ma dopo la dimissione, al domicilio del paziente, costituendo un fattore di ulteriore ricorso all’ospedale. È quindi più appropriato parlare di Infezioni correlate alle pratiche assistenziali, piuttosto che di infezioni ospedaliere.
Le ICPA sono un insieme piuttosto eterogeneo di condizioni diverse sotto il profilo microbiologico, fisiologico ed epidemiologico e sono attribuibili alla progressiva introduzione nell'assistenza ospedaliera di nuove tecnologie sanitarie sempre più invasive,
3
che se da una parte consentono la sopravvivenza di pazienti spesso critici (anziani e/o interessati da patologie croniche), dall’altra favoriscono l’acquisizione da parte di questi di infezioni da patogeni opportunisti, attraverso la creazione di condizioni favorenti l’accesso dei microrganismi a siti corporei normalmente sterili, e lo sviluppo di interazioni tra microrganismi e biomateriali utilizzati. Non ultimo, il largo uso di antibiotici a scopo profilattico o terapeutico, ha condizionato l'emergenza di ceppi antibiotico-resistenti, favoriti nella diffusione dall’inefficacia della terapia, e responsabili dell’aumento della morbosità e mortalità intra-ospedaliera.
In Europa si stima che ogni anno si verifichino circa 4 milioni di casi di infezione contratta in ambiente ospedaliero, con un tasso di prevalenza stimato tra il 5 e il 10%, e una letalità dello 0,9% (www.epicentro.iss.it). Nei reparti di chirurgia o in quelli di terapia intensiva la percentuale di infezioni sistemiche di tipo settico può salire fino all’11-12% (www.corist.it). Dati di incidenza sono di più difficile rilevazione. In Italia ogni anno sono coinvolte in infezioni correlate all’assistenza circa 400 mila persone e le sintomatologie più frequenti sono le polmoniti, le setticemie e le infezioni da catetere, che spesso rappresentano anche la causa del decesso: il 2% dei pazienti che contraggono un’infezione in corsia muore, per un ammontare di 8 mila decessi l’anno, oltre 20 al giorno.
La frequenza delle infezioni nei contesti di cura è del 5-10% in pazienti ricoverati in ospedale, 5% in pazienti residenti in strutture per anziani e 1% in pazienti assistiti a domicilio, il 5-10% delle complicanze infettive si manifesta in modo epidemico. Tali eventi si associano spesso a mortalità elevata, con problemi di natura medico-legale e un notevole impatto sull’opinione pubblica.
Localizzazioni più frequenti
32; 39% 22; 26% 15; 18% 14; 17% Tratto urinario Sito chirurgico Polmoniti Inf. Sistemiche
4
Le infezioni della ferita chirurgica si collocano al primo posto in una graduatoria di eventi avversi ordinati per costo medio annuale atteso per il sistema sanitario. Il costo annuale per le infezioni ospedaliere è stato stimato essere pari a 6,7 milioni di dollari negli Stati Uniti e a 1,7 milioni di dollari nel Regno Unito.
È stato dimostrato che il 20-30% delle infezioni nosocomiali sono prevenibili con l’adozione di corretti comportamenti professionali e di specifici programmi di intervento, con conseguente abbassamento dei costi e miglioramento del servizio sanitario. Questa stima cambia però sostanzialmente a seconda del tipo di infezione e del contesto: diversi studi hanno infatti riportato una riduzione della frequenza di complicanze infettive in seguito a programmi di intervento, che andava dal 10% al 70%.
Tra i requisiti più importanti di un programma di controllo delle infezioni acquisite nelle organizzazioni sanitarie vi è la capacità di identificazione tempestiva e corretta gestione degli eventi epidemici. Le epidemie sono infatti eventi rari, ma attesi, la cui frequenza varia, secondo i dati della letteratura, da 1 a 3 eventi ogni 10.000 ricoveri ospedalieri. Se le epidemie vengono identificate tempestivamente, vengono rapidamente adottate appropriate misure di controllo e identificate fonti e meccanismi di trasmissione, è possibile ridurne in modo significativo l’impatto, e soprattutto è possibile modificare eventuali pratiche non corrette, che possono averne condizionato l’insorgenza.
L'insorgenza di una complicanza infettiva in un paziente ricoverato in ospedale comporta costi sia in termini di perdita di salute che economici per il paziente e per l'ospedale: per il paziente, comporta il dover sopportare una patologia infettiva aggiunta alla sua patologia di base, con le eventuali conseguenze di questa in termini di disabilità temporanea o permanente o addirittura il decesso, le spese di una cura domiciliare o la perdita di giornate di lavoro; per l'ospedale, comporta i costi del prolungamento della degenza, degli esami diagnostici e degli interventi terapeutici aggiuntivi. L’impatto economico delle complicanze infettive delle procedure sanitarie è enorme: secondo Vilma Rigobello presidente Simpios (Società Italiana Multidisciplinare per laPrevenzione delle Infezioni nelle Organizzazioni Sanitarie) “L’impatto economico sul sistema sanitario non è inferiore a centomila milioni di euro all’anno e l’onere maggiore è rappresentato dal prolungamento della degenza: il 7,5-10 % delle giornate di ricovero è infatti imputabile all’insorgenza di una complicanza infettiva”.
Le ICPA sono un elemento importante per la definizione della qualità dell'assistenza e pongono problemi di natura medico-legale che hanno un elevato impatto emotivo sui mass-media. Il fatto che in tutti i Paesi occidentali le ICPA continuino a
5
costituire un'importante causa di morbilità, mortalità e costi prevenibili, nonostante la riduzione dei tempi di degenza e le ampie conoscenze disponibili, viene attribuito ad un insufficiente adeguamento di sistema delle organizzazioni sanitarie.
Motivazioni etiche ed economiche dovrebbero indicare le infezioni nosocomiali fra le aree prioritarie di intervento per il miglioramento della qualità dell’assistenza e della sicurezza del paziente. Nonostante l’elevato impatto, sia sociale che economico, dovuto alle ICPA, i sistemi di sorveglianza e di controllo e le azioni per ridurne gli effetti sono invece ancora piuttosto disomogenei da Paese a Paese e a livello nazionale, anche se negli ultimi anni sono stati messi a punto e implementati numerosi programmi.
Il Consiglio d'Europa sin dal 1971 ha emanato numerose raccomandazioni ai singoli Governi in merito al problema delle ICPA, riaffermando nel 1984 con la Raccomandazione n.5 1984/20, che la soluzione, o per lo meno il contenimento di tale complesso fenomeno, dipende dalla messa in opera di una "strategia globale" che interessi tutti i settori dell'ospedale e che necessita, per realizzarsi, della collaborazione di tutti coloro che soggiornano (degenti), frequentano (pazienti ambulatoriali, visitatori) o intervengono nell'ospedale (personale Sanitario e non, altri soggetti facenti parte dell'istituzione).
L'Organizzazione Mondiale della Sanità, sempre nel 1984, indicava a sua volta la lotta alle ICPA come prioritaria nell'ambito del progetto "Salute per tutti nell'anno 2.000" (sottoprogetto: "malattie da infezione").
Nella raccomandazione n.5 del Consiglio d'Europa per attuare la strategia globale predetta, era proposta l'istituzione, all'interno di ogni presidio ospedaliero, di un Comitato per la lotta alle ICPA. che è definito come "..l'organo centrale che sceglie ed elabora la strategia, la impone a tutte le persone in ospedale, ne controlla e ne valuta l'attuazione".
L’indicazione della costituzione del Comitato é annoverata tra le misure “fortemente raccomandate” dai Centers for Disease Control and Prevention (CDC) per la prevenzione delle ICPA.
La comunità scientifica internazionale e le Istituzioni internazionali (WHO e CDC) negli ultimi anni hanno cercato di contrastare il fenomeno infezioni ospedaliere attraverso la promozione di programmi di formazione, di prevenzione, di sorveglianza epidemiologica e di controllo. In occasione della quarta Giornata mondiale dell’igiene delle mani, l’Ecdc ha promosso l’Healthcare-associated Infections Surveillance Network (Hai-Net), la Rete europea di sorveglianza delle infezioni associate all’assistenza, coordinata dallo stesso Ecdc, dedicandogli una sezione tematica sul sito web. La prima
6
giornata mondiale dedicata all'igiene delle mani si è tenuta il 15 ottobre 2008. L'Assemblea generale delle Nazioni Unite ha designato il 2008 “Anno internazionale dell'igiene” (sanitation). Scopo della giornata è stato amplificare e rafforzare l'incitamento a migliorare le pratiche di igiene, già ripetutamente lanciato in tutti i Paesi. Il programma dell'OMS Global Patient Safety Challenge - Clean Care is Safer Care (un'assistenza pulita è un'assistenza più sicura), dedicato al miglioramento dell'igiene delle mani in tutto il mondo e in ogni luogo in cui viene erogata assistenza sanitaria, sostiene attivamente questa iniziativa.
Attualmente l’Ecdc sta sviluppando un database interattivo on line, per la consultazione dei report dell’European Surveillance System (TESSy) sui casi di infezioni correlate all’assistenza in Europa e sulla loro diffusione. Anche il World Health Organization Regional Office for Europe ha allestito un database sui risultati delle indagini di prevalenza.
Uno dei problemi relativi alla sorveglianza delle ICPA è tuttavia la loro identificazione, classificazione e quantificazione. Per cercare di risolvere questo aspetto, sono state messe a punto definizioni di caso (paziente infetto o colonizzato) dai CDC americani, ma anche da programmi europei come Helics e Earss. Negli Stati Uniti e nel nord Europa esiste un sistema di controllo e sorveglianza mentre nel nostro paese questo sistema è ancora in fase sperimentale (MICRONET). Gli studi italiani hanno però rilevato che le caratteristiche epidemiologiche delle ICPA individuate sono simili a quelle descritte dal sistema americano, il National Nosocomial Infections Surveillance System (NNIS), che costituisce quindi un valido punto di riferimento.
Il Ministero della Sanità, al fine di uniformare l'Italia alla maggior parte dei Paesi Europei ed agli Stati Uniti, con circolare n. 52 del 20/12/85 (“Lotta contro le infezioni ospedaliere”) e successivamente con circolare n.8 del 30/1/88 (“Lotta contro le infezioni ospedaliere: sorveglianza”), ed infine con la circolare n.1034 del 9/9/00, recepisce in pieno le raccomandazioni europee, ufficializzando il problema e indicando la composizione di massima del Comitato per le ICPA, nonché alcuni provvedimenti organizzativi da attuare in ciascun presidio ospedaliero.
Il Comitato predetto, è stato anche previsto dal Decreto del Ministero della Sanità 13 settembre 1988 sulla determinazione degli standards del personale ospedaliero e dall’art. 135 del DPR 28.11.90, n.384 che disciplina le "commissioni per la verifica e la revisione della qualità dei servizi e delle prestazioni sanitarie, al comma 6 lettera m indica, in particolare, gli obiettivi relativi alla valutazione di progetti di metodologie per la
7
prevenzione delle ICPA, confermato dal D.L. 30.12.92, n° 502, art. 10, riguardante il controllo di qualità dell’assistenza nei confronti della generalità dei cittadini.
L’attività di prevenzione e controllo delle ICPA deve essere intesa come un intervento multidisciplinare in cui più professionisti (direttore medico, medici, infermiere, microbiologo, infettivologo, farmacista ecc.), con le proprie specifiche competenze e responsabilità, concorrono per un obiettivo comune: "promuovere la qualità dell’assistenza prevenendo le ICPA in un processo di Continuous Quality Improvement (CQI).”.
Anche il Piano sanitario nazionale (PSN) 2006-2008 in vigore ed il prossimo 2008-2011, oltre ai Piani sanitari regionali (PSR), prendono in considerazione il problema. In particolare, tra gli obiettivi prioritari prevedono una riduzione dell'incidenza delle ICPA. con particolare riguardo alle infezioni delle vie urinarie, infezioni della ferita chirurgica, polmonite post operatoria o associata a ventilazione assistita ed infezioni associate a cateteri intravascolari. Gli stessi PSR sanciscono la piena attivazione dei comitati di controllo delle infezioni ospedaliere, all'interno dei presidi ospedalieri, mediante l'assegnazione di specifiche responsabilità gestionali a personale qualificato e la definizione di politiche di intervento e di procedure convalidate attraverso sistemi di verifica dell’applicazione di linee guida basate sulle migliori evidenze scientifiche esistenti.
In entrambi i documenti citati, tra le azioni previste per il raggiungimento dell'obiettivo, è indicata l'attivazione di un programma per la sorveglianza, la prevenzione ed il controllo delle ICPA, orientato sia ai pazienti sia agli operatori sanitari. In particolare, è posto l'accento sulla sorveglianza come strumento utile non solo per descrivere e misurare il fenomeno, ma anche, e soprattutto, per proporre interventi di gestione e di prevenzione efficaci.
Le Circolari ministeriali non indicano modalità raccomandate di funzionamento; riconoscono ampia discrezionalità al fine di adeguare gli interventi alle necessità locali.
Il CIO, va precisato, assume nelle istituzioni in cui opera un ruolo di riferimento e guida per tutte le attività di sorveglianza, controllo e prevenzione delle infezioni. In Italia è stato istituito dalla legge del 26 maggio 2004, n.138, il Centro nazionale per la prevenzione e il controllo delle malattie (Ccm), un organismo di coordinamento tra il Ministero della Salute e le Regioni per le attività di sorveglianza, prevenzione e risposta tempestiva alle emergenze. Il Ccm ha lo scopo di contrastare le emergenze di salute pubblica legate prevalentemente alle malattie infettive e diffusive e al bioterrorismo. Nel 2004, è stato creato il Sistema di allerta su isolamenti microbici –Micronet. Micronet è un progetto
8
portato avanti dal Ccm dal Cnesps, con l'obiettivo di creare una rete di laboratori sentinella per lo scambio tempestivo di informazioni sui solamenti microbici.
Sempre in Italia, a livello di associazioni private, è stata fondata la Società Italiana Multidisciplinare per la Prevenzione delle Infezioni nelle Organizzazioni Sanitarie (SIMPIOS), un’associazione che, in collaborazione con l’Istituto Superiore della Sanità, si propone di integrare i contributi delle diverse competenze coinvolte nello studio e nella prevenzione delle infezioni ospedaliere e di valorizzare la professionalità del personale dedicato, nonché di promuovere, anche di concerto con Istituzioni e Enti Pubblici, Associazioni Professionali e altre Società Scientifiche, la realizzazione di strumenti di lavoro (linee guida, procedure validate, documenti di consenso), portali di epidemiologia per facilitare l’accesso all’informazione tramite l’uso della rete internet (Epi Centro), l’organizzazione di iniziative di formazione ea ggiornamento e l’attivazione di iniziative di ricerca. Inoltre, tra gli altri organi coinvolti in questa problematica, troviamo l’ Associazione Nazionale Infermieri addetti alla Prevenzione e Controllo delle Infezioni Ospedaliere (ANIPIO), società nata agli inizi degli anni 90.Per controllare e ridurre le infezioni ospedaliere è necessario che, a livello attuativo, le strutture agiscano su più fronti, attraverso l’adozione di misure di prevenzione e di controllo delle ICPA che prevedano azioni rivolte alle strutture ospedaliere, ai sistemi di ventilazione e ai flussi d’acqua, all’igiene del personale e dell’ambiente; che individuino personale e protocolli dedicati alla sorveglianza attiva delle infezioni e un appropriato flusso informativo, che permetta l’identificazione e la quantificazione delle infezioni stesse nei diversi presidi; che provvedano alla formazione del personale dedicato al trattamento dei pazienti, soprattutto nelle aree critiche di terapia intensiva e chirurgica, e di quello dedicato alla raccolta e analisi dei dati.
Da qui l’importanza di un sistema di sorveglianza attiva tesa ad aumentare la percezione del problema ed identificare possibili aree problematiche, confermare l’esistenza di criticità, analizzare i motivi alla base dei problemi riscontrati (GastmeierP, ICHE 2000). A tal fine, è essenziale identificare i patogeni di particolare interesse in quanto frequentemente associati a epidemie o perché multiresistenti, “gli alert organisms”. Gli alert organism sono particolari ceppi responsabili di epidemie, in grado di diffondersi rapidamente in ospedale se non controllati adeguatamente, e in grado di acquisire e trasferire rapidamente resistenze agli antimicrobici. La lista deve essere concordata tra i vari reparti ed il laboratorio tenendo conto dei seguenti elementi:
9
a) i patogeni da includere devono essere caratterizzati da una elevata diffusibilità oppure patogenicità;
b) devono essere disponibili interventi per prevenirne la diffusione
Tra gli alert organism sono frequentemente inclusi, oltre a microrganismi dotati di elevata patogenicità, ceppi MDR (multi drug resistent) come:
- VRE Vancomycin-resistant enterococcus
- MRSA Methicillin-resistant Staphylococcus aureus
- ESBL+ Extended-spectrum β-lactamase-producing Escherichia coli - Bacilli Gram – non fermentanti come P.aeruginosa e Acinetobacter spp
Nell’ultimo ventennio è stato osservato, ovunque nel mondo, una sempre maggiore diffusione di microrganismi Gram negativi resistenti a diverse classi di antibiotici. In precedenza, nella pratica clinica, il fenomeno della multi resistenza agli antibiotici era stato riscontrato –tra i Gram negativi- in modo pressoché esclusivo a carico di Pseudomonas aeruginosa, per il quale l’epidemiologia delle resistenze era tuttavia assai variabile tra ospedale e ospedale, e anche, nell’ambito della singola struttura, tra reparto e reparto (Istituto Direttivo Simpios 2010).
Il fenomeno della multi resistenza ha poi interessato altri comuni batteri Gram negativi quali le Enterobacteriaceae produttrici di β-lattamasi a spettro esteso(ESBL), diffuse capillarmente nel nostro Paese già a far corso dalla metà degli anni ’90 e, più o meno nello stesso periodo, anche Acinetobacter baumannii resistente a diverse classi di antibiotici.
In tutti i casi, però, i batteri Gram-negativi risultavano ancora sensibili ai carbapenemici.
Nel 2001 è stato descritto per la prima volta in Klebsiella pneumoniae un nuovo fenotipo di resistenza dovuto alla presenza di una nuova carbapenemasi, cosiddette carbapenemasi tipo KPC (Klebsiella pneumoniae carbapenemase).
Da allora sono state descritte diverse varianti di “Klebsiella pneumoniae carbapenemases”. Esse possono causare livelli di resistenza bassi, non sempre e non facilmente riconosciuti dagli strumenti automatici di laboratorio: il microbiologo ne deve sospettare la presenza quando - utilizzando metodi “manuali” di analisi – viene in evidenziata la presenza di unEnterobacteriacea (quasi sempre K. pneumoniae, più raramente K. oxytoca, Escherichiacoli o altri Eenterobatteri) per la quale il diametro dell’alone di inibizione di ertapenem o di meropenem (ma non di imipenem, che non è un marcatore efficiente) è < 22 mm oppure quando la CMI è > 1 mg/mL.
10
In particolare, le KPC di classe A sono enzimi in grado di idrolizzare non solo i carbapenemi ma anche le penicilline, le cefalosporine e l’aztreonam.
In questo caso è necessario che il Laboratorio di Microbiologia predisponga test supplementari in grado di determinare se il ceppo microbico isolato in coltura sia effettivamente portatore del plasmide KPC. In presenza di risposta positiva, il Laboratorio di Microbiologia deve “interpretare” i risultati e provvedere alla loro refertazione sulla base dei valori di CMI ottenuti; imipenem e meropenem dovrebbero essere refertati come resistenti se la loro CMI è superiore a 8 mg/mL e non essere refertati se, al contrario, la CMI è inferiore a 8 mg/mL. Il referto dovrebbe essere corredato da una nota di commento che recita: “il microrganismo isolato in coltura è produttore di carbapenemasi: l’efficacia di antibiotici appartenenti alla classe dei carbapenemici non è provata, in tale situazione, nella pratica clinica” .
Infatti - sebbene i valori di CMI siano compatibili con una possibile efficacia terapeutica - variabili in vivo, quali la carica batterica o il volume di distribuzione del farmaco, potrebbero correlarsi con una inefficace azione dell’antibiotico; è tuttavia necessario ricordare che sono descritti casi in cui la terapia con carbapenemici a dosaggi elevati (sino a 4 g/die di imipenem/cilastatina, sino a 6 g/die di
meropenem) si è rivelata efficace. Per assumere decisioni razionali è fondamentale l’attenta valutazione della significatività clinica, potendosi spesso trattare di colonizzazioni e non di infezioni. Per poter definire il significato clinico di questi isolamenti è quindi necessaria una stretta collaborazione fra microbiologo, infettivologo e clinico.
Recentemente (2009) è stata descritta una nuova carbapenemasi in un ceppo di Klebsiella pneumoniae isolata da un paziente svedese di ritorno dal Pakistan . Si tratta di una metallo-β-lattamasi (MBL) - βlattamasi di classe B, idrolizzanti i carbapenemici - denominata NDM-1 (New Delhi Metallo-β-lactamase type 1) che presenta caratteristiche di assoluta novità.
Le MBL possono essere occasionalmente riscontrate negli enterobatteri, che possono acquisirle da Gram negativi non fermentanti il glucosio come Pseudomonas aeruginosa. Ciò è accaduto, ad esempio, in occasione di recenti epidemie in Grecia e in Turchia.
NDM-1 è invece sostanzialmente diversa dalle altre MBL riscontrate negli enterobatteri e questo fa supporre che essa potrebbe provenire da microrganismi diversi da Pseudomonas aeruginosa. Inoltre, questo enzima pare dotato della capacità di trasferirsi con estrema
11
facilità fra diversi generi di microrganismi: lo stesso plasmide NDM-1 è stato infatti riscontrato anche in un ceppo di E. coli isolato dalle feci del paziente svedese.
Recentemente è stata valutata l’epidemiologia di questo plasmide che, a partire dalla prima segnalazione in Svezia, pare ora diffondersi anche nel Regno Unit. Sinora in Inghilterra sono stati segnalati 37 pazienti che albergano Enterobacteriaceae portatrici del plasmide NDM-1: sono tutti pazienti che riferiscono viaggi in India e Pakistan.
In questi paesi la diffusione di questo tratto di resistenza parrebbe essere ancora più estesa e riguarderebbe non solo K. pneumoniae ma anche ceppi di E. coli.
Tutti questi microrganismi sono, quindi, multiresistenti: le uniche molecole efficaci in vitro sembrano essere colistina e tigeciclina, molecole che dal punto di vista terapeutico presentano limiti intrinseci per la scarsa o nulla diffusibilità tissutale (colistimetato sodico, pro-farmaco della colistina) o per l’attività esclusivamente batteriostatica (tigeciclina). L’emergenza e il diffondersi di questo fenotipo di resistenza potrebbe quindi essere veramente drammatico. A fronte di questo panorama assai preoccupante non è possibile rivolgerci con fiducia alle risorse terapeutiche in nostro possesso. Il passato può riservarci qualche gradita sorpresa (il cloramfenicolo – pur con tutti i limiti del caso - può essere considerato una possibile opzione terapeutica), ma il futuro sicuramente si prospetta povero di novità: anche la molecola sperimentale NXL104, inibitore delle carbapenemasi, testata in combinazione con cefepime e ceftazidime non sembra fornire risultati confortanti.
1.1 Il Genere Klebsiella
Il genere Klebsiella, famiglia Enterobacteriaceae, ha preso il nome da V. Trevisan nel 1885 in onore del batteriologo tedesco Theodor Albrecht Edwin Klebs (1834-1913). Il Dr Klebs è conosciuto per il suo lavoro pionierististico nel dimostrare come i microrganismi sono responsabili delle malattie infettive.
Comprende batteri Gram negativi, di aspetto bastoncellare, capsulati aerobi-anaerobi facoltativi, glucosio fermentanti, ossidasi negativi. Inoltre, producono un materiale mucoide che dà alle colonie un aspetto gelatinoso. Il genere Klebsiella fa parte del gruppo dei coliformi e si distingue da Enterobacter per essere sempre immobile (carattere unico nella famiglia delle Enterobacteriacee, condiviso col genere Shigella).
12
Sono normalmente presenti nella mucosa respiratoria e nell'intestino dell'uomo e a livello faringeo. In questa sede la loro frequenza aumenta nei soggetti alcoolisti, fino al 40%. Sono anche normali colonizzatori dei suoli e delle acque.
Il genere Klebsiella è ben noto alla maggior parte dei medici come causa di polmonite acquisita in comunità batterica, che si verificano soprattutto negli alcolisti cronici e mostrando caratteristiche anomalie radiografiche a causa di una grave infezione da piogeni, che ha un alto tasso di mortalità se non trattata.
La maggior parte delle infezioni da Klebsiella, tuttavia, sono associati ad ospedalizzazione. Come patogeni opportunisti, Klebsiella spp. principalmente infettano individui immunocompromessi che sono ricoverati in ospedale e soffrono di gravi patologie concomitanti come il diabete mellito o ostruzione cronica polmonare.
Si stima che Klebsiella spp. causano l'8% di tutte le infezioni batteriche nosocomiali negli Stati Uniti e in Europa. Non sono state notate grandi variazioni geografiche nella frequenza. Negli Stati Uniti, Klebsiella rappresenta 3 al 7% di tutte le infezioni batteriche nosocomiali, inserendoli tra gli otto più importanti patogeni infettivi negli ospedali e i dati raccolti nel Regno Unito e in Germania sono molto simili a quelli riportati dai Centers for Disease Control and Prevention.
Il tratto urinario è la sede più comune di infezione. Il genere Klebsiella rappresenta tra il 6 e il 17% di tutte le infezioni nosocomiali del tratto urinario (UTI) e mostra un'incidenza ancora maggiore in gruppi specifici di pazienti a rischio, ad esempio, i pazienti con vesciche neuropatiche o con diabete mellito. Delle batteriemie nosocomiali da gram-negativi, Klebsiella è secondo solo a Escherichia coli.
Nei reparti pediatrici, le infezioni nosocomiali da Klebsiella sono particolarmente problematiche, soprattutto nei neonati prematuri e nelle unità di terapia intensiva. Il genere Klebsiella è spesso il patogeno coinvolto nella sepsi neonatale, sia precoce manifestazione e tardo-manifestazione infezioni
Le infezioni nosocomiali da Klebsiella sono causate principalmente da Klebsiella pneumoniae, la specie più importanti del genere. In misura molto minore, K. oxytoca è stata isolata da campioni clinici umani.
Klebsiella pnumoniae, anche chiamata ‘’bacillo di Friedländer’’ fu per la prima volta descritta nel 1882 dal patologo e microbiologo Carl Friedländer. E’ meglio conosciuta come il patogeno del sistema respiratorio umano che causa la polmonite. E’ la specie più comune di tutto il genere con il 95% degli isolamenti. K. pneumoniae si comporta da patogeno opportunista e puo’ causare un gran numero di infezioni diverse.
13 1.2 Eziologia, Patogenesi ed epidemiologia
Le Klebsiellae sono dei grossi batteri capsulati, immobili, che si coltivano facilmente in laboratorio, fermentano il lattosio e danno grosse colonie mucoidi. K. pneumoniae è la specie più importante in patologia umana, seguita da K. oxytoca, che si propaga con maggiore frequenza nei ricoveri per anziani e nei reparti per lungodegenti, oltre che negli ospedali, ed è stata identificata come specie distinta dalla prima, in base all’esame del DNA, oltre ad essere indolo positiva. K. rhinocleromatis e K. Ozenae, che interessano quasi esclusivamente le prime vie aeree, sono oggi ritenute due sottospecie di K. pneumoniae e sono presenti di preferenza nei climi tropicali (Africa centrale, Asia meridionale, Sud – America), ma anche nell’Europa dell’est.
Sulla base dell’antigene capsulare k si distinguono oltre 80 sierotipi di K. pneuomoniae, tra i quali i più frequenti in patologia umana sono i tipi 1, 3, 4 e 5.
Klebsiella spp. sono onnipresenti in natura, probabilmente possiedono due habitat, uno dei quali è l'ambiente, in cui si trovano in acque superficiali, acque reflue, e del suolo e sulle piante come saprofiti ambientali; l'altro sono le superfici mucose dei mammiferi come esseri umani, cavalli, o suini, che colonizzano. A questo, il genere Klebsiella è come Enterobacter e Citrobacter, ma differisce da Shigella spp. o E. coli, che sono comuni negli esseri umani, ma non nell'ambiente.
Colonizzano spesso l’apparato digerente dei mammiferi e dell’uomo, di meno l’orofaringe, in via solo transitoria la cute, sono state ritrovate nel rino-faringe del 6% di soggetti sani e del 40% degli alcoliti sottoposti a controllo. La trasmissione più frequente è da uomo a uomo.
Fin dalla sua scoperta Klebsiella pneuomoniae è stata collegata ad infezioni respiratorie umane, incide per il 5% circa di tutte le polmoniti.
E’ significativo che questa specie è stata identificata come causa di polmonite franca da Friedlander nel 1882, quattro anni prima dello stesso pneumococco. L’alcolismo, ma anche la pneumopatia cronica ostruttiva e il diabete sono fattori predisponenti significativi delle infezioni polmonari della comunità da parte di questo batterio. Oggi tuttavia le infezioni più frequenti sono quelle ospedaliere, specie nelle unità di cura intensive, dove ha dato vere e proprie epidemie, spesso da parte di ceppi antibiotico-resistenti. La ventilazione meccanica è un fattore di rischio importante. Si sono verificate epidemie correlate all’inoculo di soluzioni saline contaminate.
14
Quando si verifica un ‘’cluster’’ di infezione da parte di questo o altre specie batteriche, è rilevante effettuare un controllo dei ceppi isolati con metodiche di analisi genetica, quali l’elettroforesi pulsata, per dimostrarne la monoclonalità e identificare l’origine e la sequenza del contagio.
I quadri clinici extraintestinali ricalcano quelli descritti per E. coli, anche se, per frequenza, sono in testa le infezioni polmonari, mentre meno frequenti sono le infezioni a carico degli altri organi. Negli esseri umani, K. pneumoniae è presente come saprofita nel naso faringe e nel tratto intestinale. I valori di colonizzazione differiscono notevolmente da studio a studio.
Nei campioni di feci varia da 5 al 38%, mentre i valori nel naso faringe il range varia tra l’1 e il 6%.
Poiché i batteri gram-negativi non trovano buone condizioni di crescita sulla cute umana, Klebsiella spp. sono raramente riscontrati e considerati semplicemente come membri transitori della flora.
Questi valori cambiano drasticamente in ambiente ospedaliero, dove possono aumentare in misura direttamente proporzionale alla durata del soggiorno. Anche il personale ospedaliero presenta valori elevati di presenza di Klebsiella.
Nei pazienti ospedalizzati si sono registrati valori del 77% nelle feci, il 19% nella faringe, e il 42% sulle mani di pazienti. L'alto tasso di colonizzazione Klebsiella nosocomiale sembra essere associato all'uso di antibiotici piuttosto che a fattori connessi alle cure in ospedale, tuttavia sia la colonizzazione che l’infezione sono correlate alla durata della degenza, all’uso di dispositivi medici e alle pratiche assistenziali invasive o immunomodulanti. Una precedente terapia antibiotica è comunque il fattore di rischio più significativamente associato con l'acquisizione di Klebsiella da parte del paziente.
In uno studio è stato osservato che 2 settimane dopo l'ammissione in ospedale, si ha un aumento da due a quattro volte dei valori di colonizzazione da Klebsiella, questo aumento si è verificato principalmente in pazienti trattati con antibiotici, soprattutto in persone che ricevono più antibiotici ad ampio spettro.
L'uso diffuso della terapia antimicrobica è stato spesso ritenuto responsabile dell'insorgenza di ceppi di Klebsiella resistenti negli ospedali. Poiché questi effetti indesiderati possono essere invertiti da un rigoroso controllo dell'uso di antibiotici, sono necessarie strategie per evitare l'uso eccessivo di antibiotici nella profilassi e nella terapia. Oltre alle apparecchiature mediche (contaminate a causa di procedure igieniche difettose e all’uso promiscuo degli apparecchi), il serbatoio principale per la trasmissione di
15
Klebsiella in ambito ospedaliero è il tratto gastrointestinale dei pazienti, mentre le mani del personale ospedaliero sono spesso il tramite per la diffusione dell’infezione. La capacità di questo organismo a diffondersi rapidamente porta spesso a focolai nosocomiali, specialmente nelle unità neonatali e in generale di terapia intensiva. Delle 145 infezioni nosocomiali epidemiche riportate in letteratura pubblicata in lingua inglese tra il 1983 e il 1991, 13 sono stati causati da Klebsiella. Secondo le statistiche dei Centers for Disease Control and Prevention, la Klebsiella spp. rappresentano l'8% delle infezioni ospedaliere endemiche e il 3% dei focolai epidemici.
1.2.1 Tassonomia
La tassonomia di Klebsiella è caratterizzata da una nomenclatura che riflette la sua storia tassonomica. Originariamente, l'importanza medica del genere Klebsiella (famiglia Enterobacteriaceae) l’ha portato ad essere suddiviso in tre specie corrispondenti alle malattie di cui sono causati: K. pneumoniae, K. ozaenae, e K. rhinoscleromatis.
Mentre la tassonomia diventava sempre più raffinata, grazie allo sviluppo di nuovi metodi, come la tassonomia numerica, la classificazione delle specie di questo genere fu continuamente rivisto. Con il tempo emersero tre principali classificazioni, quelle di Cowan, Bascomb e Orskov (Tabella 1).
Tabella 1. Classificazione delle specie del genere da Klebsiella con diversi sistemi tassonomici
Classificazione:
Cowan Bascomb Orskov
K. aerogenes K. edwardsii K. aerogenes / oxytoca / edwardsii K. pneumoniae subsp. pneumoniae
subsp. edwardsii K. pneumoniae subsp. ozaenae
subsp. atlantae sensu stricto subsp. rhinoscleromatis
K. pneumoniae sensu lato K. oxytoca
K. ozaenae K. ozaenae K. terrigena
K. rhinoscleromatis K. rhinoscleromatis K. planticola (sin. K. trevisanii) K. "gruppo senza nome"
16
Nei primi anni ‘80, Klebsiella isolate dall'ambiente, che erano state precedentemente classificate come "Klebsiella organismi simili" (gruppi J, K, L e M), sono state sempre più spesso classificate con unità tassonomiche (taxa) provvisorie.
Questi gruppi hanno dato luogo a quattro nuove specie: K. terrigena, K. ornithinolytica, K. planticola, e K. trevisanii. Nel 1986, le ultime due specie sono state combinate in una sola specie, K. planticola, a causa della loro ampia omologia di sequenza DNA. Mentre, inizialmente considerati prive di significato clinico limitato e per gli organismi acquatici, botanici, e gli ambienti del suolo, K. terrigena e K. planticola sono state recentemente riscontrate in campioni clinici umani. Secondo questi risultati, in particolare K. planticola è stata isolata da infezioni umane con una frequenza sorprendentemente alta del 3,5 al 18,5% tra gli isolati clinici di Klebsiella. Più della metà di questi isolati sono stati recuperati dalle secrezioni delle vie respiratorie; isolati da ferita e urine sono stati i più comuni. Tuttavia, poiché la maggior parte degli isolati sono stati ottenuti da campioni polimicrobiche, è difficile stimare il significato di questi ceppi come agenti causativi di malattie. Tuttavia, sei dei 94 isolati sono stati recuperati da campioni monomicrobici e potrebbe essere assegnato alle infezioni corrispondenti. Pertanto, attualmente sembra possibile che oltre a K. pneumoniae e K. oxytoca, una terza specie di Klebsiella esista e sia in grado di causare infezioni umane.
L'adozione di una nomenclatura coerente è stata ulteriormente complicata dal fatto che la Gran Bretagna e i paesi dell’ex Commonwealth aderirono alla classificazione di Cowan mentre gli Stati Uniti preferirono la classificazione di Orskov. Di conseguenza, lo stesso batterio può essere chiamato K. pneumoniae in un paese e K. aerogenes in un altro. Maggior parte dei paesi europei seguano l'esempio americano e riconoscono la classificazione a livello mondiale predominante quella di Orskov.
1.2.2 Differenziazione delle specie genere Klebsiella
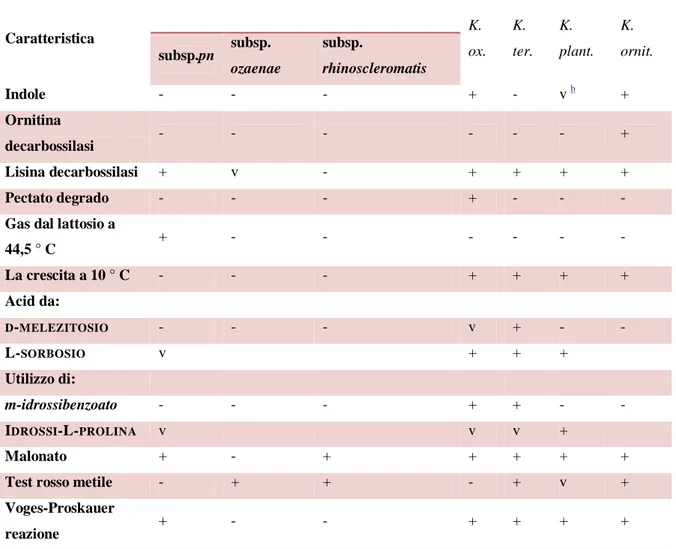
Klebsiella sono di solito identificate e differenziate in base alle loro reazioni biochimiche. Il genere è definito come contenente gram-negativi, non mobili, solitamente a forma di barra incapsulati batteri della famiglia delle Enterobacteriaceae, che producono la lisina decarbossilasi ma non ornitina decarbossilasi e sono generalmente positivo nel Voges-Proskauer. All'interno del genere Klebsiella, le singole specie possono essere differenziate sulla base delle caratteristiche elencate nella tabella Tabella 2 . Considerando che la maggior parte delle specie Klebsiella possono essere identificati tramite test di laboratorio
17
standard microbiologici, la specie K. terrigena e K. planticola richiedono speciali, reazioni non convenzionali (come l'utilizzo di m-idrossibenzoato o idrossi-L-prolina, degradazione pectato, acido da melezitosio, o crescita a 10 ° C).
Tabella 2. Reazioni biochimiche di Klebsiella spp.
Caratteristica Klebsiella pneumoniae K. ox. K. ter. K. plant. K. ornit. subsp.pn subsp. ozaenae subsp. rhinoscleromatis Indole - - - + - v b + Ornitina decarbossilasi - - - + Lisina decarbossilasi + v - + + + + Pectato degrado - - - + - - -
Gas dal lattosio a
44,5 ° C + - - - - La crescita a 10 ° C - - - + + + + Acid da: D-MELEZITOSIO - - - v + - - L-SORBOSIO v + + + Utilizzo di: m-idrossibenzoato - - - + + - - IDROSSI-L-PROLINA v v v + Malonato + - + + + + +
Test rosso metile - + + - + v +
Voges-Proskauer
reazione + - - + + + +
1.3.Isolamento ed identificazione di Klebsiella spp.
L'identificazione dei microrganismi è importante perché consente di accoppiare un nome ad un insieme di proprietà possedute da un'entità microbica rinvenibile in un determinato habitat.
Da un punto di vista scientifico, l'identificazione è importante per lo scambio, il confronto, la catalogazione, l'archiviazione e l'unificazione delle conoscenze.
18
I microrganismi possono essere identificati, come specie già note o come nuove entità tassonomiche, sulla base di caratteristiche fenotipiche e/o genotipiche.
Da un punto di vista epidemiologico, è spesso necessario determinare la clonalità dei ceppi. Ciò è particolarmente importante in caso di focolai nosocomiali endemici ed epidemici di infezioni da Klebsiella per migliorare la gestione di tali epidemie. Una varietà di metodi sono stati usati con vari gradi di successo per l’identificazione di Klebsiella e sono discusse di seguito.
Caratteristiche fenotipiche
Già l'aspetto della colonia che si sviluppa sulla piastra di isolamento può essere di rilevanza diagnostica. Questo carattere può infatti essere definito per ogni specie microbica in determinate condizioni colturali. Utili risultano i terreni selettivi-differenziali quali l’agar Mac Conkey contenente il lattosio, sul quale le colonie di Klebsiella si presentano di colore rosa chiaro o biancastro alla periferia con il centro colorato in rosa di aspetto mucoso (Fig. 1).
Figura 1 Colonie di Klebsiella Biotipizzazione
La Biotipizzazione basata su un gruppo allargato di test biochimici e colturali è sicuramente il metodo più pratico per i laboratori più piccoli che non sono attrezzati in modo ottimale. La Biotipizzazione può essere effettuata utilizzando solo test macrotube (enterotube) oppure combinando un sistema commercialmente disponibile miniaturizzato
19
come il sistema API 20E con prove macrotube supplementari, strumenti attualmente sostituiti dal sistema VITEK2 più moderno e veloce.
Tuttavia, a causa del gran numero di reazioni da testare e spesso i lunghi tempi di coltivazione, fino a 90 giorni per la dimostrazione della gelatinasi ( biotipizzazione di Klebsiella spp.) non è molto adatto come strumento epidemiologico.
Sierotipizzazione
La Sierotipizzazione è attualmente la tecnica più diffusa per la tipizzazione Klebsiella spp. Essa si basa principalmente su una ripartizione secondo gli antigeni capsulari.
Klebsiellae di solito hanno capsule polisaccaridiche ben sviluppate, che danno alle colonie il loro caratteristico aspetto mucoide. Degli 82 antigeni capsulari descritti, 77 tipi costituiscono la base per essere riconosciuti a livello internazionale. Sebbene siano stati descritti 12 diversi tipi di antigene O di Klebsiella, essi sono difficili da classificare, perché la loro determinazione è ostacolato dalle capsule termostabili. La Tipizzazione capsulare, invece, mostra buona riproducibilità ed è in grado di differenziare la maggior parte degli isolati clinici. Lo svantaggio di questo metodo è il numero elevato di reazioni sierologiche incrociate che si verificano tra i 77 tipi di capsula. In questo modo, i singoli sieri saranno impegnati con le cross reazioni degli antigeni K. Inoltre, la procedura di digitazione è ingombrante a causa del tempo necessario per eseguire il test ed è suscettibile di interpretazioni soggettive a causa di reazioni deboli che non sono sempre facili da interpretare. Poiché antisieri anti-capsula non sono commercialmente disponibili, questa tecnica è praticata soprattutto in laboratori specializzati.
L'uso combinato di biotipizzazione e tipizzazione capsulare consente la differenziazione di un gran numero di sierotipi.
Phage Typing
La Tipizzazione dei fagi di Klebsiella è stata sviluppata nel 1960. Sebbene la reazione fago è facilmente interpretabile e la riproducibilità del metodo è accettabile, questa tecnica presenta un tasso relativamente scarso di tipizzazione del 19 al 67%. Poiché non è un'alternativa alla tipizzazione capsulare, questa procedura non è diffusa ed è utile soprattutto come metodo secondario in combinazione con test sierologici.
20 Batteriocina Typing
La tipizzazione capsulare è il metodo preferito per l’identificazione di Klebsiella, tuttavia è stato consigliato di includere una funzionalità aggiuntiva indipendente dal tipo di capsula per consentire una più precisa analisi epidemiologica. Molti autori consigliano di tipizzare Klebsiella per mezzo delle batteriocine. Le Batteriocine sono sostanze battericide, solitamente le proteine prodotte da batteri per inibire la crescita di altri batteri, di solito altri membri della stessa specie.
Un isolato può essere caratterizzato sia dalla sua capacità di inibire ceppi indicatori specifici o per la sua sensibilità di batteriocine sintetizzati da un insieme di ceppi produttori. Poiché la sintesi di batteriocine non è abbastanza frequente in Klebsiella, quest'ultima tecnica è diventato il metodo di scelta per la tipizzazione batteriocina di organismi appartenenti a questo genere. Tuttavia, i due principali primi metodi, il metodo della crescita in brodo e il metodo di cross-streak, mostrano entrambi notevoli svantaggi. A causa della instabilità dei preparati di batteriocina, la riproducibilità del metodo della crescita in brodo è bassa. Inoltre, il convenzionale metodo di cross-streak provoca typability bassa di ceppi. I limiti di entrambi questi metodi sono stati sormontati da una modifica della procedura, che evita l'uso di batteriocine pre prodotte e memorizzati potenzialmente instabili. Invece, le batteriocine sono sintetizzati in agar immediatamente prima dei ceppi da tipizzare e vengono inoculati da un inoculator multipoint. Questo metodo si è dimostrato superiore per la tipizzazione batteriocina di Klebsiella in ceppi clinici e ambientali nonché di epidemie nosocomiali di Klebsiella.
I Metodi di tipizzazione molecolare maggiormente utilizzati, applicati al genere Klebsiella, sono:
Multi Locus Sequence Typing (MLST);
Amplified fragment length polymorphism (AFLP); Pulsed Field Gel Electrophoresis (PFGE).
21 MLST
Analisi di sequenze di sette geni housekeeping atta a differenziare ceppi e linee clonali. I geni housekeeping sono i componenti del cosiddetto core genome, essi sono necessari allo svolgimento delle tappe fondamentali del metabolismo cellulare, codificando proteine essenziali per la sopravvivenza del batterio; sono dunque geni conservati in tutte le specie batteriche e non sottoposti a trasferimenti orizzontali. I geni housekeeping vengono utilizzati per tracciare l'evoluzione nel tempo delle specie batteriche e per differenziare ceppi e linee clonali.
Oltre al core genome, i batteri possiedono un flexible genome, composto da geni che codificano proteine per funzioni secondarie della cellula, ad esempio l’adattamento a particolari condizioni ambientali o la scelta di un determinato habitat. Il flexible genome è rappresentato da geni associati alla virulenza, alla resistenza e da elementi genetici mobili come plasmidi e trasposoni. Per definizione questa parte del genoma batterico è in continua evoluzione mediante perdita o acquisizione di geni per trasferimento orizzontale, e per questo, non in grado di dare informazioni veritiere sull’evoluzione di una specie batterica. Un aspetto importante del MLST è rappresentato dalla possibilità di misurare la ricombinazione genetica tra ceppi batterici; questa è alla base della dinamicità, in termini evolutivi, di una popolazione batterica, infatti, quando vi è passaggio di materiale genetico tra linee clonali diverse, si creano nuovi genotipi che meglio si adattano a particolari condizioni ambientali o a specifici ospiti. Possiede un potere discriminante moderato.
AFLP
La tecnica AFLP (Amplified Fragment Lenght Polymorphism) è stata messa a punto da Vos nel 1995 ed è basata sulla combinazioni di due diverse fasi: la digestione del DNA, con una coppia di enzimi di restrizione, e l’amplificazione selettiva dei frammenti ristretti mediante l’ausilio di primers specifici complementari alle sequenze di taglio. L’AFLP presenta numerosi vantaggi, come quello di essere universalmente applicabile a tutti gli organismi viventi e quello di permettere l’analisi contemporanea di un grosso numero di loci genetici. La scelta degli enzimi di restrizione e dei due-tre nucleotidi arbitrari in terminazione 3' permette una scelta elevatissima di combinazioni che amplificheranno tratti diversi di DNA. Normalmente con una combinazione enzima-estensione si riescono ad amplificare a 10-20 (talvolta 100) frammenti (potenzialmente loci) diversi.
22 PFGE
Il DNA ‘’fingerprinting’’ mediante PFGE è un metodo semplice per confrontare il DNA che prevede la sua frammentazione mediante endonucleasi di restrizione, e la separazione di questi frammenti per poterne misurare il numero e la taglia. Si ottiene cosi un profilo di bande, che somiglia a un codice a barre, che può essere utilizzato come impronta digitale o ‘’fingerprint’’ per riconoscere il batterio.
L’acronimo PFGE raggruppa tute quelle tecniche di separazione di frammenti di DNA che si avvalgono di un campo elettrico la cui direzione rispetto alla matrice solida in cui avviene la migrazione viene variata periodicamente. L’elettroforesi in gel di agarosio convenzionalmente impiega un campo elettrico statico e riesce a separare frammenti di DNA della taglia di 20-50kb.
La PFGE permette di separare frammenti fino a 20 MB sfruttando la lentezze dei grossi frammenti nel riorientarsi ad ogni variazione del campo elettrico, lentezza che è proporzionale alla taglia del frammento.
1.3.1 Identificazione con metodica MALDI TOF
L’identificazione dei microrganismi e solitamente effettuata attraverso la valutazione delle loro caratteristiche fenotipiche quali la crescita su determinati terreni di coltura, la morfologia delle colonie, la colorazione di Gram e alcune reazioni biochimiche. Sebbene queste tecniche permettano l’identificazione della maggior parte dei microrganismi con grande accuratezza, esse sono costose
e prevedono tempi lunghi non idonei all’attività terapeutica del clinico/infettivologo. Un’alternativa a questi metodi è la cosiddetta chemotaxonomy, che teorizza l’individuazione dei batteri attraverso degli identificatori proteici detti markers.
La spettrometria di massa è un potente strumento per realizzare tale metodologia. La spettrometria classica può rilevare masse non oltre i 1000 Dalton, poiché il laser utilizzato sul campione, frammenta le masse più grandi di 1000 Dalton.
La Spettrometria MALDI-ToF (Matrix Assisted Laser Desorption/Ionisation Tine of Flight), supera tale problema utilizzando una matrice per assorbire l’energia del laser ed eliminando, quindi, la possibilità di frammentazione non voluta di masse da rilevare. La Spettrometria MALDI-ToF (Matrix Assisted Laser Desorption/Ionisation Tine of Flight) è stata sviluppata nel 1987 da
23
La tecnologia Matrix-Assisted Laser Desorption Ionization Time-Of-Flight Mass Spectrometry (MALDI-TOF) e attualmente impiegata per l’identificazione di proteine in diversi ambiti della proteomica come l’identificazione di biomarker tumorali, allergici e di patologie degenerative del Sistema Nervoso Centrale quali, ad esempio, l’Alzheimer. In Microbiologia la stessa tecnica si è dimostrata di grande utilità per l’identificazione, in tempi brevissimi, di microrganismi.
Bisogna anche ricordare che questa metodica era già stata utilizzata nella prima metà degli anni ’70 e successivamente abbandonata. Oggi è stata ripresa in considerazione della sua semplicità, economia e rapidità.
Questa tecnica usa una biomolecola immersa in un co-precipitato di una matrice che assorbe raggi UV, colpita da un impulso laser di un nanosecondo.
La maggior parte dell’energia del laser è assorbita dalla matrice, mentre la restante parte ionizza parte del campione. La spettrometria si realizza nelle seguenti fasi:
Le biomolecole ionizzate dall’impulso laser lasciano il campione.
Tali ioni vengono accelerati in un campo elettrico e convogliati in un tubo ( flight tube ).
24
Durante il volo in questo tubo, le molecole sono separate in funzione del loro rapporto massa-carica e raggiungono il rilevatore in momenti diversi, quindi ogni tipo di molecola genera segnali diversi.
I segnali che si ottengono presentano diversi picchi, che rappresentano le abbondanze degli ioni per un dato rapporto massa-carica.
Utilizzando gli spettri di massa è possibile l’analisi dei batteri isolati che utilizza componenti univoci detti biomarkers per differenziare le specie. Le configurazioni spettrali ottenute da intere cellule batteriche presentano delle cosiddette fingerprints, delle impronte digitali. Le fingerprints sono biomarkers che derivano dalla superficie cellulare cioè dalla membrana delle cellule batteriche.
Da questo viene generato un grafico delle proteine dell’intera cellula batterica e confrontato con gli spettri di altri batteri presenti nel database dello strumento. L’unicità di
25
questo grafico permette l’identificazione del microrganismo e, il grado di corrispondenza con il grafico del database, è trasformato in un valore di score che va da 0 a 3.
Questa tecnica è estremamente veloce, infatti, l’identificazione di un microrganismo si ottiene incirca 6 minuti a differenza delle tecniche convenzionali che, invece, per la stessa identificazione richiedono normalmente 18-24 ore.
Questi biomarkers sono relativi a proteine immerse in questa membrana. Attraverso queste proteine è teoricamente possibile individuare anche i ceppi batterici, che si differenziano, in gran parte, nell’interazione con l’ambiente, e l’interazione con l’ambiente avviene soprattutto grazie agli elementi immersi nella membrana cellulare.
La dipendenza dall’ambiente può, però, rendere difficile la riproducibilità di un riconoscimento. Se uno stesso batterio viene trattato in modo diverso, con diversi terreni
26
di coltura e in diversi contesti ambientali il suo spettro può variare sensibilmente. Nella seguente figura si evidenzia l’influenza del terreno di coltura sulla riconoscibilità dei batteri.
Si nota come, con il variare del terreno di coltura i ceppi batterici possono confondersi. Quindi si deve realizzare una serie di norme per standardizzare il trattamento dei batteri in modo che siano riconoscibili attraverso la spettrometria.
1.3.2 Processo di laboratorio
La spettrometria di massa MALDI-TOF è una tecnica analitica che consente di misurare in maniera estremamente accurata il peso molecolare di macromolecole di interesse biologico e di determinare la loro identità in base al rapporto massa/carica.
27 Preparazione del campione
La tecnica prevede che un campione batterico costituito da 10 - 10 cellule, proveniente da una brodocoltura o da una singola colonia, possa essere analizzato ottenendo in qualche minuto uno spettro di massa i cui segnali sono originati da componenti proteiche ribosomiali o loro frammenti rilasciate in seguito a lisi della parete batterica.
Porzioni di colonie fresche isolate, cresciute in piastra secondo protocolli di routine (18-24 ore), vengono deposte con il puntale di una micro pipetta al centro di uno spot di un "target plate" monouso in policarbonato con l’aggiunta di 1µl. di matrice realizzata in materiali organici (acido α-ciano- 4-idrossicinnamico) in grado di fungere da fonte di protoni necessari alla ionizzazione dell’analita in esame. In caso di identificazione di miceti lievitiformi/filamentosi e micobatteri, la procedura differisce da quella standard per l’utilizzo, prima dell’inoculo della matrice, di acido formico o acido trifluoroacetico (TFA) al fine di aumentare l’efficienza di estrazione delle proteine batteriche non facilmente disponibili per l’analisi. Introdotto il "target plate" nell’apposito vano dello strumento, la matrice viene successivamente bombardata in più punti e ripetutamente con un
fascio laser pulsato con frequenza nell’ultravioletto che ne determina la disgregazione e la ionizzazione grazie alla cessione di energia. Il risultato è la disgregazione del campione in numerosissimi frammenti con carica unitaria positiva (cationi monovalenti) costituiti ognuno da porzioni oligopeptidiche associate a porzioni di matrice organica. Una volta vaporizzati (desorbimento), i frammenti vengono accelerati da un campo elettromagnetico adiacente e migrano in senso lineare attraverso il cosiddetto "tubo di volo" per raggiungere e colpire una membrana che
rileverà e registrerà le masse ionizzate impattanti in tempi differenti in base alla massa stessa degli ioni. Mediante il rilevatore di ioni, gli ioni impattanti su di esso vengono misurati nel loro rapporto massa/carica (m/z) potendo così risalire al peso molecolare della molecola analizzata.
Analisi dei risultati
Lo strumento è collegato a un software in grado di acquisire i dati in arrivo, elaborarli in spettri di massa e confrontarli con spettri di riferimento presenti nel database interno. Gli spettri sono acquisiti in modalità lineare positiva nell’intervallo di m/z 2000– 20000. Ogni spettro di massa è acquisito sommando 100 spettri (30 è il numero minimo richiesto) in modo da assicurare una media più che significativa e affidabile degli spettri
28
acquisiti in vari punti della colonia. La banca dati del software contiene spettri di riferimento che rappresentano più di 500 specie/sottospecie di batteri, micobatteri, lieviti e funghi significativamente rappresentativi della popolazione di interesse clinico.
Per la costruzione del database sono stati utilizzati ceppi di origine clinica (80-90%) e ceppi di riferimento ATCC (10-15%). Ognuno degli spettri di riferimento è stato costruito utilizzando da 10 a 20 differenti isolati clinici (cresciuti in differenti terreni di coltura, con range di temperatura e tempi di incubazione differenti) in modo da poter includere negli spettri stessi tutte le possibili varianti di massa derivanti sia da differenze genetiche intra-specie, sia da differenti condizioni di coltura.
Il grado di corrispondenza con lo spettro tipico di ogni microrganismo presente nel database determina l’attribuzione di un cosiddetto Valore di Confidenza che esprime il grado di certezza con cui viene proposta l’identificazione per la specie in esame. Una corrispondenza perfetta tra lo spettro e quello univoco di un singolo organismo, o gruppo di organismi, fornisce una probabilità percentuale del 99,9. Nel caso di una singola specie proposta, se l'intervallo delle probabilità percentuali è compreso tra 60 e 99%, il Livello di
29
Identificazione viene giudicato Buono. Se la probabilità percentuale ottenuta per qualsiasi specie proposta è < 60, l'organismo non viene identificato e si procederà all’identificazione con altre metodiche disponibili in laboratorio
Lo strumento viene calibrato utilizzando come standard un ceppo di Escherichia coli ATCC 8739 che dà origine nello spettro di massa a segnali noti. Il tempo richiesto per la lettura di un singolo "target plate" completo e la trasmissione dei risultati è di circa 50- 60 minuti.
1.3.3 Identificazione delle carbapenemasi
La diffusione di ceppi di Enterobacteriaceae produttori di carbapenemasi è un problema emergente anche in Italia. Gli enzimi più diffusi sono attualmente quelli di tipo KPC, ma sono riportati anche le metallo-b-lattamasi (MBL) di tipo VIM e NDM e la carbapenemasi di tipo OXA-48. La specie maggiormente interessata è Klebsiella pneumoniae.
I laboratori di Microbiologia Clinica devono acquisire dimestichezza nel corretto riconoscimento di tali ceppi, sia per il loro elevato impatto clinico sia per l’importanza che ciò riveste al fine di implementare adeguate misure di infection control. Attualmente la conferma fenotipica della produzione di carbapenemasi nelle Enterobacteriaceae rappresenta uno dei maggiori problemi di interpretazione dell’antibiogramma. A tutt’oggi non esiste un unico test fenotipico risolutivo per
tutti i casi possibili ma l’utilizzo combinato di più test permette di riconoscere questo meccanismo di resistenza con buona sensibilità e specificità.
In linea teorica, la produzione di carbapenemasi andrebbe sospettata in tutti gli isolati di Enterobacteriaceae per i quali le MIC dei carbapenemi risultano superiori ai rispettivi cut-off epidemiologici (ECOFF) dei ceppi wild-type della specie corrispondente. I breakpoint clinici dei carbapenemi, tuttavia, sono più elevati dei valori di ECOFF, ed i sistemi utilizzati nella pratica di laboratorio per determinare la sensibilità agli antibiotici non consentono in genere di misurare valori di MIC dei carbapenemi nel range degli ECOFF.
Tenendo conto di ciò, nella comune pratica di laboratorio è consigliabile sospettare la produzione di carbapenemasi in presenza di una ridotta sensibilità al meropenem: MIC ≥ 0.5 mg/L o diametro alone di inibizione ≤ 25 mm.
I test fenotipici che possono essere utilizzati nella attività diagnostica di routine per la conferma della produzione di carbapenemasi nelle Enterobacteriaceae sono:
30
Test di sinergia con acido boronico: Si basa sul potenziamento dell’azione in vitro dei carbapenemi per inibizione delle carbapenemasi KPC da parte dell’acido boronico, valutabile mediante metodica “di combinazione” su dischetto.
Test di sinergia con EDTA o acido dipicolinico: Si basa sul potenziamento dell’azione in vitro dei carbapenemi per inibizione delle MBL da parte dell’EDTA o dell’acido dipicolinico, valutabile mediante metodica “di combinazione” su dischetto o su striscia a gradiente di diffusione (EDTA).
Test di Hodge (variamente modificato): Si basa sulla riduzione dell’attività del carbapenemico saggiato nei confronti di un ceppo indicatore sensibile mediata dalla carbapenemasi prodotta dal microrganismo in esame.
Test di combinazione su dischetto
Quando si sospetta la presenza di una carbapenemasi è consigliabile saggiare il ceppo contestualmente con il test di sinergia con acido boronico e quello con EDTA o acido dipicolinico utilizzando gli appositi dischetti commercialmente disponibili.
Posizionare su una piastra di Mueller-Hinton agar, preventivamente seminata con il ceppo da saggiare (sospensione 0,5 McFarland in soluzione fisiologica) i seguenti dischetti:
Meropenem
Meropenem + acido boronico Meropenem + acido dipicolinico
Incubare in aria ambiente a 35 ± 2°C per 18-24 ore. Viene considerato significativo un aumento dell’alone di inibizione del meropenem ≥ 5 mm.
La sinergia con acido boronico (BOR) è indicativa della produzione di KPC (Fig.7) o, più raramente, di altre carbapenemasi di classe molecolare A.
La sinergia con acido dipicolinico (DPA) come quella EDTA è indicativa della produzione di MBL (es. VIM o NDM).
31
Figura 2K. pneumoniae KPC+
Test di combinazione su striscia a gradiente di diffusione
Attualmente sono reperibili in commercio solo strisce predisposte per saggiare le combinazioni imipenem + EDTA (consigliato per Gram – non Enterobacteriaceae) o meropenem + EDTA (consigliato per le Enterobacteriaceae) .
Posizionare la striscia di meropenem + EDTA su una piastra di Mueller-Hinton agar preventivamente seminata con il ceppo da saggiare (sospensione 0,5 McFarland in soluzione fisiologica) e lasciata asciugare per 15 minuti dopo l’inoculo del campione. Incubare in aria ambiente a 35 ± 2°C per 16-20 ore.
Interpretazione:
Ceppo produttore di MBL:
rapporto tra MIC meropenem e MIC meropenem in presenza di inibitore ≥8, oppure MIC meropenem – MIC meropenem +EDTA ≥ 3 concentrazioni al raddoppio, oppure presenza di “zona fantasma”, oppure presenza di deformazione dell’ellisse per meropenem (fig. 2).
Test di Hodge
Di questo test sono state proposte numerose varianti metodologiche, per lo più sulla base delle molecole di carbapenemico utilizzate e della modalità di inoculo del ceppo da saggiare (per strisciamento superficiale o per infissione “a taglio” nell’agar).