CAPITOLO 1
1. Introduzione
1.1. Rizosecrezione
Le radici della piante producono e secernono composti naturali nella rizosfera. Circa il 10 % del carbonio fissato fotosinteticamente viene secreto dalle radici (Johansson, 1992; Shepherd e Davies, 1993). Alcune delle sostanze secrete hanno un ruolo importante in vari processi biologici: ad esempio, i flavonoidi e gli isoflavonoidi presenti negli essudati radicali di alcune leguminose attivano in Rhizobium i geni responsabili del processo di nodulazione (Peters e Long, 1988 ; Maxwell e Phillips, 1990). Si pensa che i composti secreti dalle radici delle piante abbiano una grande varietà di attività biologiche, che includono funzioni nella difesa contro stress biotici ed abiotici (Gagnon e Ibrabim, 1997). Ogni specie vegetale secerne un’insieme diverso di composti. All’interno della stessa specie il tasso di secrezione varia da sostanza a sostanza (Deneck et al., 1990).
Lo studio degli essudati radicali potrebbe rivelarsi utile per industria farmaceutica, la quale dipende ancora in buona parte dalle piante come fonte di nuove molecole. La produzione di metaboliti dalle piante può essere effettuata con varie metodiche che vanno dalla coltura di cellule alla produzione di piante transgeniche. La coltura di tessuti in vitro fornisce una produzione migliore rispetto alla coltura della pianta intera, ma risulta costosa e lenta e la produzione di metaboliti secondari può essere problematica per la natura indifferenziata delle cellule in vitro. La coltivazione delle piante che producono proteine ricombinanti offre invece il vantaggio di produrre una massa vegetale maggiore ma oltre ai problemi legati alla coltivazione di piante geneticamente modificate in pieno campo, restano da risolvere i problemi relativi all’isolamento delle sostanze di interesse poiché è necessario frammentare e distruggere il tessuto vegetale visto che la sostanza si trova all’interno del tessuto: quindi una coltura produce solo una volta. Rimane anche il problema che la sostanza di interesse va purificata da un tessuto o da un succo biochimicamente complesso –
un processo laborioso e costoso che risulta non proficuo per produzione su grande scala. La rizosecrezione in coltura idroponica potrebbe essere un’alternativa interessante perché è una forma di produzione che continua per tutto il ciclo biologico della pianta e quindi produce rese maggiori. In più gli essudati radicali sono miscele relativamente semplici rispetto al succo estratto da tessuti vegetali, fatto che facilita la purificazione di eventuali molecole di interesse (Gleba et al., 1999). In particolare, in uno studio effettuato su 120 specie vegetali, si è visto che gli essudati radicali non contenevano tannini e pigmenti e non contenevano grandi quantità di componenti strutturali biologicamente inerti (Gleba et al., 1999).
La coltura idroponica presenta il vantaggio di essere un metodo già testato e sviluppato su scala industriale. In alternativa alla coltura della pianta intera in soluzione idroponica, sono anche stati proposti sistemi di coltura di cellule in sospensioni (Firek et al. 1993; Doran, 2000) e di coltura in vitro di hairy roots (“radici pelose”). Il fenotipo Hairy root è in realtà una malattia causata dal batterio gram-negativo Agrobacterium rhizogenes il quale induce una proliferazione massiccia delle radici, con formazione di numerosi peli radicali intorno al sito d’infezione. La proliferazione delle radici è determinata da 4 geni (rolA, rolB, rolC e
rolD) che si trovano sul T-DNA del plasmide Ri (Root-inducing) (Schmulling et al.,
1998). Il T-DNA del plasmide Ri viene integrato nel genoma della pianta e viene espresso, portando alla formazione di hairy roots (Chilton et al., 1982). L’induzione di hairy roots può essere effettuato in organi della pianta diversi dalle radici, ad esempio si possono formare a partire da dischi fogliari, organi di riserva, ipocotili e fusti (Golds et al., 2001).
Oltre a composti chimici a basso peso molecolare le radici secernono anche proteine. Ad esempio grandi quantità della fosfatasi acida vengono secrete dalle radici di molte piante in condizioni di carenza di fosforo (Li e Tadano, 1996). Questa caratteristica delle piante potrebbe essere sfruttata per ottenere la rizosecrezione di proteine ricombinanti da piante transgeniche. In un esperimento (Gleba et al., 1999) sono riusciti a far secernere nel mezzo di coltura di Nicotiana tabacum la GFP (Green
Fluorescent Protein) della medusa Aequorea victoria, la SEAP (Secreted Alcaline Phosphatase) di placenta umana, e la xylanasi del batterio termofilo Clostridium thermocellum. Tutte e tre le proteine sono state rizosecrete da piante trasformate con i
geni inseriti sotto il controllo trascrizionale di promotori radice specifici e con sequenze contenenti il peptide segnale di secrezione.
Le piante sono organismi ideali per la produzione di proteine ricombinanti per la loro facilità di trasformazione e coltivazione (Giddings et al., 2000; Daniell et al., 2001). Le piante sono organismi eucarioti e le loro cellule sono in grado di processare correttamente i polipeptidi, formare ponti disolfuro, assemblare complessi multimerici, effettuare modifiche post-traduzionali e destinare le proteine alla secrezione. La produzione di proteine con cellule animali e batteriche può comportare la contaminazione con virus e prioni, mentre con le piante questo rischio non esiste. (Gleba et al., 1999; Gaume et al., 2003)
1.2. La secrezione a livello cellulare
Nella via metabolica secretoria le proteine migrano dal reticolo endoplasmatico (ER) all’apparato di Golgi e quindi dall’apoplasto al vacuolo (Vitale e Denecke, 1999). Una volta attraversato il plasmalemma le proteine si trovano nell’apoplasto delle radici che è in contatto diretto con le soluzioni esterne del terreno o del mezzo di coltura. La traslocazione attraverso la membrana dell’ER è il primo passo della via secretoria ed è anche il primo passo di altre viemetaboliche di trasporto che portano a destinazione finale proteine che devono finire in diversi sub-compartimenti cellulari incluso il vacuolo e l’apparato di Golgi (Walter e Lingappa, 1986). Le proteine cominciano la loro sintesi nel citoplasma e iniziano il loro movimento verso la secrezione grazie al riconoscimento del loro peptide segnale da parte dell’SRP (particella di riconoscimento del segnale). L’SRP si lega al peptide segnale mentre il polipeptide in via di formazione si trova ancora sul ribosoma. L’intero complesso SRP-ribosoma trasloca verso il recettore dell’SRP, situato sulla membrana dell’ER e
vi si lega. In questo modo il ribosoma viene trovarsi in vicinanza del poro translocan
Sec61 (Vitale e Denecke 1999;). Il ribosoma quindi si attacca al poro e il polipeptide
viene direttamente maturato nell’ER mentre si sta ancora formando. Dopo l’inserzione nell’ER, il peptide segnale viene rimosso dall’enzima SPP (signal
peptide peptidase) (Vitale e Denecke 1999; Reynaud e Simpson 2002)
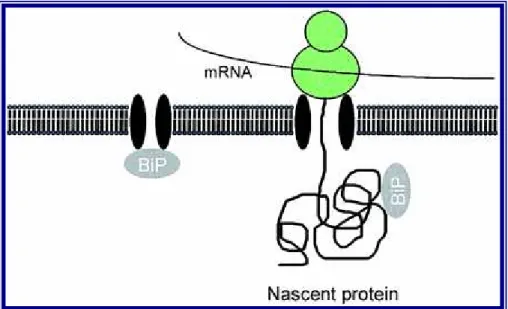
La figura 1 rappresenta la membrana rugosa dell’ER contenente a sinistra un poro al riposo, e destra un poro funzionante. Il poro al riposo viene tappato all’interno da BiP che è una proteina chaperone (HSP70). Quando un ribosoma si lega al poro dal lato esterno, il poro si dilata ed il BiP si stacca. Il ribosoma tappa integralmente il poro in modo da conservare l’integrità del lumen dell’ER. Il peptide in via di formazione
attraversa il poro e inizia a ripiegarsi, probabilmente con l’aiuto di BiP. L’ER ha il funzione di assicurare il corretto ripiegamento ed assemblaggio delle proteine neosintetizzate, ma anche di ripiegare proteine denaturate o nella conformazione sbagliata e anche di degradare le proteine malformate, se non possono essere “riparate”. Quindi possiamo dire che nell’ER viene operato un sistema di controllo qualità delle proteine, per assicurare che le proteine malformate non vengano portate a destinazione (Vitale e Denecke 1999;). Le condizioni nel lumen dell’ER favoriscono la formazione della struttura terziaria e quaternaria delle proteine. Il pH
Figura 1 : Traslocazione di una proteina in fase di sintesi attra- verso la membrana del reticolo endoplasmatico. Da: Vitale e Denecke, 1999
del lumen risulta vicino alla neutralità, e quindi è simile al pH del citoplasma (Kim et
al. 1998), ma la alta proporzione di glutatione ossidato rispetto al glutatione ridotto
crea un ambiente ossidativo in cui è favorita la formazione di ponti disolfuri (Tu e Weissman 2004). È stato dimostrato che il ripiegamento delle proteine richiede energia sotto forma di ATP (Braakman et al 1992). Le proteine iniziano a ripiegarsi mentre stanno ancora attraversando la membrana dell’ER. Alcune proteine solubili si ripiegano facilmente, altri hanno bisogno dell’aiuto di chaperoni per arrivare alla conformazione finale giusta. (Jun-Chao et al. 2006)
I chaperoni e gli enzimi che partecipano al ripiegamento si trovano spesso in complessi multimerici e agiscono in modo coordinato e con la partecipazione di cofattori (Kleizen e Braakman 2004). Cambiamenti nella concentrazione di Ca2+, Zn2+ e ATP nel lumen dell’ER potrebbero influire sulla formazione dei complessi dei chaperoni e quindi influire sulla la loro attività di assistenza al ripiegamento (Trombetta e Parodi 2003) In fig. 2 sono rappresentate due classi di chaperoni molecolari attivi nel sistema di controllo della qualità nel ER, Calnexina/Calreticulina. Ci sono altri chaperoni presenti nel reticolo, incluso BiP, le proteine regolate da glucosio, GRP 78 e GRP 94, e la PDI (proteina disolfuro isomerasi).
I primi tre sono membri della famiglia delle HSP. La PDI può aiutare la formazione di ponti disolfuro calnexina/calreticulina. La calnexina e la calreticulina sono chaperoni molecolari coinvolti nel ripiegamento, assemblaggio e ritenzione/rilascio
delle proteine. La calreticulina lega ioni Ca2+ nel lumen dell’ER. È costituita da 3 domini: due di questi, il dominio N (N terminale) ed il dominio P (centrale, residui 181 – 290), insieme sono responsabili per l’attività chaperone (Jun-Chao et al 2006). Il dominio P contiene una regione ricca di prolina e determina una struttura a forma di braccio che interagisce con altri chaperoni molecolari. Anche la calnexina, che è una proteina di membrana, possiede una struttura a forma di braccio curvo che probabilmente contiene siti di legami con carboidrati. (Jun-Chao et al. 2006).
La calreticulina riconosce il glucosio terminale e 4 mannosi interni dell’oligosaccaride (Glc1-Man9-GlcNAc2) che si trova legato a glicoproteine neosintetizzate (Michalak et al. 2002). Sia calreticulina che calnexina si comportano come lectine, legandosi a proteine monoglicosilate e si associano con la tiolo-ossidoreduttasi Erp57 – una proteina di tipo disolfuro isomerasi residente nell’ER,
Figura 2 : Struttura tridimensionale delle proteine Calnexina e Calreticulina. Fonte : http://www.glycoforum.gr.jp/science/word/qualitycontrol/QS-A01E.html
che favorisce la formazione e isomerizzazione di disolfuri nelle glicoproteine (High, S et al. 2000). Calreticulina, calnexina e Erp57 sono tre componenti che costituiscono il ciclo calnexina/calreticulina (nelle fig. 3 e 4 è mostrato il ciclo).
Le proteine in fase di ripiegamento vanno incontro a eventi di de-glicosilazione e ri-glicosilazione. Lo stato di glicosilazione determina la loro riconoscibilità da parte dei chaperoni. Una serie di enzimi sono coinvolti nella regolazione dello stato di glicosilazione delle proteine nel lumen dell’ER. Il glucosio terminale viene rimosso dalla glicosidasi. II (GLS II). Una uridina difosfato (UDP-glucosio glicoproteina transferasi o UGGT) ha il compito di ri-glicosilare le catene che erano state de-glicosilate dalla glicosidasi II. Ma la UGGT ri-glicosila le proteine solo se le riconosce in una conformazione non piegata o non piegata correttamente. In questo modo si trovano di nuovo con l’oligosaccaride (Glc1Man9GlcNAc2) completo tornano nel ciclo di ripiegamento essendo di nuovo in grado di interagire con calnexina e calreticolina. Successivamente la GLS II rimuove il glucosio terminale dell’oligosaccaride, e così facendo stacca la glicoproteina dal suo chaperone. Questo processo di legame/rilascio può ripetersi diverse volte fino ad arrivare alla conformazione finale della proteina matura. (Roth et al., 2003). La calreticulina è essenziale per il funzionamento della calnexina. Se non è presente la calreticulina, i substrati della calreticulina non vengono portati alla calnexina e si accumulano nel
lumen dell’ER. Gli chaperoni molecolari del ciclo calreticulina/calnexina hanno
n
Figura 3 : Gli enzimi coinvolti nel ciclo calnexina/calreticolina.
Fonte : http://www.glycoforum.gr.jp/science/word/qualitycontrol/QS-A01E.html
Figura 4. Polisaccaridi legati alle proteine durante il ripiegamento. Fonte : http://www.bijvoet-center.nl/cpc/research/CNX
1.3. Trasporto di proteine dall’ ER
La maggior parte del trasporto dall’ER avviene in vescicole inglobate in un rivestimento proteico. Esistono almeno 3 tipi di rivestimento delle vescicole coinvolti nel trasporto tra componenti del sistema endomembrana, costituiti dai complessi proteici Clathrin, coat protein I (COPI) e coat protein II (COPII) (Scales et al. 2000). Le vescicole con rivestimento Clathrin sono attive nell’endocitosi e nel trasporto biosintetico dal Golgi verso gli endosomi. Le vescicole con rivestimento di tipo COPI portano materiale dal Golgi verso il ER. Sono le vescicole con rivestimento di tipo COPII che trasportano le proteine neosintetizzate dall’ER al Golgi (Glick 2001). Le vescicole clathrin esistono in almeno due forme diverse, e lo stesso è probabilmente vero anche per le vescicole COPI e COPII (Muñiz et al. 2001).
Se servono vescicole di diversi tipi, è in parte perché il materiale trasportato varia in natura.
Le vescicole COPII mediano il trasporto di specifici gruppi di proteine selezionate. Mentre alcune proteine sembrerebbero uscire dall’ER in una via di trasporto aspecifica (Martinez-Menárguez et. al. 1999), la maggior parte delle proteine vengono selezionate tramite interazioni specifiche con subunità presente sulla membrana delle vescicole, oppure con recettori o appositi adattatori (Herrman et al. 1999).
1.4. Impacchettamento delle proteine cargo
Le proteine da impacchettare comprendono proteine di vari tipi (proteine cargo), incluso quelle solubili, quelle che attraversano la membrana una volta, e quelle che attraversano la membrana più volte (Glick, 2001). Un metodo per trasportare proteine di diversa natura, è di caricarle con meccanismi diversi. Le proteine da trasportare devono essere riconosciute in qualche modo. Un sistema di riconoscimento usato dalle vescicole COPII sfrutta alcune delle proteine presenti nella membrana dell’ER. Ad esempio, nel lievito Saccharomyces cerevisiae, la Shr3p
riconosce gli enzimi amminoacido-permeasi neo-sintetizzati presenti nel lumen dell’ER e li porta alla superficie di vescicole COPII nascenti (Gilstring et al. 1999). Erv14 è un altro fattore che accompagna proteine di membrana Axl2p neo-sintetizzate alle vescicole COPII (Powers e Barlowe 1998). Shr3p si stacca dalla sua proteina cargo prima che entri nella vescicola e rimane nell’ER. Erv14 invece accompagna la sua cargo fino all’apparato di Golgi, poi, arrivata a destinazione, rilascia la cargo e ritorna all’ER.
Un altro metodo per esportare proteine di diversa natura, è con l’utilizzo di diversi varianti delle vescicole COPII. È interessante il caso delle proteine agganciate alla membrana con un gruppo glycosyl-phosphatidy-linositol (GPI). Un gruppo GPI viene attaccato a proteine neo-sintetizzate nell’ER portando alla formazione di molecole ibride in cui il polipeptide si affaccia al lumen dell’ER, mentre la parte acido grasso del GPI si incorpora nella membrana. È stato visto che le proteine che legano GPI potrebbero uscire dall’ER seguendo un'altra via metabolica.
L’esistenza di zone eterogenee sulla membrana dell’ER potrebbe spiegare perché lo stesso tipo di involucro vescicolare (in questo caso COPII) può generare più tipi di vescicole. Sulla membrana dell’ER si creano zone contenenti sfingolipidi dette “zattere” (Simons e Ikonen 1997).
Nella figura 5, a sinistra è mostrata una vescicola che si sta formando in una zona dove non è presente una zattera di sfingolipidi (in grigio), mentre sulla destra è mostrata una vescicola che si sta formando contenente una zattera ( in rosso ). Alcune proteine transmembrana (viola) sono quasi assenti nelle zattere, mentre le proteine GPI (in blu) si trovano quasi esclusivamente in queste zone (Glick, 2001).
Le proteine transmembrana, ad esempio Emp24p (in verde) che funziona da recettore delle proteine cargo, sono richieste per legare l’involucro alla membrana (Glick, 2001).
1.5. I chaperoni molecolari.
I chaperoni vengono definiti come “una classe di proteine che aiutano il ripiegamento non covalente di polipeptidi in vivo ad una conformazione biologicamente corretta. I chaperoni non fanno parte della struttura finale e possono agire sia in maniera catalitica, accelerando o decelerando le reazioni chimiche, che non catalitica, inibendo il verificarsi di reazioni sbagliate.” (Evstigneeva et al., 2001). Altri autori definiscono il chaperone come “una proteina che, in modo temporaneo, si lega ad un’altra proteina per stabilizzarne la sua conformazione instabile e per determinare l’attività di quella proteina.” (Agashe e Hartl, 2000).
I chaperoni molecolari sono coinvolti in molti processi cellulari, incluso il corretto ripiegamento di polipeptidi neosintetizzati, (Beckmann et al., 1990) e di proteine denaturate (Hartl, 1996). Oltre a partecipare al ripiegamento, i chaperoni hanno anche altre funzioni: possono stabilizzare le proteine e le membrane (Wang et al., 2004), mantenere le proteine in strutture “traslocazione competenti” per permettere a queste proteine di attraversare le membrane degli organelli (Chirico et al., 1988; Deshaies et
Figura 5 : Le zone di eterogeneità sulla membrana dell’ER generano vescicole di diversi tipi. Fonte : Glick, B. S., 2001
al., 1988), possono fornire l’energia che permette agli organelli di importare
precursori proteici (Berthold et al., 1995) ed aiutano ad assemblare e disassemblare complessi proteici oligomerici (Ellis e Van der Vies, 1991). Sono anche state implicate nella degradazione proteica (Agarraberes e Dice, 2001; Tatsuta et al., 2000). I chaperoni si trovano sia nel citoplasma che negli organelli (nucleo, mitocondri, cloroplasti) e nell’ER.
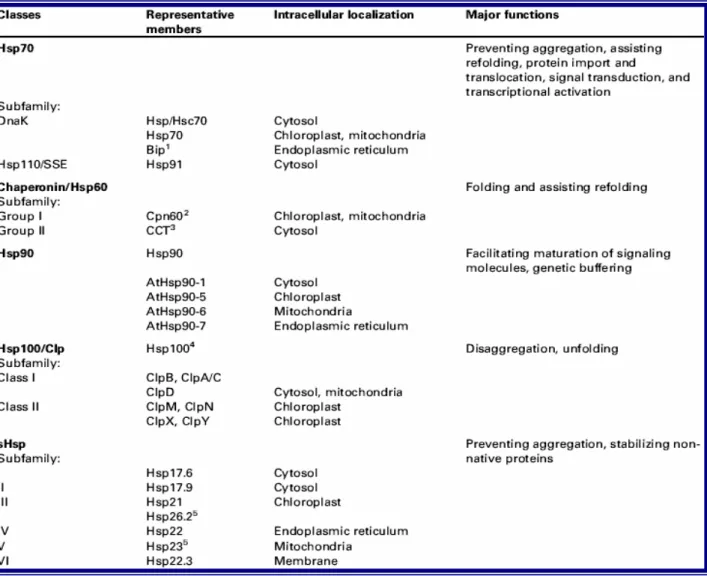
Una classe di chaperoni molecolari è rappresentata dalle HSPs (Heat Shock Proteins), perché essi sono stati scoperti come proteine indotte da stress termico; comunque non tutte le HSP sono chaperoni e non tutti i chaperoni sono HSP. Le HSP rappresentano una classe di proteine estremamente conservata che ritroviamo in tutti gli organismi viventi. Le HSP70 sono divise in 5 grandi famiglie in base al loro peso molecolare HSP100 (Clp), HSP90, HSP70 (DnaK,), Chaperonine (GroEL e HSP60) e Small HSP (sHSP)) secondo Wang et al. (2004), come mostrato in tabella 1.
Inoltre le HSP vengono distinte anche in HSP induciblili da stress e HSP costitutive, queste ultime sono denominate HSC (Heat Shock Cognate) e sono normalmente presenti a bassi livelli nelle cellule non sottoposte ad alcun tipo di stress. ERLe HSP/chaperoni sono coinvolte nella omeostasi cellulare sia in condizioni normali che in condizioni di stress (Wang et al., 2004) grazie alla presenza della forma costitutiva e alla forma inducibile.
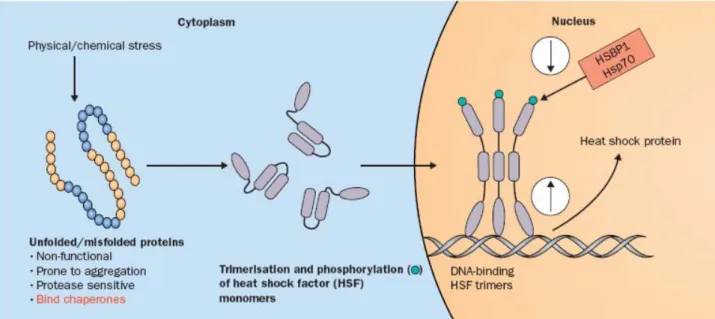
L’espressione della forma inducibile aumenta non solo in risposta al caldo, ma anche in risposta ad altri stress abiotici come stress idrico, osmotico, ossidativo e da freddo. (Waters et al., 1996; Vierling, 1991). La regolazione della trascrizione delle HSP viene svolta da fattori di trascrizione chiamati HSF (Heat Shock Factors). Uno di questi – il fattore HSF1 – è presente in condizioni normali nel citoplasma in forma inattiva. In condizioni di stress, HSF1 percepisce l’aumento delle proteine non native e viene fosforilato. Gli HSF1 si aggregano quindi per formare omotrimeri che hanno la capacità di legarsi al DNA. I trimeri traslocano nel nucleo dove interagiscono con regioni specifiche nei promotori delle HSP, per regolare la loro espressione (Fig. 6) (Pockley, 2003). Le HSP/chaperoni interagiscono con una larga gamma di proteine co-chaperoni che regolano la loro attività o partecipano al ripiegamento di alcuni substrati (Hartl, 1996; Frydman, 2001). Oltre a svolgere una funzione diretta nella
risposta a stress, le HSP/Chaperonine interagiscono con altri meccanismi di risposta a stress e insieme agiscono sinergicamente per ridurre i danni alle cellule (Wang et al., 2004).
1.6 Reti metaboliche di HSP
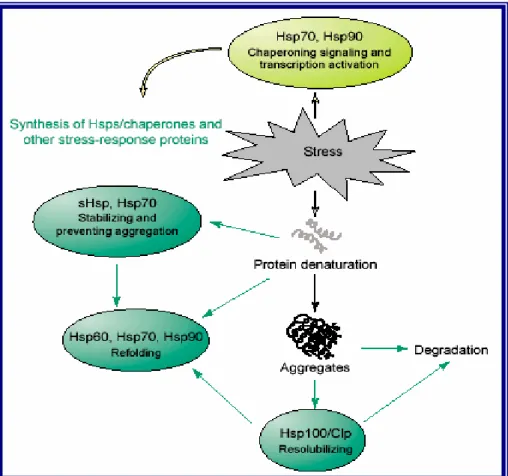
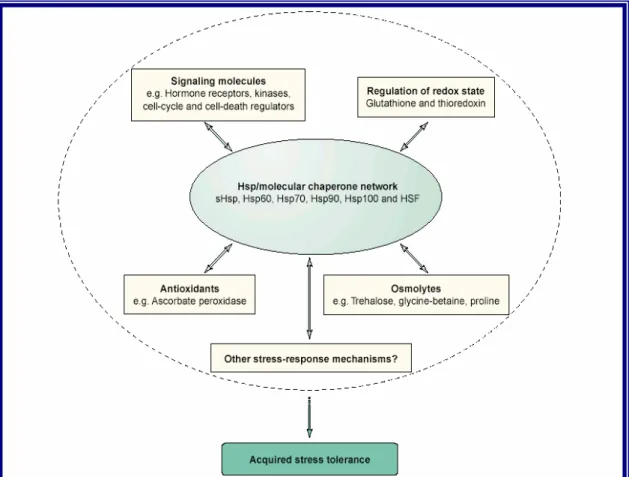
Le HSP contribuiscono al mantenimento dell’omeostasi cellulare con le loro attività svolte singolarmente, o in cooperazione con altre HSP. In Figura 7, è riportata una rete d’interazione delle HSP caratterizzata dalla cooperazione di classi diverse di HSP nella protezione delle proteine e nel ripristino dell’omeostasi cellulare negli organismi viventi in condizioni di stress abiotico (Wang et al., 2004). cellulare e della morte cellulare, dimostrando il loro ruolo chiave nei sistemi di trasduzione dei segnali cellulari (Ellen et al., 2002).
Questa rete di attività coordinata tra le differenti famiglie di HSP sembrerebbe essere collegata ad altri meccanismi di difesa adottati dalle piante nella risposta allo stress come illustrato in Figura 8. Le HSP cooperano infatti con un gruppo di composti organici a basso peso molecolare che si accumulano negli organismi in risposta a stress osmotici (Singer e Lindquist, 1998). Oltre a questa cooperazione, è ben documentata l’interazione delle HSP con numerose molecole segnale, che comprendono recettori degli ormoni nucleari, tirosine- e serine/treonine- chinasi, regolatori del ciclo
Figura 7 : Meccanismo di cooperazione tra differenti famiglie di HSP/chaperone nel mantenimento dell’omeostasi cellulare in condizioni di stress (Wang et al., 2004).
1.7 HSP 70
I membri di questa famiglia presentano un sito di legame per l’ATP e hanno la capacità di accoppiare l’idrolisi dell’ATP e il rilascio delle loro proteine substrato, di solito con la collaborazione di cofattori (Agashe e Hartl, 2000). Queste proteine si trovano ubiquitariamente in tutti batteri tranne alcuni Archea. Negli eucarioti si trovano nel citosol, nel nucleo, in mitocondri e cloroplasti e nel ER. (Craig, et. al., 1993). Oltre a contribuire al corretto ripiegamento delle proteine, le HSP70 sono coinvolte in diverse attività cellulari, come la degradazione di proteine instabili, la riorganizzazione di componenti del citoscheletro, l’inizio della traduzione, l’ingresso e l’uscita di proteine nucleari, l’assemblaggio dei ribosomi e la sintesi del DNA
Figura 8 Interazione tra il sistema HSP/chaperone e altri meccanismi di difesa adottati dalle piante nell’acquisizione della tolleranza allo stress (Wang et al., 2004).
(Forreiter e Nover, 1998). Le HSP70 sono in grado di veicolare le proteine instabili in proteosomi o lisosomi per degradarle (Hartl, 1996). Alcuni membri delle HSP70 sono coinvolti nel regolare l’attività biologica di proteine regolatrici e potrebbero fungere da repressori negativi di fattori di trascrizione che rispondono allo stress da calore (Wang et al., 2004). I ruoli specifici delle diverse HSP70 dipendono dal posizionamento subcellulare e dall’espressione differenziale a diversi stadi di sviluppo oppure dall’interazione con diversi gruppi di proteine. Per esempio l’Hsc70 citosolico inibisce l’aggregazione di proteine, favorisce il ripiegamento di proteine e mantiene alcune proteine in uno stato che permette loro di essere importate negli organelli, mentre le HSP70 del mitocondrio e del cloroplasto sono coinvolte nell’importo e nella traslocazione di precursori di proteine (Wang et al., 2004).
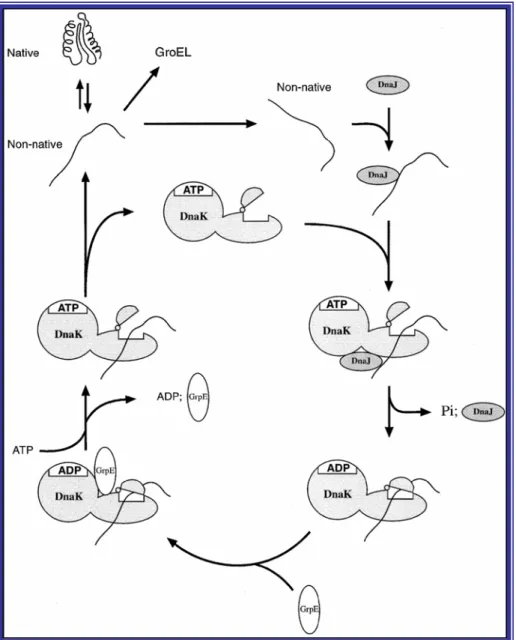
I membri della famiglia HSP70 sono altamente conservati, mostrando una percentuale di identità tra le cellule eucariotiche compresa tra il 60-78% ed un’identità del 40-60% tra l’HSP70 eucariotica e l’omologa DnaK di Escherichia coli (Caplan et al., 1993). Strutturalmente le HSP70 hanno due domini funzionali: un dominio N-terminale ATPasi (44 kDa) e un dominio C-terminale di legame a peptidi (25 kDa), quest’ultimo contiene un subdominio di tipo β-foglietto che lega peptidi e un sub-dominio α-elica che agisce da coperchio che copre il sito di legame con il peptide (Fig. 9).
Tutte le HSP70 hanno la capacità di legare corte sequenze di aminoacidi. (circa 7). L’elemento chiave del riconoscimento da parte delle HSP70 è l’affinità per specifiche regioni idrofobiche ridondanti, che sono generalmente esposte su proteine non native, ad esempio su catene nascenti su ribosomi (Agashe e Hartl, 2000). L’HSP70 mantiene in forma distesa il peptide e il ripiegamento avviene solo quando il peptide viene rilasciato. Dopo il rilascio dall’HSP70, la proteina ha la possibilità di ripiegarsi spontaneamente, di essere trasferita ad un altro chaperone o rilegarsi di nuovo all’HSP70, questo può essere importante per il polipeptide nascente quando la catena
Figura 9 : Modello di funzionamento dell’HSP70 DnaK batterica in coordinazione con GroEL (HSP60) e DnaJ (HSP40) (Agashe e Hartl, 2000).
non è ancora completa e non è ancora in grado di ripiegarsi nella struttura giusta in modo stabile (Agashe e Hartl, 2000).
1.8 Le HSP come regolatori del sistema immunitario
Come accennato in precedenza la funzione principale delle HSP è di intervenire nel corretto ripiegamento ed assemblaggio delle catene polipeptidiche nascenti e nel corretto refolding delle proteine danneggiate da stress ambientali. Negli ultimi anni, però, è emerso che le proteine HSP hanno un ruolo anche in alcune patologie, grazie alla loro capacità di legare peptidi antigenici derivati sia da proteine espresse da cellule cancerose, sia da agenti infettivi ad esempio batteri e virus (Li et. al. 2002). Infatti numerosi studi effettuati sulle HSP in modelli animali hanno evidenziato una correlazione positiva tra l’espressione delle HSP e la risposta immunitaria nei confronti di cellule cancerogene. In seguito a queste evidenze sperimentali la ricerca è stata direzionata verso l’utilizzazione delle HSP per la cura di numerose malattie. Fino ad oggi la maggior parte degli studi è stata focalizzata sia sulla loro capacità di regolare la risposta infiammatoria in malattie autoimmuni, sia sulla loro capacità di indurre risposte immuni peptide-specifiche contro i tumori e gli organismi patogeni (Li , 2004)
La base della funzione immunologica delle HSP riflette due proprietà intrinseche: • esse agiscono come adiuvanti generali dell’immunità innata, promuovendo la
maturazione e l’attivazione delle cellule dendritiche
• sono capaci di indurre la stimolazione peptide-specifica dei linfociti T, ma solo interagendo con cellule che presentano specifici recettori sulla superficie, come le cellule dendritiche.
Esempi di queste molteplici attività sono stati riportati in letteratura ed esistono numerosi articoli che riguardano i vari aspetti degli effetti terapeutici di seguito elencati:
• morte diretta o inibizione della crescita dei patogeni;
• citoprotezione mediante induzione delle HSP della cellula ospite;
• induzione delle HSP da parte del patogeno e conseguente stimolazione del sistema immunitario innato, mediante riconoscimento delle HSP del patogeno da parte dei macrofagi.
Ad esempio è stato riportato il ruolo della HSP65 nell’infezione da Toxoplasma
gondii (Hisaeda e Himeno, 1997), un protozoo che in individui sani raramente causa
problemi, ma diventa molto pericoloso in malati di AIDS. Le HSP del parassita hanno la capacità di stimolare i linfociti T dell’organismo ospite, i quali secernono citochine che stimolano i macrofagi (Moré et al. 2001). Inoltre, anche i macrofagi infettati dal protozoo producono proprie HSP che hanno la capacità di stimolare il sistema immunitario.
Un ruolo analogo viene svolto dalla HSP60 di Legionella pneumophyla , da GroEL di
E coli, dalla HSP70 di Mycobacterium tuberculosis e dalla HSP65 di Mycobacterium bovis; queste proteine contribuiscono all’attivazione del sistema immunitario innato
inducendo i macrofagi a produrre le interleuchine IL-α, IL-1β, IL-6 ed il fattore di necrosi tumorale Tn-α (Habich et al. 2003).
Di rilevante importanza è l’effetto citoprotettivo delle HSP. Infatti, è stato riportato che l’induzione delle HSP (in particolare HSP70) conferisce protezione da danni ischemici e neuronali. Topi transgenici che esprimono il gene umano per l’HSP70 sotto il controllo di un promotore costitutivo, sovraesprimono la proteina HSP70 nel cuore e in altri organi; ciò determina una riduzione del tempo di recupero che segue l’episodio ischemico (Jindal 1996).
L’effetto citoprotettivo delle HSP è stato dimostrato anche in topi sottoposti a ipertermia in cui si riscontra un aumento dell’espressione delle HSP70; in questo caso è possibile osservare anche una riduzione dei danni, causati dall’esposizione a luce
intensa, a carico dei fotorecettori. Inoltre, è stato dimostrato il ruolo delle HSP nel ridurre l’entità dei danni che i tessuti subiscono in seguito ai trapianti (Jindal 1996). Un’altra importante funzione delle HSP è quella di fungere da carrier di una ampia varietà di peptidi antigenici, come i peptidi tumorali, virali e batterici. In questo contesto, le HSP hanno un duplice ruolo, all’interno ed all’esterno della cellula. E’ stato inoltre dimostrato il ruolo dell’HSP70 nel presentare gli antigeni tumorali alle molecole del Complesso Maggiore di Istocompatibilità (MHC) di classe Ι, che hanno il compito di esporre gli stessi antigeni sulla superficie cellulare, per essere riconosciuti dai linfociti T citotossici (CTL). Inoltre le HSP agiscono anche come carrier per antigeni esterni alla cellula; infatti, in seguito ad un evento di stress come un tumore o un’infezione, la cellula sintetizza HSP che si legano ai peptidi antigenici, formando il complesso HSP-peptide antigenico. Tale complesso, in seguito a necrosi e successiva rottura della membrana plasmatica, viene liberato nel torrente circolatorio. Questa condizione rappresenta un segnale di allarme per il sistema immunitario. In particolare il complesso HSP-peptide antigenico presente in circolo può penetrare all’interno di una cellula APC (Antigen Presenting Cell, rappresentata da una cellula dendritica) attraverso un processo di endocitosi mediato da recettore. Il legame recettore-HSP è necessario per la maturazione delle cellule dendritiche e determina la produzione di citochine e chemochine. Successivamente il complesso si scinde e l’antigene si lega ad una molecola MHC tramite la quale sarà esposto sulla superficie della cellula dendritica. Solo in questo momento l’antigene può essere riconosciuto dai linfociti T, che verranno attivati contro le cellule che lo espongono (Kammerer et al. 2002).
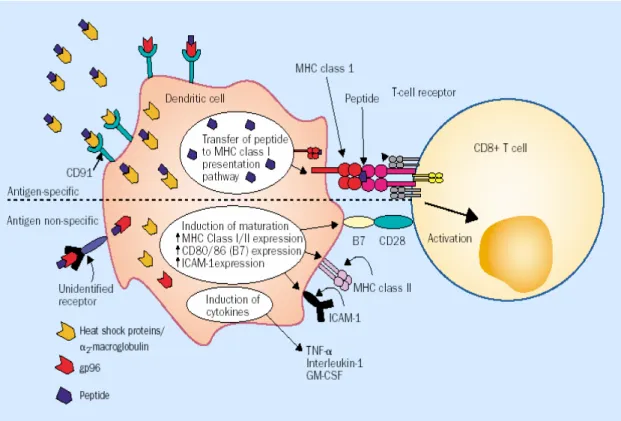
Il caso dell’interazione tra un HSP di 96 KDa, chiamato gp96 e il recettore CD91 è un ottimo esempio di questo meccanismo ed è illustrato nella figura 10. Gp96 – che è riconosciuto come avendo un ruolo attivo nella protezione contro tumori (Chandawarkar et al., 1999) – si lega a peptidi antigenici, e il complesso gp96-peptide viene riconosciuto da CD91 che è un recettore α2-macroglobulinica presente
complesso viene internalizzato e il peptidi antigenico viene liberato e esposto sulla superficie della stessa cellula dove interagisce con linfociti di tipo T-CD8. (Pockley, 2003).
Lo stesso recettore potrebbe legare diverse forme di HSP, ad esempio CD91 riconosce anche altre HSP, incluso la HSP70, HSP90 e la calreticulina (Pockley, 2003), anche se l’affinità di legame varia mentre la HSP60 non lega CD91 ma può legarsi ai macrofagi. Ciò mette in evidenza il ruolo diverso delle varie forme delle HSP che nella immunoregolazione (Habich et al., 2002).
Come è evidente, le HSP rivestono un ruolo fondamentale nel processo di attivazione del sistema immunitario sia innato che adattativo. Per questo motivo si è pensato di utilizzare le HSP sia nella prevenzione, che nella terapia di varie patologie (Jindal 1996).
In particolare è stata sfruttata la capacità delle HSP di legare i peptidi antigenici e permettere così la produzione di anticorpi specifici per una risposta immunitaria specifica. Quantità estremamente piccole di antigene (nano o picogrammi) sono sufficienti a determinare una potente risposta immunitaria, soltanto se l’antigene è legato alla HSP (Audibert 2003). Altri proteine in grado di complessare peptidi antigenici – ad esempio le albumine – non sono immunogeniche.
Numerosi test eseguiti su animali e sull’uomo hanno dimostrato l’efficacia di vaccini costituiti da complessi HSP-peptide estratti e purificati da tumori autologhi nella terapia dei tumori (Castelli et al. 2001). In particolare è stato estratto e purificato un complesso HSP70-peptide da cellule di melanoma umano, che ha dimostrato di avere un ruolo fondamentale nell’attivazione di cloni di linfociti T diretti verso l’antigene tumorale.
In questo contesto è interessante notare che la dose della proteina usata per la terapia ha una importanza fondamentale come dimostrato da gp96 che – da un’azione protettiva a basse dosi – passa ad avere una funzione inibente della risposta immunitaria se somministrata ad alte dosi. Il meccanismo con il quale alte dosi di gp96 inibiscono la risposta immunitaria rimane ancora da chiarire (Pockley, 2003). Inoltre la capacità delle HSP di stimolare la sintesi di citochine, è di fondamentale importanza nella risposta di attivazione immunitaria, dato che senza l’induzione di uno stato infiammatorio mediato dalle citochine, non è possibile avere una attivazione ed una espansione adeguate delle cellule effettrici come i linfociti. Come esempio, prendiamo il caso delle HSP 70, 60 e gp96 – che sono in grado di favorire la maturazione delle cellule ACP tramite l’induzione di una maggior secrezione di
tumor necrosis factor-α, le interleukine (IL) –1β, (IL) – 6 e (IL) –12 dalle cellule di
tipo macrofagi o monociti e dalle cellule dendritiche. (Millar et al., 2003) Così viene stimolata l’espressione di B7 e MHC classe 2 sulla superficie delle cellule dendritiche, la migrazione delle cellule dendritiche verso i linfonodi e la secrezione di chemokina dai macrofagi e dalle cellule dendritiche (Millar et al., 2003).
Tale scoperte avvalorano l’idea che le HSP agiscono da segnale per la maturazione delle APC e che potrebbero avere un ruolo nel determinare l’induzione della risposta immunitaria oppure la tolleranza immunitaria – fattore determinante nello sviluppo delle malattie auto-immune (Millar et al., 2003).
Su queste evidenze sperimentali negli Stati Uniti sono state fondate delle Società (Antigenics) per la purificazione del complesso HSP-antigene tumorale isolate da tumori del rene e melanoma e re-iniettate nello stesso paziente.
La maggior parte degli studi a livello clinico sono stati fino ad ora effettuati con HSP di origine umana. Come accennato in precedenza le proteine HSP sono proteine altamente conservate infatti la similarità tra le proteine umane e vegetali è di circa il 70-85%.
E’ stata anche dimostrata, però, l’attività immunostimolante di HSP eterologhe; ciò probabilmente è dovuto all’alto grado di conservazione di queste proteine. Infatti sono stati verificati numerosi parallelismi tra i meccanismi di resistenza a patogeni presenti nelle piante ed i sistemi di immunità innata degli animali svelando la presenza di una regolazione simile nelle piante e negli animali per fronteggiare l’invasione di patogeni (Owens-Grillo et al. 1996; Holt et al. 2003). In questi studi è stato dimostrato che proteine HSP isolate da piante interagiscono con il complesso delle chaperonine umane e che proteine umane purificate interagiscono con sistema delle chaperonine vegetali, con una completa intercambiabilità, suggerendo che esiste una elevata conservazione della interazione proteina-proteina e del sistema chaperone tra il regno animale e vegetale (Stancato et al. 1996)
Un altro esempio è costituito da uno studio effettuato sui topi, in cui è stato dimostrato l’effetto immunogenico del complesso costituito da HSP70 di
Mycobacterium tubercolosis e da una nucleoproteina (pNP) derivata dal virus
dell’influenza. Ciò ha permesso di sintetizzare in vitro tutta una serie di complessi HSP-antigene per la costituzione di vaccini diretti verso numerose patologie. Le HSP sono dei potenti adiuvanti per vaccini, che però non presentano gli effetti collaterali degli adiuvanti attualmente utilizzati.