1. INTRODUZIONE
1.1 Il sistema nervoso centrale (SNC)
Nei vertebrati il sistema nervoso centrale (SNC) si sviluppa lungo la linea mediana dorsale dell'asse antero-posteriore (A-P) a partire dalla chiusura del tubo neurale, una struttura di origine ectodermica situata dorsalmente alla notocorda. Il primo passo nello sviluppo del futuro SNC è rappresentato dal processo di induzione neurale, ossia la specificazione verso un destino neurale delle cellule dell'ectoderma dorsale. Questo avviene durante la gastrulazione in cui si ha un ispessimento dell'ectoderma nella parte dorso mediale. Le cellule di questa regione formano un neuroepitelio pseudostratificato chiamato neuroectoderma, dal quale si forma la piastra neurale. La piastra neurale si sviluppa in risposta al rilascio di molecole secrete dal mesodema sottostante. La parte anteriore della piastra neurale corrisponde alla regione presuntiva del cervello mentre quella più posteriore alla regione presuntiva del midollo spinale. Durante la neurulazione, la piastra neurale inizia a piegarsi ventralmente lungo la sua linea mediana e a chiudersi dorsalmente per dare origine al tubo neurale, il quale poi risulterà isolato dal restante ectoderma superficiale che invece formerà l'epidermide. Successivamente alla chiusura del tubo neurale nella sua parte anteriore, primordio del futuro cervello, si ha la formazione di tre vescicole distinte da cui prenderanno origine il prosencefalo, il mesencefalo e il rombencefalo.
1.2 Il rombencefalo
1.2.1 Regionalizzazione antero-posteriore
La regionalizzazione antero-posteriore (A-P) della piastra neurale inizia durante la gastrulazione quando le cellule mesodermiche vanno incontro al processo di ingressione che permette interazioni con l'ectoderma soprastante. Nieuwkoop propose un modello per tale regionalizzazione basato su due fasi successive: inizialmente segnali “attivanti” secreti dal mesoderma durante la fase precoce di ingressione inducono nel neuroectoderma una differenziazione neuronale anteriore, successivamente i segnali “trasformanti” che derivano dal mesoderma durante la fase di ingressione tardiva portano ad una differenziazione neurale posteriore (Nieuwkoop, 1952). Marcatori molecolari, individuati nell'organizzatore degli anfibi (organizzatore di Speman), suggeriscono che i meccanismi che permettono la regionalizzazione e l’induzione del neuroectoderma sono eventi conservati nel corso dell’evoluzione. Esistono tre marcatori molecolari che appartengono alla famiglia dei fattori di trascrizione (goosecoid, HNF-3? e LIM-1) e tre appartenenti al gruppo di proteine secrete (follistatin, nogging e chordin) che hanno la capacità di conferire carattere neurale alle cellule ectodermiche.Secondo la definizione di Nieuwkoop questi sono candidati come segnali attivatori, e quindi hanno un carattere anteriorizzante. Questi induttori neurali agiscono antagonizzando i fattori appartenenti alla famiglia dei Bone Morphogenetic Proteins (BMPs), i quali invece impediscono all'ectoderma di intraprendere un destino differenziativo neurale. Per quanto riguarda invece i segnali con carattere posteriorizzante, definiti trasformanti da Nieuwkoop, i candidati sono
rappresentati da proteine secrete che fanno parte sia della famiglia dei geni Wnt (McGrew et al., 1997; McGrew et al., 1995) che della famiglia dei geni FgF (McGrew et al.,1997) ma anche l'acido retinoico (RA), che è prodotto principalmente nell'organizzatore (Hogan et al.,1992; Kraft et al., 1994) e che ha la capacità di posteriorizzare il tubo neurale anteriore.
E' stato osservato che trattando gli embrioni precoci di rana con acido retinoico (AR) si ha come effetto riduzione della parte anteriore del cervello ed espansione della sua parte posteriore (Durston et al., 1989). Nel topo è stato dimostrato che un eccesso di AR ind uce la formazione di strutture posteriori tra la zona caudale del cervello e lo scheletro assiale (Kessel e Gruss, 1991; Marschall et al., 1992). Questi segnali trasformanti agiscono come gradienti (spaziali o temporali) la cui corretta combinazione è fondamentale per garantire la posteriorizzazione del neuroectoderma.
Sotto l'azione di questi segnali, il tubo neurale in via di formazione mostra, in senso rostro-caudale, distinte caratteristiche molecolari che sono alla base della futura suddivisione del SNC. Alcune delle molecole in questione sono, ad esempio, il fattore di trascrizione Emx2, espresso nel prosencefalo, Otx2, espresso nel pro- e mesencefalo, Krox20 e kreisler, espressi nel rombencefalo.
1.2.2 La segmentazione del rombencefalo
La segmentazione rappresenta una strategia di sviluppo che ha avuto molto successo durante l'evoluzione. Questo processo permette di costruire strutture complesse e variabili partendo da singoli elementi omologhi introducendo variazioni
locali con una periodicità caratteristica. L'organizzazione segmentale può essere osservata in numerosi organismi invertebrati, in particolare negli artropodi. Nei vertebrati questo genere di organizzazione è evidente nel mesoderma che dà origine ai somiti Questi sono all'origine dei futuri elementi metamerici delle scheletro assiale, le vertebre. Un'organizzazione metamerica può essere osservata anche a livello del faringe, con la formazione degli archi faringei, del rombencefalo, che a stadi precoci di sviluppo appare segmentato in una serie di compartimenti lungo l'asse A-P, ed a livello dei gangli dei nervi cranici. Questo suggerisce che lo sviluppo di alcune parti del SNC, della testa e del collo avviene attraverso un processo di segmentazione.
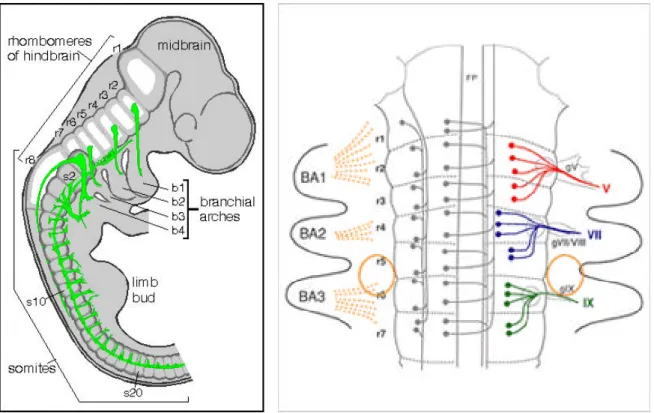
L’osservazione di una suddivisione in segmenti, denominati rombomeri, nella regione del rombencefalo risale al 1828 (von Baer, 1828). I rombomeri, delimitati da costrizioni che appaiono nel neuroepitelio, sono strutture di natura transitoria. Nel topo essi sono otto e sono visibili tra 8.5 e 10.5 dpc (days post coitum) (fig 1).
Figura1: Microfotografia al microscopio a scansione della porzione dorsale di rombencefalo di embrione di topo a stadio 8.5 dpc dello sviluppo in cui è visibile la suddivisione in rombomeri. r1: rombomero 1; r2: rombomero 2
Nel rombencefalo in questo intervallo di tempo si stabiliscono molte strutture di base e si differenziano tipi neuronali distinti. I neuroni che per primi si differenziano
sono quelli della sostanza reticolare che appaiono inizialmente nei rombomeri pari (r2, r4, r6) e, solo più tardi, si ritrovano anche nei rombomeri dispari (Lumsden e Keynes, 1989). Alla fine ogni rombomero contiene distinti tipi di neuroni reticolari (fig 4; Clarke e Lumsden, 1993). Grazie all’analisi dello sviluppo dei motoneuroni rombencefalici branchiali, effettuata nel pollo, è stato messo in evidenza che i nervi trigemino (V), facciale (VII) e glossofaringeo (IX) originano rispettivamente dai rombomeri 2, 4 e 6 (r2, r4 e r6) (fig 2). Ciò ha permesso di dimostrare la presenza di un’organizzazione metamerica del rombencefalo (Lumsden e Keynes, 1989; Simon e Lumsden, 1993). Successivi studi di marcatura retrograda, volti ad individuare i corpi cellulari dei motoneuroni che compongono i nervi cranici, hanno messo in evidenza un’organizzazione ripetitiva basata su una cooperazione tra due rombomeri successivi: la componente motoria che forma un nervo riceve gli assoni che originano sia dal rombomero dal quale uscirà il nervo, sia dal rombomero successivo. Così la componente motoria del nervo trigemino è formata da assoni che hanno origine sia da r2 che da r3, quella del nervo facciale è composta da assoni che originano da r4 e r5, e quella del nervo glossofaringeo è formata da assoni che derivano da r6 e r7 (Lumsden e Keynes, 1989). Il processo di segmentazione che avviene durante lo sviluppo del rombencefalo è di fondamentale importanza sia per l’organizzazione finale di questo territorio, ad esempio l’uscita dei nervi cranici in distinti rombomeri, sia per la sua attività.
Figura 2: Organizzazione metamerica dei nervi cranici e dei loro gangli nel rombencefalo di embrione di topo.
Nel corso degli ultimi dieci anni, è stata fatta luce sui meccanismi cellulari che stanno alla base della formazione dell’organizzazione metamerica del rombencefalo. La suddivisione in rombomeri è il risultato di un processo di compartimentalizzazione cellulare che permette di conferire proprietà e caratteristiche distinte alle cellule che vi originano (Lawrence e Struhl, 1996). L’espressione di specifici geni all’interno di ciascun rombomero fa sì che questi acquisiscano identità distinte.
Per comprendere il ruolo svolto da un gene, sono stati fondamentali gli esperimenti compiuti su vari organismi modello come Xenopus, zebra fish e topo. In Xenopus e zebra fish questi esperimenti prevedono l’utilizzo di tecniche che portano o alla sovraespressione del gene (esperimenti di “gain of function”) o al suo silenziamento (esperimenti di “loss of function”). Nel topo, non essendo possibile attuare queste tecniche a causa dello sviluppo interno degli embrioni, si utilizzano
animali transgenici ottenuti mediante ricombinazione omologa nelle cellule staminali embrionali.
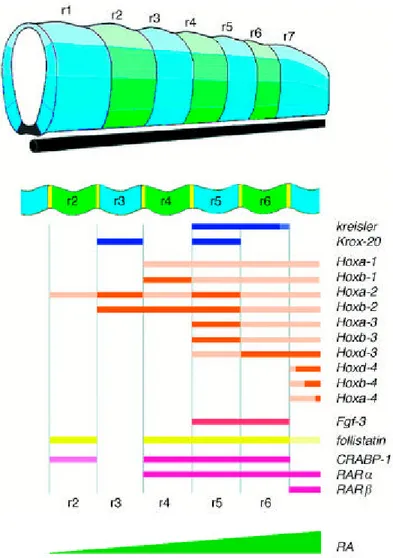
L’eliminazione di interi territori del rombencefalo e modificazioni della loro estensione riscontrate in topi mutanti per il fattore di trascrizione Krox20 ha permesso di giungere alla conclusione che questo gene è implicato nel processo di segmentazione. Il gene Krox20 mostra un’espressione segmentale nel rombencefalo (Wilkinson et al, 1989b). La sua espressione, ristretta r3 e r5 precede la formazione morfologica dei rombomeri. Topi mutanti per il gene Krox20 non presentano il r3 e r5 (Schneider-Maunoury et al., 1997; Schneider-Maunoury et al., 1993; Swiatek e Gridley, 1993). Altri due geni, codificanti per fattori di trascrizione come Krox20, sembrano controllare il processo di segmentazione. Il loro dominio d’espressione riflette la segmentazione del rombencefalo. Il primo di questi geni è kreisler (kr); esso è espresso nei territori presuntivi di r5 e r6 prima della comparsa della segmentazione morfologica (Cordes e Barsh, 1994); i topi mutanti per kreisler mostrano una parziale delezione di r5 e r6 e una perdita di segmentazione morfologica nella regione di r4 e r7 (Frohman et al., 1993; McKay et al., 1994; Moens et al., 1998). Il secondo gene è Gbx2 che è espresso ad uno stadio precoce dello sviluppo del SNC, a partire da 7.5 dpc, e ha un limite anteriore di espressione nella regione tra il mesencefalo e il rombencefalo, denominata istmo (Wassarman et al., 1997). L’inattivazione di questo gene porta ad una riduzione e ad una specificazione anormale della regione anteriore del rombencefalo (r1-r3) e dell’istmo.
Dopo che è avvenuta la segmentazione devono essere definiti i confini dei rombomeri. Le cellule dei diversi rombomeri presentano proprietà adesive di membrana distinte, date dall’espressione di distinti geni. Queste proprietà si ritrovano
lungo l’asse A-P e fanno sì che cellule provenienti da rombomeri pari non si mischino con quelle provenienti da rombomeri dispari (Guthrie e Lumsden, 1991; Guthrie et al, 1993). I segnali intercellulari hanno un ruolo fondamentale nella restrizione del destino cellulare in uno specifico segmento e componenti chiave per lo stabilirsi di queste interazioni sono i recettori con attività tirosina -chinasica (RTK). Tra le varie famiglie di RTK individuate, il gruppo dei recettori Eph e dei loro ligandi, ephrins (Xu e Wilkinson, 1997), presentano un dominio di espressione, nel rombencefalo, ristretto ai rombomeri. Per esempio EphA4 è fortemente espresso in r3 e r5 e debolmente in r2 (Gilardi-Hebenstreit et al., 1992; Nieto et al., 1992). L’iniezione, in embrioni di Xenopus e zebra fish, di mRNA codificante la proteina EphA4 nella forma troncata porta alla presenza di cellule appartenenti ai rombomeri 3 e 5 nei rombomeri 2, 4 e 6 (Xu et al.,1995).
Una volta che è avvenuta la segmentazione e i confini dei vari rombomeri sono stati definiti si ha espressione di un’altra famiglia di geni che specifica l’identità definitiva dei rombomeri, i geni Hox. Questi geni sono gli omologhi dei geni HOM-C di Drosophila. La famiglia dei geni Hox comprende un gruppo ben caratterizzato di geni che mostrano un’elevata omologia di sequenza a livello di un dominio proteico, detto homeobox, il quale codifica per un dominio di 60 aminoacidi capace di legare il DNA in maniera sequenza-specifica. In Drosophila i geni Hox si trovano sullo stesso cromosoma e formano i complessi Antennapaedia e Bithorax. Nei vertebrati tetrapodi questi geni sono raggruppati in quattro complessi (HoxA, HoxB, HoxC e HoxD) localizzati su quattro cromosomi. I quattro complessi genici dei vertebrati derivano dalla duplicazione di un unico gene ancestrale, di conseguenza i geni corrispondenti dei quattro complessi sono molto simili tra loro. I geni originati per duplicazione e
divergenza all’interno di una specie sono detti paraloghi (si veda per un compendio Krumlauf, 1992). Lo studio della distribuzione dei trascritti ha mostrato che l’espressione dei geni Hox è finemente regolata nel tempo e nello spazio e che il dominio di espressione dei geni Hox dipende dalla loro posizione nel complesso. Esiste infatti una stretta correlazione tra l’ordine dei geni lungo il cromosoma e la loro espressione lungo l’asse antero-posteriore dell’embrione: i geni più vicini al 5’ mostrano un’espressione più caudale rispetto ai geni Hox localizzati più vicino al 3’, che invece sono espressi in domini progressivamente più rostrali. Nel topo i geni Hox dei gruppi paraloghi da 1 a 4 hanno domini di espressione che si estendono nel rombencefalo ed il loro dominio anteriore di espressione coincide con quello tra due rombomeri adiacenti, con un periodicità di due segmenti (Gauntetal 1989; Gaunt at al., 1988; Hunt at al., 1991; Murphy e Hill, 1991; Prince e Lumsden, 1994; Wilkinson at al., 1989). Ad esempio il confine anteriore di espressione di Hoxb2 corrisponde al limite tra r2 ed r3 e quello di Hoxb3 corrisponde al limite tra r4 e r5. L’importanza dei geni Hox nel determinare l’identità dei rombomeri è dimostrata, ad esempio, dall’analisi di mutanti per il gene Hoxa2. Gli embrioni mutanti presentano un’assenza di elementi scheletrici del secondo arco branchiale, come la staffa dell’orecchio interno, mentre sviluppano strutture che derivano dal primo arco branchiale, come la cartilagine di Meckel, un elemento precursore della mandibola (Rijli et al., 1993). All’interno dei rombomeri, oltre ai geni Hox, sono espressi altri geni che determinano la specificazione delle cellule. Una famiglia di questi geni è Wnt. Molti dei membri di questa classe sono espressi durante le fasi precoci della formazione del tubo neurale (si veda per un compendio McMahon, 1992; Nusse e Varmus, 1992; Parr et al., 1993). Diversi geni Wnt sono espressi in domini definiti lungo l’asse A-P del sistema
nervoso, suggerendo un loro ruolo nello stabilire territori specifici. Questi geni codificano per glicoproteine secrete, che agiscono come molecole di segnale intercellulare durante lo sviluppo (Van den Heuvel et al., 1989; Gonzalez et al., 1991; Jue et al., 1992). Grazie all’utilizzo della ricombinazione omologa nelle cellule embrionali staminali è stato possibile produrre embrioni mutanti per Wnt1 (McMahon e Bradley, 1990; Thomas e Capecchi, 1990). Questi esperimenti hanno mostrato che l’assenza di Wnt1 ha come risultato la delezione del mesencefalo e la delezione di una sostanziale parte del rombencefalo rostrale (McMahon et al., 1992). Quindi è evidente che Wnt1 è necessario, in modo diretto o indiretto, al corretto sviluppo della regione mesencefalica e rombencefalica.
Un'altra classe di geni che è implicata nella specificazione di un fenotipo neurale delle cellule del sistema nervoso è data dai fattori di trascrizione “basic-helix-loop-helix” (bHLH) (Bertrand et al., 2002). I fattori di trascrizione neurali bHLH dei vertebrati sono classificati in sottogruppi in base alla loro espressione temporale (Lee, 1997) e alla loro omologia con i geni proneurali di Drosophila (Bertrand et al., 2002). Il fattore Math1 è omologo del gene atonal di Drosophila (Akazawa et al., 1995) e appartiene al sottogruppo dei fattori bHLH che si esprimono precocemente nello sviluppo. Math1 è espresso transientemente in vari precursori neuronali del sistema nervoso, incluso la parte dorsale del rombencefalo e del tubo neurale, (Akazawa et al., 1995; Ben-Arie et al., 2000; Ben-Arie et al., 1996; Helms eJohnson, 1998). Analisi su topi knockout per questo gene hanno dimostrato il suo ruolo nella formazione di specifiche popolazioni di neuroni (Ben-Arie et al., 1997; Bermingham et al., 1999; Bermingham et al., 2001; Fode et al., 2000; Gowan et al., 2001; Guillemot et al., 1993; Parras et al., 2002). Inoltre analisi fatte utilizzando le tecniche di
“gain-of-function” in vari sistemi animali modello hanno fornito evidenze che questo gene è capace di indurre differenziazione neurale (Cai et al., 2000; Cepko, 1999; Farah et al., 2000; Kim et al., 1997). A livello del rombencefalo si hanno anche geni regolati da segnali secreti da altre strutture dell’organismo in via di sviluppo, come ad esempio la regolazione da parte della molecola Sonic hedgehog, secreta dalla notocorda, su alcuni membri della famiglia Pax. All’interno di questa classe il membro più caratterizzato è Pax6. (si veda per un compendio Gruss e Walther, 1992; Hill e Hanson, 1992; van Heyningen, 1998; Gehring e Ikeo, 1999). Durante lo sviluppo questa molecola è espressa nella parte dorsale del telencefalo (Walther e Gruss, 1991; Stoykova etal., 1997; Pratt et al., 2000; Osumi, 2001). Nel rombencefalo e nel midollo spinale Pax6 è espresso nella regione ventrale, e gioca un ruolo chiave nella formazione di neuroni ventrali come ad esempio i motoneuroni branchiali e i motoneuroni somatici. Il ruolo di questo gene nello sviluppo di strutture rombencefaliche è dimostrato dall’analisi condotta su topi mutanti omozigoti, in cui si ha un’assenza dei motoneuroni somatici e un incremento dei motoneuroni branchiali (Osumi et al., 1997; Ericsonet al., 1997; Sun et al., 1998).
L’identità posizionale delle cellule viene quindi ad essere il risultato dell’integrazione, a livello di ogni singolo rombomero, di tutte le informazioni che contribuiscono allo sviluppo del rombencefalo, dovuta alla presenza di gradienti morfogenetici precoci, geni di segmentazione, geni che codificano per proteine di adesione, geni Hox., e geni di specificazione (fig. 3).
Figura 3: Rappresentazione schematica dell’ espressione di vari geni importanti per lo sviluppo del rombencefalo di topo.
1.3 Il “rhombic lip”
Il rombencefalo dà origine al cervelletto, al ponte ed al midollo allungato. A livello di queste strutture sono presenti la componente motoria ed i nuclei sensoriali dei nervi cranici V-XII, i neuroni del sistema reticolare, gli interneuroni deputati alla regolazione di numerose attività vitali quali ad esempio la frequenza respiratoria ed il battito cardiaco. Dalla regione più dorsale del rombencefalo, al confine tra il tessuto neurale
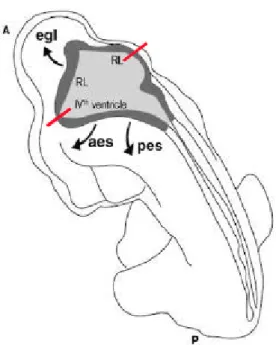
e l’ectoderma adiacente, hanno origine, a stadi precoci dello sviluppo embrionale, le cellule della cresta neurale cranica. Da questa popolazione di cellule, che migra negli archi faringei, si sviluppa il sistema nervoso periferico, la maggior parte delle ossa del cranio della testa e del collo, e le cellule del connettivo associato alla muscolatura facciale (Lumsden et al., 1991; Kontges e Lumsden, 1996). La regione del rombencefalo all’interfaccia tra il tubo neurale e il tetto del quarto ventricolo nota come “rhombic lip”, costituisce un neuroepitelio germinativo specializzato in grado di dare origine a numerose ed importanti strutture del SNC adulto (fig 4).
Figura 4: Rappresentazione schematica del “rhombic lip” e delle vie di migrazione dei precursori neurali. egl: via di migrazione delle strato germinale esterno; aes: migrazione extramurale anteriore; pes: migrazione extramurale posteriore
Studi morfologici relativi al destino dei precursori del “rhombic lip” furono condotti oltre un secolo fa da His (1891) e da Essick (1912). Successivamente, studi di ablazione (Harkmark, 1954), di “birhtdating” (Taber Pierce, 1966; Ellenberg et al.,
1969; Altman e Bayer, 1987) e di marcatura con perossidasi (Bourrat e Sotelo, 1990a, 1990b) hanno fornito evidenze, seppur indirette, della presenza nel “rhombic lip” di precursori neuronali in grado di raggiungere, attraverso precise vie di migrazione, la porzione ventrale del rombencefalo. Negli anni ’50 esperimenti di “fate mapping” condotti su pollo hanno permesso di stabilire che dal neuroepitelio germinativo del “rhombic lip” hanno origine i precursori dello strato germinale esterno (EGL) del cervelletto da cui, dopo la nascita, derivano le cellule granulari della corteccia cerebellare. Inoltre, il “rhombic lip” è anche la sorgente dei precursori dei nuclei che compongono il sistema precerebellare.
1.3.1 Il sistema cerebellare
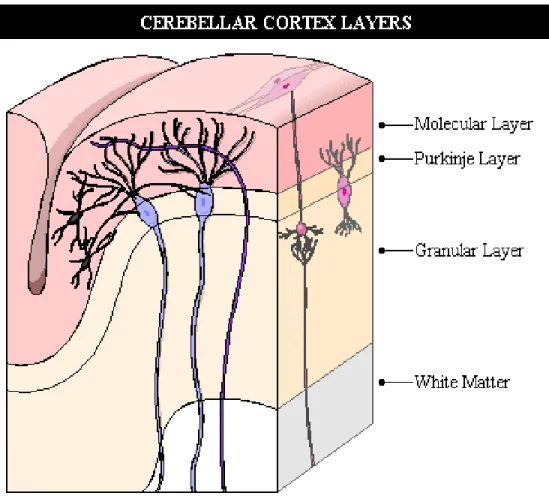
Il cervelletto è una delle regioni del sistema nervoso i cui “patterns” d’organizzazione sono più conosciuti. La corteccia cerebellare con la sua apparente semplicità e disposizione geometrica ha attratto l’interesse di molti ricercatori nel tentativo di comprendere i meccanismi implicati nello sviluppo del sistema nervoso. La corteccia cerebellare è composta da otto diverse popolazioni di neuroni. Nel 1911 Cajal aveva descritto sei di queste popolazioni: le cellule del Purkinje , le cellule del Golgi, le cellule del Lugaro, le cellule granulari, le cellule a canestro, e le cellule stellate; recentemente sono state scoperte le cellule unipolari a cespuglio e le cellule a candelabro (Laine e Axelrad, 1994; Mugnaini e Floris, 1994). Queste cellule si trovano nei tre strati che compongono la corteccia. Partendo dalla regione più superficiale troviamo lo strato molecolare, una zona in cui si ha una bassa densità
cellulare e un’elevata presenza di sinapsi. Lo strato intermedio, chiamato anche strato delle cellule del Purkinje, è un monostrato di corpi cellulari delle cellule del Purkinje. Lo strato più profondo è lo strato granulare, in cui è presente la più alta densità cellulare di ogni altra struttura del SNC (fig 5).
Figura 5: Schema degli strati che compongono la corteccia.
Il cervelletto deriva sia dalla vescicola mesencefalica sia da quella rombencefalica. Usando le tecniche di trapianto è stato visto che il primordio del cervelletto deriva dalla parte più caudale del mesencefalo e dalla regione più anteriore del
rombencefalo che corrisponde ad r1 (Martinez e Alvarado-Mallart, 1989; Marin e Puelles, 1995). I precursori delle cellule granulari della corteccia cerebellare prendono origine dal) “rhombic lip” di r1 e, nel topo, iniziano a proliferare da 10 dpc. Successivamente, attraverso un processo di migrazione tangenziale, queste vanno a ricoprire la zona superficiale del primordio del cervelletto formando l’EGL dove ha luogo la ripresa di un’intensa attività mitotica. Dopo la nascita, cessa la proliferazione cellulare e i neuroni iniziano un processo differenziativo e, attraverso una migrazione di tipo radiale, raggiungono la loro destinazione finale nello strato granulare. Dopo circa due settimane dalla nascita si ha la scomparsa dell’EGL e la formazione delle cellule granulari.
Il cervelletto svolge un ruolo primario sul controllo dell’equilibrio e della coordinazione del movimento, del movimento degli occhi, dell’apprendimento motorio, e, verosimilmente, sembra anche essere implicato in funzioni cognitive (Mauk et al., 2000; Gao et al., 1996; Schmahmann, 1997). Informazioni di tipo visivo, acustico e somatosensoriale giungono al cervelletto mediante le fibre rampicanti e muscoidi che sinaptano rispettivamente con le cellule del Purkinje e con le cellule granulari. (Altman e Bayer, 1997; Brodal e Bjaalie, 1992; Sotelo e Chedotal, 1997).
1.3.2 Il sistema precerebellare
Il sistema precerebellare, al contrario del sistema cerebellare, solo negli ultimi anni è stato oggetto di un intenso studio, e ciò grazie all’identificazione di marcatori molecolari specifici i quali hanno permesso di conoscere alcuni dei meccanismi che ne regolano lo sviluppo. Infatti, i primi studi risalgono ai lavori di Altman e Bayer, effettuati alla fine degli anni settanta, sullo sviluppo prenatale del sistema precerebellare. Tuttavia, lo studio dei meccanismi implicati nella formazione dei nuclei che compongono il sistema precerebellare rappresenta un aspetto affascinante che comprende meccanismi di specificazione neuronale, migrazione cellulare e proiezione assonale. Inoltre, la conoscenza di tali meccanismi risulta di fondamentale importanza tenendo in conto che questo sistema media la maggior parte delle informazioni che arrivano al cervelletto ed ha quindi un ruolo cruciale nella coordinazione dell’attività motoria. Infatti, l’analisi di alcune forme di atassia ha rivelato anomalie a livello dei nuclei che compongono il sistema cerebellare. Un esempio è OPCA (“olivopontocerebellar atrophy” IV), una forma di atassia ereditaria in cui sono presenti malformazioni a carico del nucleo olivare e del nucleo pontino.
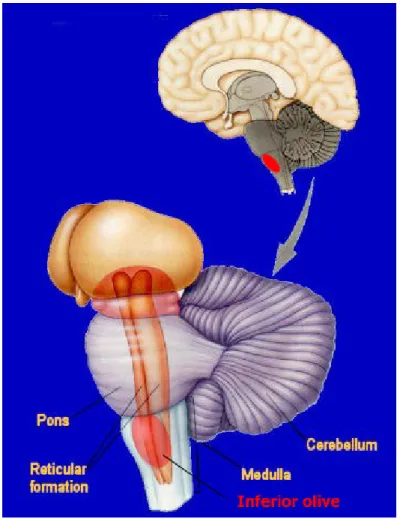
Il sistema precerebellare, in senso rostro-caudale, è composto da cinque nuclei distinti: il nucleo pontino (PN), il nucleo reticolo tegmentale (RTN), il nucleo reticolare laterale (LRN), il nucleo cuneato esterno (ECN) e il nucleo dell’oliva inferiore (ION) (si veda per un compendio Altman e Bayer, 1987) (fig 6).
Figura 6: Rappresentazione schematica del cervello adulto posteriore. In rosso è evidenziata la posizione del nucleo dell’oliva inferiore a livello del midollo allungato.
I nuclei precerebellari che, come già detto sopra, si originano dal “rhombic lip” sono localizzati nella parte ventrale del rombencefalo dove giungono in seguito ad un preciso processo di migrazione durante l’embriogenesi. Esperimenti di “fate mapping” nel pollo hanno evidenziato un’origine multi-segmentale di due dei principali nuclei precerebellari, il nucleo pontino, ed il nucleo dell’oliva inferiore. Il “rhombic lip” compreso tra r2 ed r8 contribuisce alla formazione del nucleo pontino (Marin e Puelles, 1995), mentre il nucleo olivare deriva dal rombomero 8 e dal midollo spinale anteriore (Cambronero e Puelles, 2000).Negli ultimi anni sono stati identificati vari geni, codificanti per fattori di trascrizione e molecole di segnale
diffusibili, che mostrano in fasi precoci dello sviluppo un’espressione localizzata a livello del neuroepitelio dorsale rombencefalico. Uno di questi geni è Wnt1. Recenti studi hanno proposto che i precursori del “rhombic lip” che esprimono questo gene danno origine a quattro dei cinque nuclei del sistema precerebellare mentre ci sono contraddizioni sull’origine dei precursori del nucleo dell’oliva inferiore (Rodriguez e Dymecki, 2000).
a) IL NUCLEO PONTINO
Il nucleo pontino si trova nella parte rostro-ventrale del ponte, ed i precursori che andranno a formare questo nucleo iniziano la loro migrazione a 12 dpc, con un picco tra 14 e 15 dpc raggiungendo la destinazione finale nella porzione ipsilaterale (Bourrat e Sotelo, 1988,1990) del ponte a partire da 16 dpc. Questi neuroni compiono una migrazione superficiale (sub-piale), in senso dorso-ventrale e anteriore, definita extramurale anteriore. Tra 18.5 dpc e P0, i neuroni del nucleo pontino iniziano ad emettere i loro assoni alla parte controlaterale del cervelletto formando le fibre muscoidi. Queste fibre vanno a sinaptare con le fibre parallele delle cellule granulari a livello dello strato granulare. Le fibre parallele sinaptano poi con le cellule del Purkinje.
Oltre al nucleo pontino le fibre rampicanti prendono origine anche dai nuclei reticolo tegmentale, reticulare laterale e cuneato esterno.
b) IL NUCLEO DELL’OLIVA INFERIORE
Il nucleo dell’oliva inferiore si trova nella parte ventro-caudale del midollo allungato. La migrazione dei precursori che daranno origine all’oliva inferiore ha inizio a 12 dpc (Bourrat e Sotelo, 1988; Altman e Bayer, 1987). La migrazione non è superficiale come per il nucleo pontino, ma è interna, e segue una direzione dorso-ventrale e posteriore. Questa via di migrazione è esclusiva del nucleo olivare ed è definita intramurale. I neuroni dell’oliva inferiore, una volta raggiunta la locazione finale, si fermano senza attraversare la linea mediana, rimanendo ipsilaterali (Bourrat e Sotelo, 1988,1990). A 13 dpc, mentre i precursori continuano la loro migrazione, inizia la proiezione degli assoni. A 14 dpc quasi tutti i precursori hanno ragginto la loro destinazione finale. A 17 dpc i neuroni olivari hanno terminato la loro migrazione, e gli assoni, che formano le fibre rampicanti, iniziano ad attraversare la linea
mediana per sinaptare con l’albero dendritico delle cellule del Purkinje nello strato molecolare della parte controlaterale del cervelletto (Bloch-Gallego et al., 1999).
1.4
Tecniche di “FATE MAPPING”nei mammiferi
Nel topo le informazioni relative allo sviluppo e all’origine dei precursori neurali derivano, per estrapolazione, dagli esperimenti di trapianto effettuati nel pollo.
Da qualche anno, però, grazie alle nuove tecniche di genetica molecolare è possibile anche nel topo effettuare studi sull’origine dei vari precursori, “fate mapping”, che portano alla formazione di distinte strutture nel sistema nervoso.
neurali, e seguire poi i processi differenziativi successivi. Queste tecniche sono rese possibili dall’impiego della ricombinasi Cre in grado di promuovere eventi di ricombinazione somatica in linee transgeniche che esprimano un gene “reporter” inducibile. La ricombinasi Cre e le linee transgeniche verrano analizzate in dettaglio successivamente.
1.4.1 Ricombinazione omologa: knockout, knockin
All’inizio degli anni ’80, sono state isolate per la prima volta cellule staminali embrionali, le cellule ES. Queste sono cellule totipotenti in grado di dare origine a tutti i tipi cellulari presenti in un organismo, compresa la linea germinale. Le cellule ES possono essere mantenute in coltura e la totipontenza preservata grazie alla presenza nel mezzo di coltura del fatttore LIF (Leukemia Inhibitor Factor). In queste condizioni le cellule ES possono essere manipolate geneticamente e, grazie ai processi di ricombinazione omologa, un frammento di DNA esogeno può essere integrato in loci precisi del genoma. Così facendo, è possibile introdurre mutazioni in geni specifici. L’uso di appropriati vettori a DNA ha permesso di sfruttare questo evento per introdurre un frammento di interesse nel genoma e di selezionare le cellule che hanno avuto ricombinazione omologa (Capecchi, 1989).
Il vettore per la ricombinazione omologa è costituto da un plasmide a Dna che contiene sequenze omologhe al gene bersaglio di interesse interrotte da un marcatore che permette una selezione, ad esempio il gene neo che conferisce resistenza alla neomicina. Il vettore viene inserito mediante elettorporazione nelle cellule ES (Thomas e Capecchi, 1987). Il marcatore presente nelle sequenze ha un duplice ruolo: da un lato, la presenza del gene neo permette di selezionare i cloni
delle cellule ES che hanno integrato il vettore, dall’altro, interrompe la sequenza codificante inattivando il gene. Successivamente, possono essere selezionati ed isolati i cloni di cellule ES che hanno integrato il frammento nel genoma tramite ricombinazione omologa (Thomas e Capecchi, 1987). L’integrazione del frammento portato dal vettore nel genoma può avvenire anche con una ricombinazione non-omologa in regioni diverse del genoma. In questo caso queste cellule possono essere controselezionate inserendo nel vettore il gene codificante per la Timidina Chinasi (TK) del virus Herpes simplex (Mansour et al., 1988). Le cellule ES eterozigoti per il gene che è stato bersagliato possono a questo punto essere reintrodotte in una blastocisti per ottenere topi chimerici. Da questi attraverso successivi incroci è possibile ottenere animali knockout Una procedura sempre più in uso negli ultimi anni nota come knockin genico prevede l’introduzione di un gene “reporter” come LacZ (Le Mouellic et al., 1992; Mansour et al., 1990) o GFP (Green Fluorescent Protein) (Chalfie et al., 1994) in “frame” con la sequenza codificante del gene d’interesse, . In questo modo possono essere ottenuti topi mutanti per il gene d’interesse in cui il gene reporter è espresso in maniera da riprodurre l’espressione completa del gene endogeno mutato. Quindi, le cellule che normalmente esprimono il gene mutato possono essere identificate per la presenza del reporter ed è possibile seguire il loro destino differenziativo sia nel contesto del topo eterozigote sia nel mutante.
1.4.2 Topi transgenici
Vettori a DNA costituti da promotori tessuto specifici per il controllo trascrizionale di geni reporter o della ricombinasi Cre possono essere iniettati nel macronucleo di zigoti di topo. In condizioni ottimali il vettore può essere integrato nel genoma dell’embrione che può essere reimpiantato in utero. Questo offre la possibilità di generare linee transgeniche di topo in grado di esprimere il gene reporter utilizzato, o la ricombinasi Cre, nei tessuti in cui il promotore utilizzato sia attivo.
1.4.3 Transgenesi binaria
La generazione di topi mutanti per specifici geni ha permesso lo studio di molti processi biologici, dall’embriogenesi al comportamento degli animali. Purtroppo, non tutti questi processi possono essere studiati tramite l’inattivazione di geni o l’uso di inattivazione genica (knockout) convenzionale descritto sopra; ad esempio, molti geni espressi in tessuti adulti ricoprono anche un ruolo fondamentale durante lo sviluppo embrionale e la loro mutazione porta ad un fenotipo letale che rende impossibile la comprensione del loro ruolo nelle fasi post natali. Questo problema è stato in parte risolto grazie all’avvento di un nuovo strumento di genetica molecolare: il sistema della transgenesi binaria. In questo sistema l’espressione di un gene è controllata dall’interazione di due componenti: un transgene “effettore” e un transgene “target”. La transgenesi binaria può essere di due tipi. In un caso l’”effettore” attiva la trascrizione del transgene “target”, mentre nell’altro caso l’”effettore” è una ricombinasi sito-specifica che agisce sul gene “target” attivandolo o silenziandolo.
Durante gli ultimi anni il numero dei sistemi di transgenesi binaria sono andati sempre crescendo ma, uno dei sistemi più efficaci e utilizzati è il sistema Cre/loxP.
1.4.4 Ricombinasi Cre e linee transgeniche
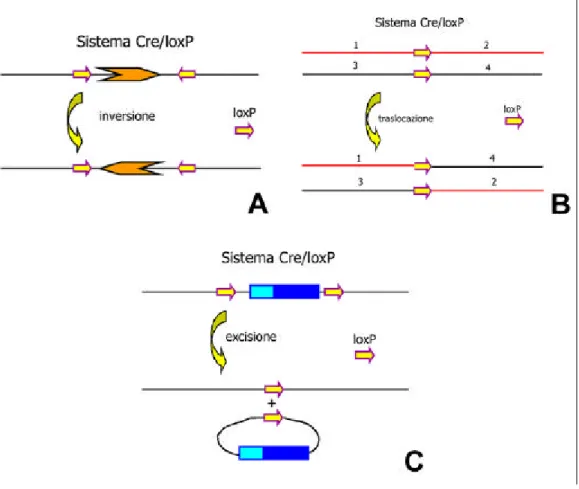
La ricombinasi Cre è un enzima che fa parte della famiglia delle integrasi. Questo enzima, presente in natura nel batteriofago P1 (Sauer e Henderson, 1989), ha un ruolo nel ciclo lisogenico, in quanto serve per l’integrazione del genoma fagico in quello batterico. La ricombinasi Cre è in grado di riconoscere sequenze sito specifiche chiamate siti loxP, che fiancheggiano una sequenza di DNA, che si dice “floxata”. Questi siti sono costituiti da due sequenze palindromiche interrotte da una sequenza “spacer”, per un totale di 34 nucleotidi. In base alla direzione della sequenza “spacer” l’enzima è in grado di catalizzare tre tipi di reazione:
1) Inversione: i siti loxP si trovano sullo stesso filamento di DNA ed hanno direzioni opposte. Il prodotto della reazione è un’inversione del frammento di DNA “floxato”.
2) Traslocazione: i siti loxP si trovano su due filamenti diversi. Il prodotto della reazione è una traslocazione cromosomica.
3) Excisione: i siti loxP si trovano sullo stesso filamento ed hanno la stessa direzione. Il prodotto della reazione è la formazione di un “loop” tra i due siti, e l’excisione di tutto ciò che si trova all’interno. Il frammento exciso in vivo viene rapidamente degradato rendendo il processo di excisione è irreversibile.
Figura 7: Rappresentazione schematica dei meccanismi d’azione della ricombinasi Cre. (A) inversione; (B) traslocazione; (C) excisione.
Queste particolarità fanno del sistema Cre/loxP un mezzo particolarmente utile per realizzare alterazioni fini nel genoma sia negli animali che nei vegetali. Al contrario di altri sistemi non sono necessari né cofattori né siti accessori, come sequenze regolatrici, e la sua direzionalità permette in generale un’alterazione stabile nel genoma. Il sistema Cre/loxP può essere usato per analisi sulle linee cellulari. Un metodo usato per questo tipo di indagini è la produzione di una linea in cui un gene “reporter”, attivabile dall’azione della ricombinasi, si trova nel locus di un gene espresso ubiquitariamente. Due esempi di queste linee transgeniche sono R26R e
a) R26R
Il gene ROSA26 è un gene ubiquitario. Nella linea transgenica “reporter” la regione codificante del gene è stata sostituita con la regione codificante del gene della ? -galattosidasi (? -gal). Nel costrutto è presente una cassetta di STOP floxata che reprime la trascrizione del gene ? -gal. Incrociando un topo transgenico R26 con un topo transgenico Cre si ha espressione del gene ? -gal in quanto la ricombinasi excide la cassetta di STOP e dereprime la trascrizione. L’avvenuta trascrizione del gene ? -gal è rilevabile mediante l’uso di un substrato cromogenico analogo al -galattosio (X-GAL) (Soriano, 1999).
Figura 8: Schema del costrutto utilizzato per ottenere il trangene R26R
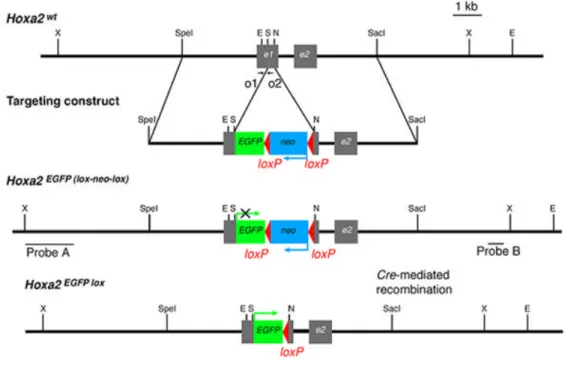
b) Hoxa2 EGFP(loxP -neo-loxP)
In questa linea la sequenza codificante del gene Hoxa2, un gene omeotico espresso nel r2 implicato nella segmentazione rombencefalica, è stata sostituita con la regione codificante del gene EGFP (Enhanced Green Fluorescent protein). La presenza, a valle della regione del gene EGFP, del gene neo, usato per selezionare le cellule ES modificate, interferisce con la normale espressione del gene “reporter” reprimendone la trascrizione. La presenza di siti loxP ai lati del gene neo ne permette l’excisione mediata dalla ricombinasi Cre. Come conseguenza si ha la derepressone
della trascrizione della EGFP all’interno del dominio di espressione di Hoxa2 che può essere visualizzata mediante l’uso di un microscopio a fluorescenza (Pasqualetti et al., 2002).
Figura 9: Schema della strategia utilizzata per la preparazione del costrutto Hoxa2 EGFP(loxP-neo-loxP)
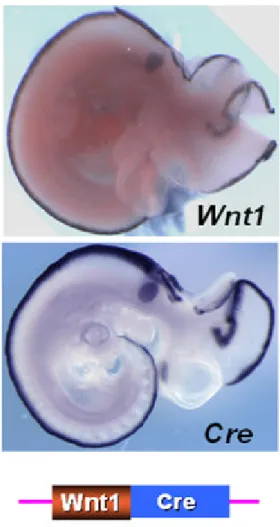
c) Wnt1Cre
Incrociando una linea cui la Cre è controllata da un promotore tessuto e tempo specifico con una linea che porta un gene “reporter” inducibile è possibile seguire il destino delle cellule che esprimono (o hanno espresso) la ricombinasi. Una di queste linee è rappresentata dalla linea Wnt1/Cre, in cui l’espressione della ricombinasi è controllata dal promotore del gene Wnt1.
l’espressione nel mesencefalo viene ridotta alla linea mediana dorsale e compare nella regione più dorsale del rombencefalo, che comprende il “rhombic lip” (Wilkinson et al., 1987; McMahon et al., 1992; Parr et al., 1993).
La linea Wnt1/Cre in accoppiamento alle linee reporter inducibili menzionate può essere impiegata per studiare il destino dei precursori neuronali del rhombic lip che esprimono il gene Wnt1.
Figura 10: Comparazione dell’espressione genica di Wnt1 (figura in alto) con l’espressione del costrutto transgenico Wnt1/Cre (figura in basso) in embrioni di topo a stadio 10 dpc. Lo schema in basso rappresenta il costrutto utilizzato per la transgenesi.
1.5 Scopo della tesi
La comprensione delle interazioni molecolari e cellulari, che stanno alla base dello sviluppo del cervello, necessita come base di partenza una approfondita
conoscenza dell’origine dei distinti precursori neuronali e della loro localizzazione nel cervello adulto. La conoscenza dell’origine di questi precursori è fondamentale per comprendere le funzioni svolte dalle diverse strutture che costituiscono il sistema nervoso centrale. L’origine embrionale dei diversi nuclei che compongono il sistema precerebellare rappresenta una questione importante da approfondire per la
neurobiologia dello sviluppo.
Durante il mio internato di tesi ho condotto esperimenti tesi allo studio della sequenza temporale attraverso cui si giunge alla formazione del nucleo dell’oliva inferiore ed alla caratterizzazione molecolare dei precursori neurali da cui ha origine questo importante nucleo del sistema precerebellare. A tale proposito, ho effettuato un “time-course” dell’espressione di Brn3.2, un marcatore molecolare specifico per i neuroni dell’oliva inferiore, mediante esperimenti di ibridazione in situ su sezione di embrioni di topo a stadi compresi tra 12.5 dpc e 17.0 dpc. Successivamente, ho impiegato embrioni di topo ottenuti dall’incrocio delle tre linee transgeniche R26R, Hoxa2 EGFP(loxP-neo-loxP), Wnt1/Cre su cui ho effettuato esperimenti di immunistochimica ed ibridazione in situ su sezioni seriali allo scopo di marcare i precursori neurali che danno origine al nucleo olivare e, in parallelo, la sottopopolazione dei neuroni positivi per l’espressione di Wnt1 originati dal “rhombic lip”.