Studio capacità depurativa
La membrana semipermeabile di dialisi rappresenta senza dubbio il nucleo centrale del sistema dell’emodialisi. Infatti tutte le tecniche dialitiche consistono nel far giungere il sangue dal paziente a contatto con una membrana semipermeabile, che separa il sangue dal dializzato, è a livello della membrana che avviene la dialisi.
Le membrane da emodialisi esercitano un’azione di barriera semipermeabile per il trasporto dell’acqua e soluti dal compartimento del sangue al compartimento del dializzato.
In questa tesi l’attenzione è stata incentrata prevalentemente sull’analisi delle capacità depurative delle membrane di dialisi.
I pazienti, in trattamento emodialitico cronico (MHD), vengono comunemente trattati con bicarbonato dialisi standard (SHD) impiegando membrane a basso flusso (LF) che depurano il sangue dalle tossine di piccolo peso molecolare (urea, elettroliti, acido urico, etc.) ma non rimuovono molecole potenzialmente tossiche nel range delle proteine di medio peso molecolare (LMWP).
Lo studio prevede la valutazione dell’efficienza della bicarbonato dialisi con membrane ad alto flusso (HF) nella rimozione di molecole a piccolo-medio peso molecolare. L’efficienza è stata testata con due metodiche. La metodica biochimica e quella proteomica. In particolare il mio lavoro di dottorato prende in considerazione l’aspetto proteomico.
Per quanto riguarda l’approccio biochimico le analisi chimico-cliniche svolte hanno messo in evidenza la capacità depurativa delle membrane di molecole a basso peso molecolare. Si può
per le proteine a peso molecolare medio, infatti la loro rimozione è più alta con le membrane ad alto flusso (HF) rispetto a quella a basso flusso (LF) (fig. 3-4).
Fig.1: L’istogramma mostra la capacità dialitica per molecole di basso peso molecolare . L’efficienza delle differenti membrane è calcolata come reduction ratio (RR). La rimozione di piccole molecole è molto alta con tutte le membrane sia LF(F8) che HF(Poly 210H, FX80, N190 FH).
% 0 10 20 30 40 50 60 70 80 90 F 8 Poly 210 H FX 80 N 190 FH
Urea RR Creat RR Phosphate RR
NS NS NS NS
Fig.2: L’istogramma mostra la capacità dialitica per molecole di basso peso molecolare di tre membrane HF . L’efficienza delle differenti membrane è calcolata come reduction ratio (RR). La rimozione di piccole molecole è molto alta con tutte e tre le membrane.
Rimozione di molecole a basso peso
0 1 2 3 4 5 6 7 8
9 Urea RR Creatinine RR Phosphate RR
Rimozione molecole medio peso molecolare
-30
-20
-10
0
10
20
30
40
50
60
70
80
90
F 8
Poly 210 H
FX 80
N 190 FH
%
beta 2 RR cistatina RR Estraz Mb
*** # ### # *** ## *** ## *** *** * *** # *** ###
Fig.3: il grafico mostra come la rimozione delle molecole di medio peso molecolare sia più alta con le membrane HF
rispetto alle LF. La più alta efficienza nella rimozione è stata osservata con N190FH: N190FH mostra un alta RR per la mioglobina:
* p<0.05, ** p<0.01,*** p<0.001 significatività statistica VS F8 ° p<0.05, °° p<0.01, °°° p<0.001 significatività statistica VS Poly # p<0.05, ## p<0.01,### p<0.001 significatività statistica VS FX80 $ p<0.05, $$ p<0.01, §§§ p<0.001 significatività statistica VS N190FH.
Fig.4: : L’istogramma mostra la capacità dialitica per molecole di medio peso molecolare di tre membrane HF . L’efficienza delle differenti membrane è calcolata come reduction ratio (RR). La rimozione di piccole molecole è molto alta con tutte e tre le membrane.
0
10
20
30
40
50
60
70
80
90
100
B2M RR Cystatin C RR Myoglobin RR Polyflux 210H N190 FH FX 1000Valutazione proteomica
Grazie all’approccio proteomico che prevede l’utilizzo delle tecniche elettroforetiche SDS-PAGE e 2DE, abbiamo ottenuto dei dati sulla capacità depurativa di molecole a medio peso molecolare, in accordo con i risultati biochimici. Le membrane che abbiamo studiato sono le medesime quattro ad alto flusso, Triacetato di Cellulosa (Nipro Superflux 190FH), Helixone (Fresenius FX 80), Helixone (Fresenius FX1000), Poliamide (Gambro Polyflux 210H) ed quella a basso flusso Polisulfone (Fresenius F8).
Il meccanismo di depurazione delle membrane di dialisi si basa su tre processi che sono:
la diffusione, per gradiente di concentrazione. Efficiente per soluti non legati a proteine e proteine di medio-basso peso molecolare.
La convezione ed ultrafiltrazione, per trasferimento attraverso la membrana di molecole a medio-alto peso molecolare, che possono passare attraverso i pori della membrana.
L’adsorbimento per ritenzione all’interno della membrana. Tale meccanismo permette la depurazione di proteine a basso medio ma soprattutto a medio-alto peso molecolare.
Con le tecniche proteomiche abbiamo analizzato questi tre meccanismi prendendo rispettivamente in esame:
Come primo passaggio sono state eseguite elettroforesi monodimensionali con lo scopo di avere un primo riscontro sulle capacità depurative delle singole membrane. In fig.5 sono riportate SDS-PAGE di sieri di inizio e fine dialisi di cinque membrane ( F8, FX80, FX1000, N190, Poly 210 H). La prova è stata effettuata per potere apprezzare, non solo differenze di capacità depurativa, che non sono particolarmente evidenti in questo campione, ma anche un’ eventuale variabilità intra-individuale e quindi poter decidere i passaggi successivi dello studio. La quantità di campione caricata nei pozzetti del gel è eccessiva per una buona risoluzione delle proteine ad alto peso molecolare, infatti, come si può notare dall’immagine (fig.5) abbiamo una saturazione a pesi molecolari alti da 68 kDa in su, questo è dovuto alla preponderante presenza di albumina, che costituisce quasi il 60% di tutte le proteine plasmatiche e che, per questo, nella quantificazione proteica causa un sovrastima dell’effettiva concentrazione delle altre proteine meno rappresentate, è quindi necessario caricare una quantità tale di campione che consenta di evidenziare le numerose proteine che nel campione presentano una concentrazione minore.
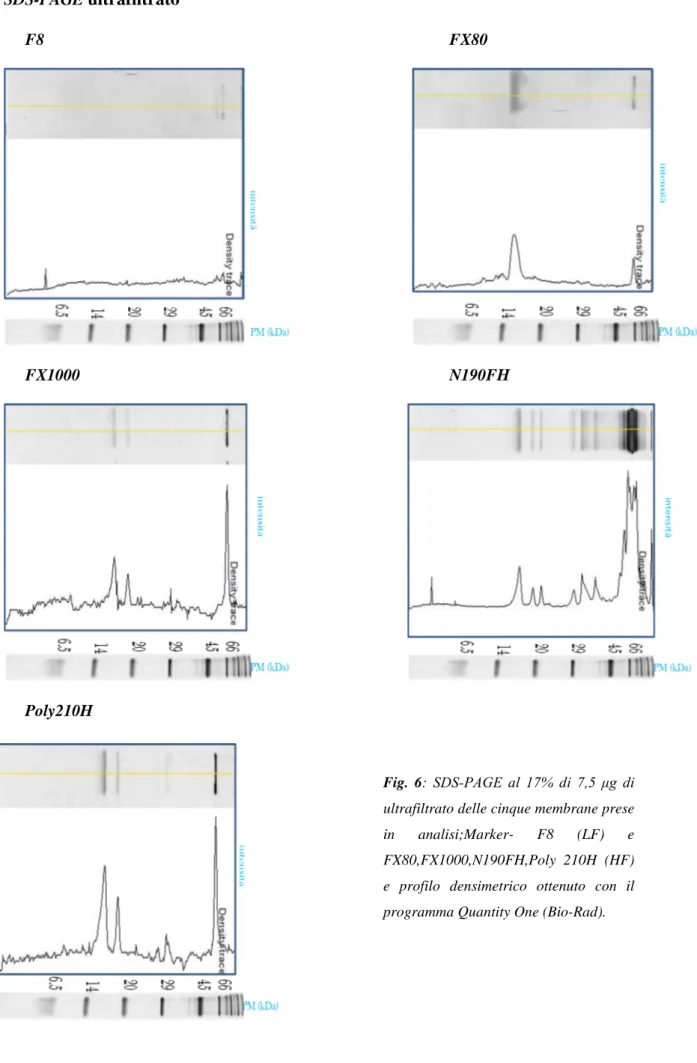
I risultati più interessanti sono stati ottenuti sul dializzato. L’utilizzo dell’SDS-PAGE ci ha consentito di determinare il profilo proteico dell’ultrafiltrato di diversi pazienti. Dallo studio degli ultrafiltrati, in accordo con l’ analisi biochimica, emerge che le membrane ad alto flusso hanno una maggior capacità di trattenere molecole a medio-alto peso molecolare rispetto alla membrana a basso flusso, infatti, come mostrato in fig.6-7, nella membrana F8 (lane evidenziata) non c’è traccia di proteine.
Fig.5: SDS-PAGE di siero inizio e fine dialisi. Sono stati caricati 15 μg di proteine totali in gel al 17% di poliacrilamide. Sotto le lanes sono riportati i nomi delle membrane, con la lettera M (marker), C (controllo) ID (inizio dialisi), FD (fine dialisi).
SDS-PAGEultrafiltrato
F8 FX80
FX1000 N190FH
Poly210H
Fig. 6: SDS-PAGE al 17% di 7,5 μg di ultrafiltrato delle cinque membrane prese in analisi;Marker- F8 (LF) e FX80,FX1000,N190FH,Poly 210H (HF) e profilo densimetrico ottenuto con il programma Quantity One (Bio-Rad).
Con la tecnica dell’SDS-PAGE abbiamo analizzato anche il pattern proteico di adsorbimento cinque membrane. L’adsorbimento oltre ad essere il meccanismo di depurazione responsabile della rimozione della maggior parte delle proteine ad alto peso molecolare, gioca un ruolo fondamentale nella determinazione della biocompatibilità delle membrane di dialisi.
Un accurato studio sull’efficacia delle membrane prevede anche l’analisi della loro bio(in)compatibilità. La biocompatibilità è fonte giustificabile di preoccupazione nell’ambito dei trattamenti emodialitici, essa è determinata prevalentemente dalle proteine adsorbite alla superficie delle membrane. Il contatto con il biomateriale di cui sono composte determina interazioni e conseguenti alterazioni degli elementi del sangue. La bioincompatibilità del dializzatore è considerata essere un importante fattore coinvolto nelle complicanze che possono accompagnare il trattamento emodialitico cronico (MHD). Infatti le proteine adsorbite possono innescare diverse vie di risposta biologica, come per esempio la cascata della coagulazione del sangue, l’attivazione del complemento, dei leucociti delle piastrine. L’attivazione di fattori immunitari può causare sia la comparsa di reazioni acute, sia un progressivo danno organico derivante dalla permanenza e dalla reiterazione dello stimolo flogistico. Ma non ci sono solo effetti negativi legati ai processi di adsorbimento, infatti l’adsorbimento a livello della membrana di dialisi di frammenti del complemento C3a e C5a, il fattore D (P.Valette 1999) e del chininogeno ad alto peso molecolare possono aumentare il profilo di biocompatibilità dei dializzatori. Inoltre l’adsorbimento può diminuire la concentrazione plasmatica di sostanze potenzialmente nocive quali la β2-microglobulina che si sa essere una sostanza amiloidogenica (M. Goldman, 1987) ed il fattore di necrosi tumorale (M. Bonomini, et al. 2006). Per studiare il profilo proteico di adsorbimento abbiamo trattato le membrane con soluzioni capaci di recuperarne dalla superficie le proteine rimaste adsorbite (J. Mares, 2009) il protocollo èarticolato in due steps; il primo prevede il passaggio di una soluzione di EDTA 3mM, che è risultata essere la migliore nello staccare eritrociti e leucociti, il secondo di acido acetico al 40%. Il passaggio delle soluzioni ha permesso che le proteine adsorbite venissero rilasciate dalla superficie. In fig.7 è riportata un’SDS-PAGE frutto di uno studio preliminare svolto per individuare l’agente di eluizione migliore, il quale è risultato essere, per il secondo step, l’acido acetico.
Fig.7: SDS-PAGE al 12% di 20 μg di eluato di membrana trattata con SDS al 10% e PBS pH=7,4.
I lavori in letteratura presi in esame prevedevano un’ analisi del contenuto proteico di adsorbimento della superficie interna dei capillari che costituiscono il dializzatore, vale a dire la parte che viene a contatto col sangue. Per avere un quadro più completo del pattern proteico di adsorbimento delle membrane, nel nostro lavoro abbiamo analizzato anche l’eluato che deriva dal risciacquo della parete esterna dei capillari quella a contatto col liquido di dialisi.
Le proteine ottenute dall’eluizione con soluzioni di EDTA ed acido acetico sono state separate mediante SDS-PAGE (fig.8). I risultati dell’ SDS-PAGE sono stati analizzati con un programma, QuantityOne, che permette di avere una stima dell’intensità di ciascuna banda e quindi un’analisi semiquantitativa del contenuto proteico del campione.
M SieroFD F8 a F8 b FX80 a FX80 b
M SieroFD FX1000 a FX1000 b N190 a N190 b Poly a Polt b
Fig.8: SDS-PAGE al 12% di 15 μg di eluato di membrane dialitiche trattate con acido acetico: a) dalla superficie a
contatto con il sangue, b) dalla superficie della membrana a contatto con il liquido di dialisi e dallo spessore della membrana. Marker, Siero fine dialisi, membrana a basso flusso (F8), membrane al alto flusso (FX80, FX1000,N190H,Poly210H) .
La concentrazione totale media di proteine che siamo riusciti ad eluire dalle diverse membrane per quanto riguarda l’eluizione con acido acetico (parte a contatto col sangue sommata alla parte dello spessore della membrana) sono: 493 mg/l per la F8; 397,5 mg/l per la FX80; 420,5 mg/l per la FX1000; 2053 mg/l per la N190H; 495 mg/l per la Ploly210H. Mentre per l’EDTA: 212,4 mg/l per la F8; 250,24 mg/l per la FX80; 280,14 mg/l per la FX1000, 1970 mg/l per la N190H; 253,8 mg/l per la Poly210H. La differenza della concentrazione proteica nei due eluati non è statisticamente significativa (P=0,67). La fig.9 mostra la quantità totale di proteine che siamo riusciti ad eluire dalle diverse membrane. Dall’istogramma di fig.9 si può ottenere un’ informazione indicativa sulla capacità depurativa delle membrane. Nei grafici di fig.10-11 sono riportate le distribuzioni, secondo tre range di pesi molecolari (70-45; 44-10; <10) delle proteine adsorbite sia nella parte della superficie che resta a contatto col sangue (fig.10) sia nello spessore della membrana (fi.11). la membrana N190 e Poly sembrano essere quelle che nel lato sangue trattengono maggiormente proteine ad alto peso molecolare (70-45 kDa), mentre in tutte le membrane si ritrovano in percentuale maggiore molecole dell’ intervallo (44-16). Mentre nello spessore delle membrane N190FH e Poly210H è evidente che restano adsorbite maggiormente proteine ad alto peso molecolare (70-45 kDa) nella F8 e FX80 esclusivamente proteine appartenenti all’intervallo 15-4 kDa e nello spessore della FX1000 invece restano proteine dell’intervallo 44-16 kDa.
Paziente A
Paziente B
Fig.9 (A-B): Quantità di proteine eluite in totale, suddivisa in quantità proveniente dalla superficie a contatto col sangue e dalla superficie a contatto con il liquido di dialisi e quantità proveniente dalla spessore delle membrane, in
Fig.10: Distribuzione percentuale delle proteine che rimangono adsorbite sulla superficie a contatto col sangue nelle cinque membrane secondo tre intervalli di peso molecolare (70-45, 44-16,15-4 kDa). Dati ottenuti per mezzo del software Quantity One (BioRad).
Fig.11: Distribuzione percentuale delle proteine adsorbite nello spessore della membrana nelle cinque membrane secondo tre intervalli di peso molecolare (70-45, 44-16,15-4 kDa). Dati ottenuti per mezzo del software Quantity One (BioRad).
2 DE SDS-PAGE
I risultati ottenuti in SDS-PAGE sono stati ulteriormente approfonditi mediante elettroforesi bidimensionale. Anche in questo caso siamo partiti da 2DE effettuata con i sieri prelevati prima e dopo il trattamento dialitico fig.10 (A-B-C-D). Già dal confronto dei due gels presi ad esempio, emerge che la membrana N190FH (alto flusso), rispetto alla F8 sottrae al sangue un maggior numero di proteine soprattutto ai pesi molecolari medio alti. Ma anche in questo caso l’ultrafiltrato ci ha fornito i risultati migliori fig.11. Nel dializzato della membrana N190 FH (fig.11D) possiamo notare numerosi spot proteici nel range 20-68 kDa. Anche nell’ultrafiltrato della Poly 210H (fig.11E) sono presenti, anche se in quantità minore rispetto alla N190FH, proteine di peso molecolare compreso tra 14,5-68 kDa. Le 2DE del dializzato con FX80 e FX1000 hanno evidenziato pochi spot proteici con un MW stimato intorno a 10-12 kDa. Nessuno spot proteico è stato rilevato nell’elettroforesi di ultrafiltrato della membrana LF F8(Fig.11 A). In Tabella 1 sono riportate le proteine identificate, mediante massa, nell’ultrafiltrato delle cinque membrane.
Siero Inizio e Fine Dialisi
Fig.11 : 2DE al 12% di 80 μg siero a) prelevato prima del trattamento dialitico con membrana ad alto flusso N190; b) dopo il trattamento dialitico con membrana N190; c) siero prelevato prima del trattamento dialitico con membrana a basso flusso F8; d) dopo il trattamento dialitico con membrana F8. Le ellissi indicano alcuni spots presenti prima
Ultrafiltrato
Fig.12: 2DE-SDS-PAGE al 12% di 300 μg di campione A): UF proveniente da una membrana F8; B) membrana FX80; C) membrana FX1000; D) membrana N190; E) membrana Poly 210 H.
Le membrane non sono inerti, infatti il contatto con il biomateriale di cui sono composte determina interazioni e conseguenti alterazioni degli elementi del sangue e questo può avere ripercussioni a
molecolare, è uno step importante per capire come migliorare le prestazioni delle membrane e limitare le ripercussioni cliniche. Avvalendoci della 2DE siamo riusciti a separare il contenuto proteico degli eluati delle membrane in studio. In totale abbiamo analizzato 25 gels derivati da eluati, plasma inizio e fine dialisi ed ultrafiltrati in fig.12-13-14-15-16 sono riportati 10 gels rappresentativi rispettivamente di quattro membrane ad alto flusso (FX80, FX1000,N190,Poly) ed una a basso flusso (F8).
Eluati: lato sangue e spessore membrana
A
Fig13: 2DE-SDS PAGE al 12% di 300 μg di eluato di membrana a basso flusso F8; A) dalla superficie della membrana a contato con il sangue, B) dallo spessore della membrana.
A B
Fig14: 2DE-SDS PAGE al 12% di 300 μg di eluato di membrana ad alto flusso FX80 A) dalla superficie della membrana a contato con il sangue, B) dallo spessore della membrana.
A B
Fig16: 2DE-SDS PAGE al 12% di 300 μg di eluato di membrana ad alto flusso N190 A) dalla superficie della membrana a contato con il sangue, B) dallo spessore della membrana.
A B
Nella membrana Nipro Superflux 190FH (fig. 15) sia a livello della parte a contatto col sangue sia a livello dello spessore, possiamo osservare la presenza di proteine con peso molecolare compreso tra 70 e 6,5 kDa. Nella membrana Fresenius F8 (fig.12) restano adsorbite nella parte a contatto col sangue proteine appartenenti ad un ampio intervallo di pesi molecolari (da110 a 6,5 kDa), l’identificazione ha mostrato che si tratta in prevalenza, soprattutto a livello dei bassi pesi molecolari, di emoglobina che non ha valore nell’ottica di questo studio e albumina e suoi frammenti. La presenza di frammenti di albumina è giustificabile con l’elevata concentrazione nel plasma dei pazienti con sindrome uremica (C. Donadio et al.2012). Inoltre la letteratura ci informa largamente sull’aumento della concentrazione di alcune proteine quali la β2-microglobulina (De Sain-van der Velden et al., 1998), e Retinol Binding Protein (Yamada et al., 1997) in presenza di infiammazione o sindrome nefrotica.
Spettrometria di massa
Una volta separate le proteine mediante 2DE e confrontato i gels, anche con l’ausilio del software PDQuest (BioRad) per identificare le proteine presenti nei campioni, sieri, ultrafiltrati ed eluati, abbiamo prelevato gli spots e gli abbiamo fatti analizzare mediante spettrometria di massa, in particolare per questo lavoro l’analisi è avvenuta con un analizzatore a tempo di volo MALDI (Matrix assisted laser desorption ionisation) TOF/TOF. La tabella 1 mostra le proteine identificate nei sieri, sono elencate quelle che dal confronto dei gels risultano presenti nel sangue prima del trattamento dialitico ed assenti alla fine del trattamento. In tabella 2 sono riportate le proteine identificate negli ultrafiltrati delle cinque membrane mentre, in tabella 3 troviamo tutte le 26 proteine che costituiscono una parte del proteoma di adsorbimento, identificate nell’eluato delle
flusso. In fig.17 è riportato un gel riassuntivo. Il gel è il risultato di una 2DE-SDS-PAGE dell’eluato della membrana N190 H (parte a contatto col sangue). Sul gel sono indicati i nomi delle proteine identificate. Per quanto riguarda la biocompatibilità un dato importante è la presenza del fattore D del complemento (Cheung A.K, 1990) nel pattern proteico di adsorbimento delle membrane ad alto flusso, eccetto della FX80, il suo adsorbimento, insieme al C3a (Pascal M., et al. 1996), pare essere coinvolto nel miglioramento della biocompatibilità. Le proteine saranno ulteriormente analizzate per approfondire lo studio sulla efficacia depurativa e biocompatibilità delle membrane.
F8 MW(kDa) A β2-Microglobulin 12 B Hemoglobin 16 C Transthyretin 16 D Clusterin 43 N190FH MW(kDa) A Transthyretin 16 B Complement Factor D. 25 C Haptoglobin 39 D Clusterin 43 E Fibrinogen γ-chain 50 F Fibrinogen β-chain 53 G Serum Albumin 69 H Serumtransferrin 77
FX80 MW(kDa) Albumin frag. 29-14-<6,5 β2-Microglobulin 12 FX1000 MW(kDa) Albumin frag. 29-14-<6,5 β2-Microglobulin 12 Retinol Binding P. 21 Albumin frag. 48 N190FH MW(kDa) Albumin frag. 29-14->6,5 B2-Microglobulin 12 Retinol Binding p. 21 Complement Factor D 25 A1-Antitripsin 54 Serum Albumin 69 Poly 210H MW(kDa) Albumin frag. 29-14-<6,5 Cystatin C 11,5 β2.Microglobulin 12 Retinol Binding p. 21
F8 MW(kDa) Albumin frag. 29-14-<6,5 B2-microglobulin 12 Hemoglobin <14 Retinol Binding p. 21 Aurora Kinase A 46 Serum Albumin 69 FX80;FX1000;Poly210H MW(kDa) Albumin frag. 14<6,5 Apolipoprotein CI 8,8 Apolipoprotein CIII 11 Albumin frag. 29;14;<6,5 β2-Microglobulin 12 Serum Amyloid p. 14 Transthyretin 16 Retinol Binding p. 21 Complement Factor D. 25 Ig chain K 25 Apolipoprotein AI 28 Apolipoprotein AIV 29 Haptoglobin 39
Aurora Chinase (noFX1000) 46
Fibrinogen γ-chain 50 α-1 Antitrypsin 54
N190FH MW(kDa) Albumin frag. 14<6,5 Protein S 100-A12-A8-A9 6,5-14 Apolipoprotein CII 8,8 Apolipoprotein CIII 11 Cystatin C 11,5 β2-Microglobulin 12 Hemoglobin <14 Serum Amyloid A p. 14 Transthyretin 16 Retinol Binding p. 21 Complement Factor D 25 Apolipoprotein A-I 28 Haptoglobin 39 Clusterin 43 β2-Glycoprotein 45 Apolipoprotein AIV 46 Aurora kinase A 46 Fetuina-A 49 Fibrinogen γ-chain 50 Fibrinogen β chain 53 α-1 Antitrypsin 54 Vimentin 58 Vitamin D-Binding p. 58
Fig.18: Immagine rappresentativa di tutte le proteine identificate mediante spettrometria di massa. Il gel al 12% è il
risultato di una 2DE di eluato della membrana N190 H (parte a contatto col sangue). La figura più piccola mostra una parte di un altro gel di eluato di N190H (parte a contatto col liquido di dialisi).
Per concludere, un aspetto molto interessante da approfondire è rappresentato dalle modifiche post-traduzionali. Una delle funzioni della spettrometria, oltre a fornire misure precise della massa dei polipeptidi o dei loro prodotti di frammentazione proteolitica ed a descriverne la struttura primaria, quindi, di ottenerne la completa identificazione, è quella di poter individuare modifiche post-traduzionali. In fig.18 è riportato un esempio di un’elettroforesi in cui mediante spettrometria abbiamo identificato due proteine con fosforilazione. L’analisi di queste ed altre modifiche post-traduzionali identificate potrebbe far luce su alcuni meccanismi fisiopatologici. Inoltre la presenza
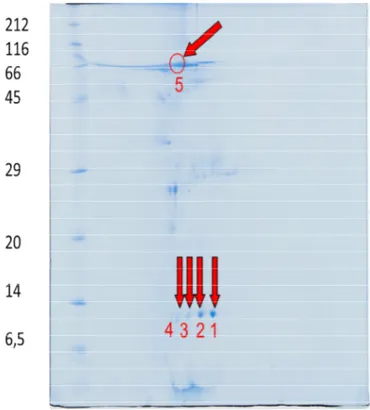
Fig. 19: SDS-PAGE 12% di ultrafiltrato, membrana Poly210, le frecce indicano gli spots identificati con la massa e le
relative modifiche post-traduzionali: Spot 1 e 2→ β2-microglobulina (spot 1 forma fosforilata della proteina) Spot 5 → sieroalbumina Spot 3, 4 sieroalbumina frammenti (spot 3 forma fosforilata della proteina).