Tecniche software
4.1 Introduzione
In questo capitolo saranno descritte tutte le tecniche software utilizzate per l’elaborazione e l’analisi delle immagini ottenute fotografando le cellule in coltura. Gli algoritmi implementati in Matlab verranno invece riportati integralmente nell’Appendice B.
Lo scopo di queste tecniche di analisi è quello di estrarre il maggior numero possibile di informazioni sulla morfologia, sulla topologia e sull’organizzazione delle cellule.
L’importanza dello studio metrico della morfologia delle cellule di Purkinje e della connettività delle reti neurali, deriva dal fatto che nella patologia dell’autismo esse subiscono una forte alterazione e soltanto dopo averne compreso e misurato in maniera accurata e precisa tutti i dettagli, è possibile chiarire quali sono i danni a cui esse vanno incontro.
Per quanto riguarda le cellule di Purkinje, l’estrazione di variabili relative all’albero dendritico può essere realizzata racchiudendo la cellule all’interno di un sistema di riferimento utilizzando il metodo di Sholl [14], mentre le variabili globali possono essere estratte direttamente dall’immagine elaborata. Per effettuare la scelta delle variabili da estrarre ci si è bastai su alcuni studi morfometrici realizzati su diversi tipi cellulari [56, 77, 78].
Per quanto riguarda le reti invece, è possibile modellarle come grafi ed estrarre le variabili di interesse applicando tutte le tecniche della teoria dei grafi [79, 80, 81].
Queste analisi possono essere realizzate in modo quasi completamente automatico, implementando degli appositi algoritmi che operano sulle immagini ottenute dopo una serie di elaborazioni eseguite sulle fotografie delle cellule. Il programma utilizzato per l’implementazione di tali algoritmi è Matlab. In particolare è stato utilizzato un insieme di tool di Matlab chiamato GUIDE che consente di creare in modo piuttosto semplice e veloce una GUI ovvero una Grafical User Interface.
Questo strumento, sviluppato e implementato per l’analisi delle immagini delle cellule di Purkinje, è stato applicato anche all’analisi delle reti dei neuroni mesencefalici, riorganizzando gli algoritmi già implementati nel lavoro precedente a quello presente.
Nella prima parte del capitolo sarà spiegato in che cosa consiste una GUI, come viene progettata e programmata e quali sono i vantaggi che comporta. Si passerà quindi ad una descrizione dettagliata delle diverse tecniche implementate per l’estrazione delle variabili di interesse dalle immagini di Purkinje. Ciascuna componente della GUI verrà considerata separatamente e verrà spiegato il tipo e il significato della variabile o delle variabili che vengono misurate eseguendo l’algoritmo corrispondente e come questo è stata implementato con i comandi Matlab. Per ogni tecnica utilizzata la cui comprensione richieda una qualche conoscenza teorica, verrà riportata brevemente la teoria a cui si è fatto riferimento. Per quanto riguarda l’analisi della frattalità, il suo significato e la sua rilevanza nei sistemi biologici, verranno illustrati con particolare cura e attenzione nell’Appendice A. Questa tecnica infatti fornisce indicazioni molto importanti sull’organizzazione di un sistema ed stata applicata anche alle immagini in fluorescenza delle reti di
neuroni mesencefalici al fine di comprendere se può esistere una qualche relazione tra organizzazione locale e globale.
Lo stesso tipo di descrizione verrà fatto, in maniera più concisa, in relazione alla GUI in cui sono stati riorganizzati gli algoritmi implementati per l’analisi delle reti mesencefaliche.
Entrambe le GUI prevedono la costruzione di una matrice di dati in cui vengono raccolte tutte le variabili estratte dalle immagini, in diversi istanti di tempo, di una stessa cellula o rete. La matrice viene inizializzata a zero all’inizio della GUI ed ogni volta che viene eseguita un’operazione il cui risultato è il calcolo di una variabile, questa viene aggiunta in una posizione specifica della matrice. I dati raccolti possono essere quindi poi estratti e visualizzati tramite grafici, in modo tale da trarre delle informazioni utili, sia statiche che dinamiche, sulla morfologia delle cellule e la topologia delle reti. Oltre a queste tecniche, che consentono l’analisi di diverse cellule o reti in diversi istanti di tempo, sono state utilizzate anche delle tecniche di analisi multivariata. Queste tecniche sono la 3-way PCA, che ha permesso di confrontare e analizzare ulteriormente parte delle variabili estratte dall’analisi delle cellule di Purkinje, e l’analisi di clustering che, applicata alle immagini dei neuroni mesencefalici in fluorescenza, ha consentito una migliore comprensione dell’organizzazione topologica delle reti.
Anche queste tecniche sono state realizzate con appositi algoritmi implementati in Matlab. Mentre gli algoritmi per l’analisi di clustering sono stati implementati in questo lavoro di tesi, per quanto riguarda la 3-way PCA, sono stati utilizzati degli algoritmi implementati da Leardi e collaboratori. In questo capitolo verranno descritte tutte le tecniche utilizzate sia per l’elaborazione delle immagini che per la loro analisi mentre i risultati ottenuti dalla loro applicazione verranno riportati e discussi nel prossimo capitolo.
4.2 Grafical User Interface (GUI)
La GUI è uno schermo grafico che contiene diversi componenti che permettono all’utente di svolgere compiti interattivi. I componenti di una GUI possono essere menù, bottoni, liste, assi per la visualizzazione di immagini o grafici, etc. Ciascuno di essi è associata ad una parte di programma, chiamata “callback”, scritta dall’utente nel linguaggio di programmazione di Matlab, che viene eseguita quando l’utente compie una particolare operazione sul componente a cui la callback corrisponde, come ad esempio il clic del mouse, la selezione di un campo del menu, o il passaggio del cursore.
Per creare una GUI viene utilizzato un insieme di tool forniti da Matlab chiamato GUIDE. Questi tool semplificano la creazione della struttura della GUI e la sua programmazione.
Figura 4.1 Schermata iniziale della GUI
Il primo passo del processo di creazione della GUI è l’inserimento all’interno dell’area della GUI di tutti i componenti necessari. L’aspetto della GUI può essere cambiato secondo le proprie esigenze variandone le dimensioni, modificando le caratteristiche dei componenti e disponendoli in maniera opportuna nello spazio.
Una volta salvata la grafica della GUI, Matlab genera automaticamente un
M-file che può essere usato per programmare la GUI. Questo M-file contiene
inizialmente il codice per inizializzare la GUI e una struttura all’interno della quale poter inserire le callback ciascuna delle quali è associata ad uno dei componenti inseriti nell’area della GUI. Le callback sono parti di programma che vengono eseguite quando l’utente agisce sui componenti a cui esse corrispondono. Una volta creato l’M-file quindi questo può essere utilizzato
per implementare gli algoritmi necessari ad eseguire le operazioni volute [82, 83].
La scelta di utilizzare una GUI è dovuta al fatto che essa offre numerosi vantaggi rispetto alla programmazione in un editor di Matlab, soprattutto quando è necessario eseguire molte operazioni correlate tra loro ed estrarre un grande numero di variabili come in questo caso. Essa infatti consente di avere un programma, anche molto lungo in unico file, ma permette anche all’utente di scegliere se eseguire tutto il programma insieme o se accedere a diverse parti del programma separatamente e nell’ordine che egli stabilisce a seconda di come attiva i componenti. Essa inoltre permette un facile interfacciamento al programma anche da parte di utenti diversi dal programmatore e consente la visualizzazione di un insieme di dati e di immagini all’interno dello stesso schermo. In questo modo è possibile avere una visione globale del risultato delle diverse operazioni eseguite.
4.3 Analisi delle cellule di Purkinje
4.3.1 Elaborazione delle immagini
Per poter analizzare le immagini delle cellule con le tecniche che verranno descritte nei prossimi paragrifi, è necessario ottenere immagini binarie in cui è presente lo scheletro della cellula in nero su sfondo bianco. Per scheletro si intende la struttura della cellula ridotta a linee di spessore pari ad un pixel. L’elaborazione viene fatta a partire dalle fotografie realizzate ad un ingrandimento di 20X.
L’elaborazione della fotografie realizzate alle cellule è risultata piuttosto lunga e complessa data la bassa risoluzione delle fotografie ottenute e lo
scarso contrasto tra cellule e sfondo. La procedura di elaborazione è stata realizzata utilizzando Mipav in combinazione con Image J, due software specifici per l’elaborazione delle immagini scaricabili gratuitamente in rete e si articola in diverse fasi:
1. Crop: dato che nella stessa fotografia sono presenti più cellule, ciascuna cellula di interesse deve essere tagliata dal resto dell’immagine e copiata in un altro file. Essa viene copiata mantenendo le sue dimensioni nell’immagine di partenza. Il crop è stato realizzato utilizzando Adobe Photoshop (Fig. 4.2).
Figura 4.2 Crop dell’immagine originaria
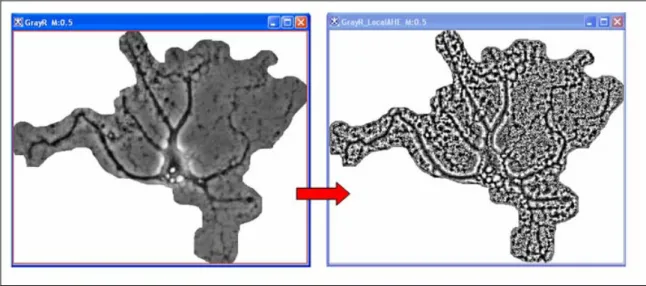
2. Equalizzazione dell’istorgamma: l’immagine tagliata è stata caricata in Mipav ed è stata eseguita una Macro che trasforma l’immagine in livelli di grigio, seleziona l’immagine sul canale rosso e di questa esegue l’equalizzazione dell’istogramma “Neighborhood adaptive”. In questo modo viene aumentato il contrasto e vengono messi in risalto i contorni della cellula (Fig. 4.3).
Figura 4.3 Equalizzazione dell’istogramma dell’immagine in livelli di grigio
3. Selezione del “Volume Of Interest” (VOI): sempre utilizzando Mipav è possibile selezionare la parte dell’immagine che si vuole estrarre. Posizionando il cursore del mouse su un punto della cellula viene costruita una regione che comprende tutti i pixel che hanno un livello di grigio simile a quello selezionato. L’ampiezza dell’intervallo dei livelli di grigio può essere regolata (Fig. 4.4).
4. Immagine binaria: viene eseguita poi un’altra macro in Mipav che trasforma l’area selezionata in una maschera, ovvero in un immagine binaria (Fig. 4.5).
Figura 4.5 Realizzazione della maschera
5. Dilatazione e ottenimento dello scheletro: la maschera viene portata in Image J, subisce una serie di dilatazioni in modo tale che le varie parti che compongono la cellula vengano collegate tra loro ed infine l’immagine dilatata viene ridotta a scheletro (Fig. 4.6).
Figura 4.6 Riduzione a scheletro della maschera
4.3.2 Implementazione della GUI
Viene riportato in Fig. 4.7 lo schema generale della GUI implementata in Matlab per l’analisi della morfologia delle cellule di Purkinje.
Nei prossimi paragrafi verrà preso in considerazione ciascun pannello della GUI e verrà illustrato quale funzione svolge l’algoritmo ad esso correlato.
4.3.2.1 Caricamento dell’immagine
Le fotografie delle cellule e le immagini ottenute dalla loro elaborazione, sono state caricate all’interno della GUI per effettuare l’analisi.
Viene riportato in Fig. 4.8 il pannello per il caricamento delle immagini.
Figura 4.8 Pannello per il caricamento e la visualizzazione dell’immagine
Dopo aver selezionato negli edit la coltura a cui l’immagine appartiene, il numero che corrisponde ad una specifica cellula ed i giorni totali in cui questa è stata mantenuta in coltura, il programma carica tutte le foto e le immagini elaborate relative a quella cellula in diversi istanti di tempo. Negli assi è possibile visualizzare lo scheletro della cellula, l’immagine binaria o la fotografia selezionando il campo opportuno del menù. È possibile anche visualizzare le fotografie scattate in giorni diversi in successione, in modo tale da vedere come si è sviluppata la cellula. Questo può essere fatto cliccando sul bottone “Filmato”.
Le immagini relative a giorni diverse vengono analizzate una alla volta: una volta caricata la prima immagine vengono eseguite in successione tutte le
callback dopodiché viene caricata la seconda immagine e così via fino a che
le immagini non sono esaurite.
4.3.2.2 Analisi di Sholl
La prima tecnica applicata alle immagini elaborate delle cellule è stata l’analisi di Sholl. L’analisi di Sholl è un metodo per studiare in maniera quantitativa il pattern di distribuzione radiale dell’arborizzazione dendritica intorno al soma della cellula [14]. Essa fornisce indicazioni sulla geometria dei dendriti, sulla ricchezza delle ramificazioni e sul pattern di biforcazione dei dendriti e deve essere applicata ad immagini binarie che rappresentino lo scheletro della cellula.
L’analisi si basa su quattro punti principali:
1. Costruzione di cerchi concentrici centrati nel centro del soma della cellula
2. Conta del numero di intersezioni che i dendriti formano con ciascun cerchio
3. Definizione di alcune variabili che caratterizzino l’andamento del numero di intersezioni con l’aumentare del raggio dei cerchi
4. Scelta di tecniche matematiche appropriate per il processamento e la presentazione dei dati
e può essere applicata in tre diverse varianti [77]:
1. Metodo di Sholl lineare: plot del numero di intersezioni per area del cerchio in funzione del raggio
2. Metodo di Sholl semi-log: plot del logaritmo del numero di intersezioni per area del cerchio in funzione del raggio
3. Metodo di Sholl log-log: plot del logaritmo del numero di intersezioni per area del cerchio in funzione del logaritmo del raggio.
E’ stato dimostrato che applicando il secondo e il terzo metodo si ha che in almeno uno dei due casi i dati approssimano una linea retta.
E’ stato visto che in alcuni casi funzione meglio metodo Semi-Log, in altri il metodo Log-Log; per scegliere il metodo migliore nel caso specifico è necessario quindi applicarli entrambi e vedere in quale dei due casi i dati approssimano meglio una linea retta.
Utilizzando il metodo lineare si possono determinare alcuni parametri che possono servire per discriminare cellule diverse:
• rc (raggio critico): raggio del cerchio sul quale si ha il maggior numero
di intersezioni
• Nm : numero massimo di intersezioni
• indice di ramificazione di Schoenen: rapporto tra il numero massimo di intersezioni e il numero di dendriti primari (dendriti che hanno origine direttamente nel soma della cellula)
Le variabili utilizzate per caratterizzare come varia il numero di intersezioni con il raggio nei metodi Semi-Log e Log-Log, sono invece le seguenti:
• k (coefficiente di regressione di Sholl): pendenza della retta che fitta i dati. È una misura del rate di decadimento del numero di ramificazioni con la distanza dal soma
• Δ (rapporto di determinazione) : 22 LL LS R R = Δ
permette di valutare i risultati ottenuti con l‘applicazione dei due metodi: se Δ<1 il metodo Log-Log è migliore del metodo Semi-Log, se Δ>1 il metodo Semi-Log è migliore del metodo Log-Log.
Per poter applicare questa analisi, il primo passo consiste nell’individuare le coordinate del centro dello scheletro della cellula, il raggio minimo a partire dal quale costruire i cerchi ed il raggio massimo. Le prime due variabili sono state determinate con un clic del mouse sull’immagine mentre il raggio massimo è stato calcolato misurando la distanza tra il centro ed il pixel appartenente allo scheletro più distante dal centro. I valori risultanti vengono visualizzati negli edit corrispondenti. Viene riportato in Fig. 4.9 il pannello in cui vengono calcolate queste variabili.
Figura 4.9 Pannello per l’individuazione del centro, del raggio minimo e del raggio
massimo
A questo punto è stato possibile racchiudere lo scheletro all’interno del sistema di riferimento di Sholl. In realtà si è scelto di costruire due sistemi di riferimento: uno a numero di cerchi costanti (10), cioè in cui il numero di cerchi non varia da un’immagine all’altra, e uno a distanza inter-cerchio costante (60 pixel) in cui il numero di cerchi varia a seconda delle dimensioni
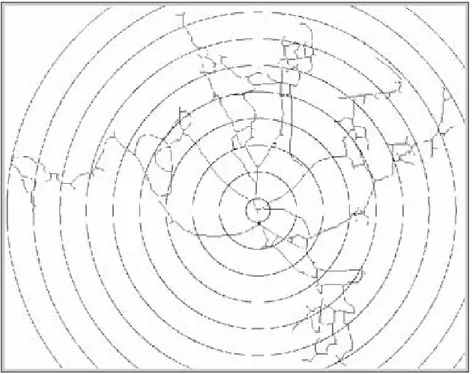
del cellula. La costruzione di ciascuno dei due sistemi di riferimento e l’estrazione delle variabili relative a ciascun sistema, vengono realizzate in successione. In entrambi i casi al clic del mouse sul bottone “Disegna”, vengono disegnati negli assi i cerchi sovrapposti alla cellula. Viene riportata in Fig. 4.10 l’immagine dello scheletro della cellula racchiuso all’interno del sistema di riferimento a cerchi costanti.
Figura 4.10 Sistema di riferimento di Sholl
Sono state quindi calcolate le intersezioni per ciascun cerchio e tutte le variabili caratteristiche dei metodi lineare, Semi-Log e Log-Log. Il calcolo delle intersezioni è stato fatto sommando l’immagine dei cerchi e l’immagine dello scheletro. Dato che entrambe sono immagini binarie, in corrispondenza delle intersezioni si avranno dei gruppi di pixel di valore 2. Contando questi gruppi tramite l’utilizzo dell’operatore di segmentazione, è possibile ricavare il numero di intersezioni. I valori delle intersezioni ottenute per ciascun cerchio vengono visualizzate in successione all’interno delle list-box. Il
calcolo delle intersezioni viene effettuato al clic del mouse sul bottone “Calcola Intersezioni”.
Conoscendo il numero di intersezioni per ciascun cerchio è possibile poi ricavare tutti gli altri parametri il cui valore viene visualizzato negli edit corrispondenti (Fig. 4.11).
Figura 4.11 Pannello per l’estrazione delle variabili dell’analisi di Sholl
4.3.2.3 Percorso minimo, vettori delle lunghezze minime ed angoli tra i vettori
Una volta racchiuso lo scheletro della cellula in un sistema di riferimento è stato possibile calcolare anche il percorso minimo, i vettori delle lunghezze minime e gli angoli compresi tra tali vettori. Queste misurazioni sono state fatte per entrambi i sistemi di riferimento e considerando due traiettorie: una che va dall’estremità della cellula (intesa come il pixel più distante dal centro) al centro e l’altra che va dall’estremità dell’asse principale al centro. Come
asse principale si intende il dendrite di diametro maggiore rispetto agli altri che determina la direzione in cui si estende la cellula.
Mentre l’estremità della cellula viene calcolata automaticamente dal programma, l’estremità dell’asse principale viene individuata con un clic del mouse.
I vettori delle lunghezze minime vengono calcolati a partire dall’estremità fino al centro della cellula: vengono misurate le distanze tra l’estremità e tutte le intersezioni del cerchio precedente e viene presa la minima, l’intersezione per cui si ottiene la minima distanza diventa la nuova estremità e viene eseguito lo stesso procedimento fino a che non sia trova l’intersezione sul cerchio di raggio minimo. L’ultimo vettore viene calcolato misurando la distanza tra questa intersezione ed il centro.
Una volta determinati i vettori delle lunghezze minime, sia in modulo che in direzione, sono stati calcolati gli angoli compresi tra due vettori consecutivi. Sommando infine i moduli di tutti i vettori si può determinare il precorso minimo dal centro all’estremità della cellula o dell’asse principale. Il calcolo viene effettuato rispettivamente al clic del mouse sul bottone “Estremità” o “Asse principale” e i valori ottenuti vengono visualizzati negli edit corrispondenti (Fig 4.12).
Figura 4.12 Pannello per la misurazione del percorso minimo, dei vettori delle lunghezze
I vettori delle lunghezze minime vengono visualizzati negli assi. Viene riportato in Fig. 4.13 un esempio di costruzione di un percorso dall’estremità al centro della cellula.
Figura 4.13 Percorso dall’estremità al centro della cellula
Cliccando poi sui bottoni “Grafico Estremità” o “Grafico Asse principale”, è possibile graficare i moduli dei vettori delle lunghezze minime e degli angoli calcolati rispettivamente lungo il percorso dall’estremità al centro o dall’estremità dell’asse principale al centro, in funzione dei cerchi.
4.3.2.4 Estensione radiale della cellula
L’estensione radiale della cellula è stata determinata calcolando la distanza tra il centro della cellula ed il pixel, appartenente all’immagine della cellula, più lontano da questo. Il centro viene selezionato con un clic del mouse per cui può essere affetto da errori: per aumentare la precisione della misura, il
calcolo della distanza viene realizzato facendo la media su misure ripetute realizzate effettuando più clic del mouse. Il risultato, calcolato a seguito del clic del mouse sul bottone “Calcola”, viene visualizzato nell’edit corrispondente (Fig 4.14).
Figura 4.14 Pannello per il calcolo dell’estensione radiale della cellula
4.3.2.5 Dimensione del soma
La dimensione del soma viene calcolata approssimando il soma ad un’ellisse e andando quindi a determinare l’asse maggiore e l’asse minore.
Dato che nello scheletro della cellula il soma è rappresentato come un punto, per determinarne l’area è necessario visualizzare negli assi la fotografia della cellula.
Gli estremi degli assi vengono individuati con un clic del mouse dopodiché l’algoritmo determina la lunghezza degli assi misurando la distanza tra i punti e quindi, applicando la formula dell’area dell’ellisse, calcola l’area del soma in pixel. Il risultato, calcolato a seguito del clic del mouse sul bottone “Calcola”, viene visualizzato nell’edit corrispondente (Fig. 4.15).
4.3.2.6 Area del cono in cui è racchiusa la cellula
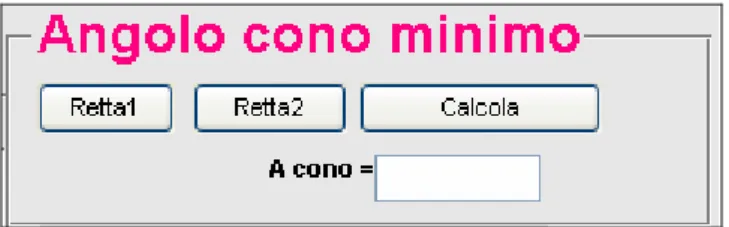
Nel calcolo dell’area del cono in cui è racchiusa la cellula si devono distinguere due casi. Se la cellula si estende in tutte le direzioni, l’angolo del cono viene posto pari a 360°. Per dare al programma questa indicazione viene effettuato un clic del mouse con il tasto destro. Se invece la cellula si estende lungo un direzione preferenziale è necessario calcolare l’angolo di estensione. Per far questo viene selezionato con un clic del mouse il vertice del cono che corrisponde alla posizione del soma, dopodiché compare la prima direttrice che ha origine nel vertice e può essere posizionata con il mouse in modo che sia tangente alla cellula. La selezione del vertice e il posizionamento del mouse, vengono realizzati cliccando sul bottone “Retta1”. Una volta posizionata la prima direttrice, cliccando sul bottone “Retta2”, compare la seconda, sempre con origine nel centro, che deve essere tangente allo scheletro dal lato opposto (Fig. 4.16).
A questo punto, ciccando sul bottone “Calcola”, viene calcolato l’angolo compreso tra le due direttrici utilizzando il teorema di Carnot e il risultato viene visualizzato nell’edit (Fig. 4.17).
Figura 4.17 Pannello per il calcolo dell’angolo del cono minimo in cui è racchiusa la
cellula
4.3.2.7 Dimensione frattale
L’analisi della frattalità è stata effettuata utilizzando degli algoritmi scaricati dalle librerie di Matlab che implementano il metodo del Box-counting.
Tale metodo consiste nel sovrapporre all’immagine una griglia quadratica, con maglie di lato r, e nel contare il numero di maglie occupate dall’oggetto, variando la dimensione delle maglie. Inizialmente r è pari alla dimensione dell’immagine, poi viene reso progressivamente più piccolo. Riportando il logaritmo del numero di maglie occupate dall’oggetto in funzione del logaritmo della dimensione della maglia, si ottiene un grafico interpolabile con una retta di pendenza S negativa. Tale pendenza costituisce una stima della dimensione frattale:
S DFrattale =
Il metodo è versatile, infatti può essere applicato a qualsiasi struttura piana e può essere esteso anche a strutture tridimensionali, semplice e facilmente implementabile in algoritmi veloci ed attendibili che analizzano immagini digitalizzate [84].
Un oggetto frattale è un oggetto che esprime la proprietà di autosimilarità: è dato dall’unione di un numero di parti che, ingrandite di un certo fattore, riproducono tutto l’oggetto; in altri termini è unione di copie di se stesso a scale differenti. Questa proprietà è espressa i sistemi biologici solo in un certo intervallo di dimensioni per cui non ha senso calcolare la dimensione frattale su tutto l’insieme di valori assunti dalla dimensione delle maglie della griglia. In questo caso si può calcolare la pendenza locale della retta che fitta i dati in corrispondenza dei valori assunti da r, graficare la pendenza in funzione di r e vedere in quale intervallo la curva che si ottiene è lineare. La dimensione frattale viene poi viene calcolata considerando solamente i punti appartenenti alla zona di linearità. Questo è quello che viene fatto nell’algoritmo
Box-counting slope ed è stato applicato alle immagini delle cellule.
Nel pannello della GUI che corrisponde all’analisi frattale, premendo il bottone “Box-counting” viene aperto il grafico che rappresenta l’andamento delle pendenze locali in funzione del raggio e la list-box viene popolata con i valori assunti da tale pendenze (Fig. 4.18).
Figura 4.18 Pannello per il calcolo della dimensione frattale
In base all’osservazione del grafico e dei valori nella list-box, negli edit in alto vengono indicati dall’utente il valore iniziale e il valore finale dell’intervallo di linearità e il programma restituisce nell’edit in basso il valore della dimensione frattale calcolata su questo intervallo.
4.3.2.8 Memorizzazione dei dati
Ogni volta che viene eseguita una callback, le variabili calcolate vengono inserite in una posizione specifica del data-matrix inizializzato all’inizio del programma. La struttura finale del data-matrix è quella riportata nella tabella seguente (Tab. 4.1). Nella prima riga sono indicati i nomi delle variabili salvate (l’indice 1 indica il sistema di riferimento a cerchi costanti, il 2 il sistema di riferimento a distanza inter-cerchio costante), nella seconda le colonne del data-matrix corrispondenti.
Intersezioni1 rc1 Nm1 Schoenen1 ksl1 Rsl1 kll1 Rll1 Δ1
1 ÷ 10 11 12 13 14 15 16 17 18
Pestr1 Lminestr1 Aestr1 Passe1 Lminasse2 Aasse1
19 20 ÷ 29 30 ÷ 38 39 40 ÷ 49 50 ÷ 59
Intersezioni2 rc2 Nm2 Schoenen2 ksl2 Rsl2 kll2 Rll2 Δ 2
60 ÷ 79 80 81 82 83 84 85 86 87
Pestr2 Lminestr2 Aestr2 Passe2 Lminasse2 Aasse2
88 89 ÷ 108 109 ÷ 128 129 130 ÷ 149 150 ÷ 169
Estensione Area Soma Angolo cono Dimensione frattale
170 171 172 173
Raggio minimo Raggio massimo xc yc n
174 175 176 177 178
Tabella 4.1 Data-matrix contenente le variabili estratte dall’analisi delle cellule di
Purkinje (rc: raggio critico, Nm: numero massimo di intersezioni, Schoenen: indice di Schoenen, ksl: coefficiente di regressione del metodo Semi-Log, Rsl: coefficiente di correlazione del metodo Semi-Log, kll: coefficiente di regressione del metodo Log-Log, Rll: coefficiente di correlazione del metodo Log-Log, Δ: rapporto di determinazione, Pestr: percorso minimo dall’estremità al centro, Lminestr: vettori delle lunghezze minime dall’estremità al centro, Aestr: angoli tra i vettori dall’estremità al centro, Passe: percorso minimo dall’estremità dell’asse principale al centro, Lminasse: vettori delle lunghezze minime dall’estremità dell’asse principale al centro, Aasse: angoli tra i vettori dall’estremità dell’asse principale al centro, xc: ascissa del centro, yc ordinata del centro, n: numero di valori di r scelti per il calcolo della dimensione frattale
Una volta applicata la GUI alle immagini di tutte le cellule, è possibile costruire un data-matrix globale dato dall’unione dei data-matrix relativi a ciascuna cellula.
Da questo data-matrix globale possono essere estratte le variabili necessarie per poter dedurre osservazioni statiche e dinamiche generali confrontando i grafici relativi a cellule diverse. Il metodo utilizzato per la costruzione del data-matrix e le osservazioni dedotte dall’esame dei grafici, verranno riportati nel prossimo capitolo.
4.3.3 La 3-way PCA (Analisi delle Componenti Principali)
Per analizzare l’importanza delle variabili estratte e le correlazioni che esistono tra di esse, è stata eseguita un’ulteriore analisi ovvero la 3-way PCA [85, 86]. In questo paragrafo verrà descritto in che cosa consiste tale tecnica mentre i risultati ottenuti dalla sua applicazione ai dati, verranno riportati nel prossimo capitolo.
La 3-way PCA è una tecnica di analisi multivariata che deriva dalla PCA, una tecnica molto nota utilizzata per l’analisi di dati che provengono dall’osservazione di un certo numero di variabili su un certo numero di soggetti od oggetti. Mentre la PCA viene applicata su una matrice di dati bidimensionale, la 3-way PCA è stata modellata per lo studio di dati raccolti all’interno di una matrice tridimensionale. Spesso infatti le variabili vengono misurate in un certo numero di situazioni o di istanti temporali per cui è necessario aggiungere una terza dimensione al data-matrix. Le tre dimensioni associate a tale data-matrix vengono chiamati i tre “modi” della matrice.
Un data-matrix tridimensionale potrebbe essere analizzato aggregando i dati su una delle tre dimensioni, oppure analizzando tutte le matrici bidimensionali contenute al suo interno. Queste analisi tuttavia non tengono conto delle
relazioni tra le variabili sulle tre dimensioni per cui potrebbero portare a conclusioni errate o incomplete. La forza della 3-way PCA è la capacità di analizzare i dati nelle loro correlazioni sulle tre dimensioni e quindi giungere a conclusioni complete ed esaustive.
Sia la PCA che la 3-way-PCA hanno lo scopo di riassumere tutte le informazioni contenute all’interno di un data-matrix in poche componenti e di farlo in modo efficace. Questo è utile in particolar modo nel caso di data-matrix tridimensionali. Infatti, senza una riduzione delle componenti da analizzare, una descrizione completa delle correlazioni tra le variabili sulle tre dimensioni, richiederebbe l’analisi di un elevatissimo numero di dati il che non è fattibile almeno che il data-matrix non sia di dimensioni ridotte.
Sono state proposti diversi modelli di analisi 3-way tra cui quelli più conosciuti sono:
• PARAFAC (Parallel Factor Analysis)
• 3MPCA (Three-mode Principal Component Analysis) • Tucker3 Analysis
Di seguito verrà descritta soltanto la tecnica Tucker3 dato che è quella che è stata utilizzata per l’analisi dei dati in questo lavoro di tesi e risulta anche essere il modello più generale.
Prima di parlare del modello Tucker3, verrà descritto brevemente in che cosa consiste la PCA dato che la 3-way PCA è una generalizzazione di tale tecnica. 4.3.3.1 Analisi delle Componenti Principali (PCA)
Lo scopo della PCA è quello di trovare un basso numero di variabili, che vengono chiamate “componenti”, costruite a partire dalle variabili osservate, in modo tale che esse racchiudano la maggiore quantità di informazione possibile contenuta nelle variabili osservate.
Data una matrice X,
I
×
J
, dove I sono gli oggetti e J le variabili, la PCA determina, attraverso una procedura iterativa, una matrice delle varianze delle componenti A I ×Q e una matrice di pesi B J × . Le varianze delle Qcomponenti e i pesi sono scelti in modo tale da minimizzare la somma dei residui
(
)
2 1 1 1∑ ∑
= =−
∑
= I i J j Q q iq jq ija
b
x
per tutti i possibili valori di varianza e pesi.
Questa tecnica permette quindi di trovare quelle componenti che esprimono la massima varianza dei dati.
Per determinare le componenti principali si scompone la matrice di covarianza dei dati in una matrice di autovalori e autovettori che verificano la condizione di ortonormalità. Gli autovettori della matrice di covarianza costituiscono le colonne della matrice di rotazione T per la cui trasposta viene moltiplicata la matrice dei dati. Il risultato del prodotto è la matrice delle componenti principali Y:
X
T
Y
=
tQueste componenti hanno la proprietà di esprimere la massima varianza dei dati. Tra queste componenti vengono scelte le prime due o tre e si costruisce un piano, i cui assi rappresentano queste componenti, in cui si vanno a rappresentare i dati.
Per dare un significato al risultato dell’analisi è necessario dare un’interpretazione alle componenti e capire quali variabili possono rappresentare. Osservando quale sia la posizione delle variabili all’interno del grafico rispetto agli assi, si può determinare quanto esse siano correlate alle componenti che la PCA ha trovato.
4.3.3.2 Il modello Tucker3
La 3-way PCA ha lo stesso scopo della PCA bidimensionale cioè quello di riassumere il contenuto degli oggetti e delle variabili rispettivamente nelle matrici A e B, ma a queste viene aggiunta una terza matrice C che riassume il contenuto delle condizioni. In aggiunta a queste matrici viene determinata anche una matrice cubica, chiamata “core array” e indicata con G, che
riassume le informazioni delle tre dimensioni del data-matrix. In definitiva le matrici calcolate sono quattro:
1. A,
I
×
P
2. B,J
×
Q
3. C,K
×
R
4. G,P
×
Q
×
R
dove I sono gli oggetti, J le variabili K le condizioni e P, Q e R denotano il numero di componenti con cui vengono rappresentate le entità in ciascuno dei tre modi.
Il modello generale della 3-way PCA è il seguente:
pqr kr jq P p Q q R r jp ijk
a
b
c
g
x
∑∑∑
= = =≅
1 1 1dove sono gli elementi del data-matrix originario, , , e sono rispettivamente gli elementi delle matrici A, B, C e G.
ijk
x
a
jpb
jqc
krg
pqrLa tecnica Tucker 3, utilizzata per l’analisi con la 3-way PCA, può essere
suddivisa in 6 step principali:
1. Analisi della varianza per capire se la 3-way PCA è indicata per l’analisi dei dati considerati
2. Pre-processing dei dati
3. Bilanciamento del fitting e scelta del numero di componenti
4. Studio dettagliato del fitting e dei residui e scelta della rotazione ottimale
5. Studio della stabilità della soluzione 6. Interpretazione dei risultati.
Nella diagramma di flusso, riportato in Fig. 4.19 sono indicati i vari passaggi dell’analisi.
Di seguito viene descritto brevemente che cosa viene eseguito in ciascuno step.
¾ Step 1: Analisi della varianza
Prima di iniziare l’analisi viene effettuato un semplice test ANOVA per capire se la tecnica della 3-way PCA è utile per la analisi dei dati in ingresso. Se tutta l’informazione è catturata all’interno di soli due dei tre modi, non ha molto senso analizzare i dati in tre dimensioni ed è sufficiente un’analisi con la PCA.
¾ Step 2: Pre-processing dei dati
Per facilitare l’analisi i dati devono essere sottoposti ad un pre-processing che
ha lo scopo di centrare i dati e di normalizzarli.
Tale pre-processing può essere eseguito in vari modi. Nella tecnica Tucker3
viene fatto quello che viene chiamato “j-scaling” dei dati: dato che spesso le
variabili hanno scale e unità di misura diverse, sono presenti degli offset che possono essere eliminati centrando i dati rispetto alle variabili.
¾ Step 3: Bilanciamento del fitting e scelta del numero di componenti La scelta di quante componenti utilizzare si basa su tre criteri:
1. Bilanciamento del fitting 2. Interpretabilità
In base a questi criteri, tra le infinite alternative possibili, viene scelto il modello migliore.
I fitting possibili vengono valutati e confrontati e trovando un compromesso con la necessità di mantenere una complessità del modello non troppo elevata. Il numero delle componenti viene scelto sulla base dello “scree test” grazie al
quale si può determinare il numero minimo di componenti che consente di ottenere un buon fitting dei dati.
¾ Step 4: Studio del fitting e dei residui e scelta della rotazione
Per valutare se il fitting è stato scelto adeguatamente esso viene partizionato per ciascuna entità di ciascuno dei tre modi del data-matrix. Se alcune delle entità non vengono fittate bene è possibile che la modellizazione dei dati sia incompleta. Per valutare la completezza del modello si possano studiare i residui che rappresentano quegli aspetti dei dati che non sono rappresentati bene dal modello. Se tali residui hanno valori elevati, il modello risulta incompleto e si può decidere di aggiungere altre componenti sempre trovando un compromesso con il cercare di mantenere una certa semplicità del modello. Come è già stato detto precedentemente, la soluzione del modello non è unica: si possono trovare descrizioni dei dati equivalenti ruotando la matrici A, B e C. Il passo successivo consiste quindi nell’andare a valutare per quale rotazione si ottiene la soluzione più facile da interpretare.
Tale scelta viene fatta eseguendo una serie di rotazioni ortogonali delle matrici e andando a vedere per quale orientamento comune delle tre matrici il
“core array” è il più diagonale possibile.
Nel caso in cui non si riesca a trovare una soluzione facile da interpretare, è necessario modificare il numero di componenti scelto.
¾ Step 5: Studio della stabilità
Dopo aver ottenuto una soluzione che fitta bene i dati e che è facile da interpretare, l’ultima cosa da fare è andare a vedere se tale soluzione è stabile per fluttuazioni dei campioni. La stabilità è collegata alla generalizzabilità: la soluzione scelta deve andare bene anche per campioni della stessa popolazione ma diversi da quelli su cui è stata effettuata l’analisi. La stabilità e la generalizzabilità sono caratteristiche molto importanti per cui se la soluzione scelta non ha queste proprietà, è necessario rivalutare la scelta del numero di componenti e della rotazione.
¾ Intermezzo
La scelta della rotazione, per determinare la soluzione più facile da interpretare, può non essere semplice senza un’indicazione sulla stabilità della soluzione. Per questo tra lo step 3 e lo step 4 è utile eseguire uno step intermedio in cui viene effettuato un controllo preliminare sulla stabilità della soluzione: la soluzione viene ruotata e viene valutata la stabilità di questa nuova soluzione. Se questa soluzione è stabile lo sarà anche quella ottenuta dopo la rotazione dato che essa avrà una struttura più raffinata.
In caso di esito negativo di questo controllo deve essere determinata una nuova soluzione scegliendo un numero diverso di componenti.
¾ Step 6: Interpretazione dei risultati
Una volta scelta la soluzione che meglio rappresenta i dati, è possibile interpretare i risultati dell’analisi.
4.4 Analisi delle reti di neuroni mesencefalici
4.4.1 Organizzazione degli algoritmi nella GUI
Dati i vantaggi ottenuti analizzando le immagini tramite una GUI, si è pensato di organizzare gli algoritmi che erano stati implementati per l’analisi delle reti, all’interno di una GUI di cui viene riportata la struttura (Fig. 4.20).
Figura 4.20 GUI per l’analisi delle reti di neuroni mesencefalici
Viene analizzata anche in questo caso la funzione di ciascun pannello.
4.4.1.1 Caricamento dell’immagine
Nel pannello relativo al caricamento dell’immagine viene indicato nell’edit il
numero della rete che si desidera analizzare dopo di che, premendo il bottone “Carica” negli assi viene visualizzata la fotografia effettuata il primo giorno di coltura alla rete. Una volta completata l’analisi viene caricata l’immagine successiva e così via fino all’ultimo giorno di coltura (Fig. 4.21).
Figura 4.21 Pannello per il caricamento e la visualizzazione delle immagini
4.4.1.2 Individuazione dei nodi
I nodi vengono individuati cliccandoci sopra con il tasto sinistro del mouse e per ognuno vengono mostrati il numero che lo identifica e le coordinate spaziali. Il clic destro del mouse identifica l’ultimo punto. Questa operazione viene eseguita cliccando sul bottone “Rilevamento nodi” nel pannello rappresentato in Fig. 4.22.
Figura 4.22 Pannello per l’individuazione dei nodi della rete
Quello che compare negli assi è l’immagine in cui per ogni nodo vengono mostrati il numero che lo identifica e le coordinate spaziali (Fig 4.23).
Figura 4.23 Nodi della rete identificati
4.4.1.3 Analisi della raggiungibilità
La prima analisi che viene effettuata è l’analisi della raggiungibilità. L’algoritmo prevede innanzi tutto una serie di elaborazioni dell’immagine riassunte in Fig. 4.24.
Figura 4.24 Fasi dell’elaborazione delle immagini delle reti
Questa serie di elaborazioni viene realizzata a seguito del clic sul bottone “Livelli di grigio” prima e “Rilevamento” poi (Fig. 4.25). Ogni volta che il programma esegue un passo dell’elaborazione, negli assi viene visualizzata l’immagine ottenuta.
Figura 4.25 Pannello per l’elaborazione delle immagini
Prima di iniziare l’elaborazione è necessario indicare nell’edit il valore del fudge factor, un parametro di sogliatura che deve essere scelto
opportunamente in funzione dell’immagine.
L’immagine finale è un’immagine segmentata ottenuta racchiudendo all’interno di una stessa regione tutti i pixel 8-connessi tra loro.
A ciascun oggetto identificato viene inoltre assegnato un determinato colore (Fig. 4.26).
La matrice di raggiungibilità è una matrice quadrata di dimensione pari al numero dei nodi (N), il cui generico elemento rij=1 se esiste almeno un percorso, senza condizioni sulla sua lunghezza, da i a j, altrimenti rij=0.
In questo caso, per costruire la matrice di raggiungibilità, viene analizzata l’immagine segmentata: due nodi sono raggiungibili se appartengono allo stesso oggetto, ovvero se i pixel che li rappresentano hanno gli stessi valori di R, G, B.
Per ogni nodo i viene realizzato un confronto tra i suoi valori di R, G, B e
quelli di ogni altro generico nodo j e, se tali valori sono uguali, viene messo 1 nella posizione i,j della matrice di raggiungibilità, diversamente 0.
Una volta realizzata la matrice di raggiungibilità viene disegnato il grafo di raggiungibilità, in cui esiste una freccia tra il nodo i e il nodo j se l’elemento i,j della matrice è pari a 1.
L’analisi della raggiungibilità viene realizzata cliccando sul bottone “Matrice di raggiungibilità”: nella list-box viene visualizzata la matrice ottenuta ed in una finestra viene rappresentato il grafo (Fig. 4.27).
Figura 4.27 Pannello per il calcolo della matrice di raggiungibilità (sinistra) e grafo di
4.4.1.4 Costruzione del grafo e analisi della connettività
Dati i nodi della rete, viene costruita la matrice delle adiacenze e quindi il grafo (Fig. 4.28). Anche la matrice delle adiacenze, così come la matrice di raggiungibilità, è una matrice quadrata di dimensione pari al numero dei nodi
(N), in cui però il generico elemento aij=1 se e i e j, sono direttamente
connessi tra loro.
Una volta determinati i nodi, nella list-box compare una matrice di zeri di
dimensione pari al numero di nodi; guardando l’immagine poi vengono sostituiti degli 1 nella posizione i,j se i nodi i e j sono adiacenti.
Dalla matrice delle adiacenze è inoltre ricavato un vettore contenente il grado di ogni nodo, ovvero il numero totale di connessioni con altri nodi, ottenuto sommando gli elementi della i-esima colonna (o riga) della matrice delle
adiacenze, e la connettività media ottenuta come media aritmetica delle connettività di ogni nodo. Entrambi sono visualizzati negli edit corrispondenti.
Figura 4.28 Pannello per il calcolo della matrice delle adiacenze (sinistra) e grafo di
4.4.1.5 Lunghezze del cammino
Si definisce distanza tra 2 nodi i e j (dij) o lunghezza del camminoil numero di
archi del più breve cammino che li connette. Le lunghezze del cammino
vengono calcolate a partire dalla matrice delle adiacenze: l’elemento i,j della matrice Al è il numero di cammini di lunghezza l dal nodo i al nodo j.
La lunghezza del grafo o lunghezza media di un cammino (L) è la media delle
distanze tra coppie di nodi ottenuta come media aritmetica su tutte le coppie di nodi.
Il vettore delle lunghezze del cammino e il valore della lunghezza media, vengono visualizzati negli edit corrispondenti rispettivamente al clic del mouse sui bottone “Distribuzione” e “Lunghezza media” (Fig. 4.29).
Figura 4.29 Pannello per il calcolo delle lunghezze medie del cammino per ciascun nodo e
della lunghezza media del cammino di tutta la rete
4.4.1.6 Studio delle distanze
Dopo aver individuato i vari nodi, viene calcolata la distanza euclidea tra di essi. Viene costruita una matrice quadrata di dimensione pari al numero dei nodi e l’elemento i,j di tale matrice rappresenta la distanza tra il nodo i e il
nodo j.
La matrice viene visualizzata nella list-box del pannello riportato in Fig. 4.30
Figura 4.30 Pannello per il calcolo della matrice delle distanze
4.4.1.7 Coefficiente di clustering
Il coefficiente di clustering del grafo è calcolato come:
∑
==
N j jN
C
G
C
(
)
1dove Cj è il coefficiente di clustering del noto i, dato da:
2
/
)
1
(
−
=
i i j ik
k
E
C
ki sono gli archi del nodo i, che lo connettono ad altrettanti nodi. Questi nodi
sono i vicini del nodo i.
ki (ki-1)/2 è il numero massimo di archi che esistono tra i vicini del nodo i,
quando ogni vicino del nodo i è connesso ad ogni altro vicino del nodo i. Ei rappresenta il numero effettivo di archi esistente tra i vicini del nodo i.
Il coefficiente di clustering così calcolato viene visualizzato nell’edit
Figura 4.31 Pannello per il calcolo del coefficiente di clustering
4.4.1.8 Test small-world
Per poter capire se una data rete neuronale in vitro appartiene alla classe degli
small-worlds, sono stati calcolati il coefficiente di clustering e la lunghezza media di un cammino, così da poter effettuare il cosiddetto “test small-world” [87].
Per poter classificare una rete come small-world essa deve soddisfare due condizioni:
1. il coefficiente di clustering della rete deve essere maggiore di quello di
un grafo random con lo stesso numero di nodi e la stessa connettività media, ovvero la rete deve avere un’elevata tendenza a formare gruppi locali molto ben connessi
2. la lunghezza media di un cammino deve essere minore di quella di un grafo regolare con lo stesso numero di nodi e la stessa connettività media e maggiore di quella di un grafo random sempre con lo stesso numero di nodi e la stessa connettività media.
Dopo aver calcolato il coefficiente di clustering e la lunghezza media del
cammino sia del grafo random che del grafo regolare, i risultati vengono visualizzati negli edit (Fig. 4.32) e compare un messaggio in cui viene
Figura 4.32 Pannello per l’esecuzione del test small world
4.4.1.9 Memorizzazione dei dati
Anche in questo caso, così come nella GUI realizzata per l’analisi delle cellule di Purkinje, ogni volta che viene eseguita una callback, le variabili
calcolate vengono inserite in una posizione specifica del data-matrix inizializzato all’inizio del programma. La struttura finale del data-matrix è quella riportata nella tabella seguente (Tab. 4.2).
k kmedia Distanze Lunghezze Lmedia
1 ÷ 10 11 12 ÷ 111 112 ÷ 156 157
C Crand Lrand Lreg SW n
158 159 160 161 162 163
Tabella 4.2 Data-matrix contenente le variabili estratte dall’analisi delle reti (k: grado,
kmedia: connettività media, Distanze: matrice delle distanze, Lunghezze: lunghezze medie del cammino di ciascun nodo, Lmedia: lunghezza media del cammino della rete, C: coefficiente di clustering, Crand: coefficiente di clustering di un grafo random, Lrand: lunghezza media del cammino di un grafo random, Lreg: lunghezza media del cammino di un grafo regolare, SW: risultato del test small-world, n: numero di nodi
Ancora una volta unendo i data-matrix relativi alle singole reti si ottiene un data-matrix globale da cui estrarre tutte le informazioni di interesse.
4.4.2 Analisi delle immagini in fluorescenza
4.4.2.1 Elaborazione delle immagini
Per poter analizzare l’organizzazione della rete è necessario estrarre la sua sagoma dall’immagine. Nell’immagine infatti sono presenti anche detriti e cellule gliali che non devono essere considerati nell’analisi.
La procedura di elaborazione si articola in diverse fasi:
1. Selezione dell’intervallo: Per capire quali valori assume la sagoma di interesse, l’immagine viene innanzi tutto convertita in double (Fig. 4.33) e viene costruito il suo istogramma. Osservando l’istogramma si capisce in quale intervallo di valori è racchiusa la sagoma per cui viene visualizzata solamente quella parte dell’immagine (Fig. 4.34).
Figura 4.34 Selezione dell’intervallo in base all’istogramma dell’immagine
2. Dilatazione: L’immagine ottenuta è dilatata per delineare meglio i contorni e ripristinarne la continuità, eliminando eventuali gaps (Fig.
4.35). Si sono utilizzati elementi strutturali verticali seguiti da elementi strutturali orizzontali.
Figura 4.35 Immagine dilatata
3. Divisione in regioni: In base alla connettività dei vari pixel, l’immagine è divisa in regioni o oggetti: tutti i pixel 8-connessi tra loro appartengono ad una stessa regione. A ciascun oggetto identificato viene inoltre assegnato un determinato colore. Viene quindi selezionata e mostrata la sagoma di interesse (Fig. 4.36).
Figura 4.36 Selezione della sagoma di interesse dall’immagine segmentata
4. Erosione: La sagoma viene rifinita erodendo l’immagine 2 volte. L’erosione ripristina le dimensioni originali degli oggetti, mantenendo uniti quelli molto vicini (Fig. 4.37).
Figura 4.37 Erosione della sagoma
4.4.2.2 Analisi frattale
L’analisi frattale è stata realizzata sulle immagini elaborate utilizzando gli stessi algoritmi che sono stati applicati anche alle immagini delle cellule di Purkinje. Anche in questo caso lo scopo dell’analisi è stato quello di comprendere quale sia il livello di organizzazione ma in questo caso si tratta di un’organizzazione a livello globale. Si ottiene quindi come risultato dell’analisi la dimensione frattale di tutta la rete che è una misura di quanto la rete sia organizzata e regolare.
4.4.2.3 Analisi di clustering
L’analisi di clustering può essere applicata sia a dati che ad immagini e
permette di individuare dei raggruppamenti all’interno dei dati in ingresso. Nel caso in cui l’analisi venga realizzata su immagini, come in questo caso, i
dati in ingresso sono i valori dei pixel da cui è composta l’immagine. Esistono diverse tecniche di clustering, quella utilizzata per l’analisi delle immagini delle reti di neuroni mesencefalici è l’analisi gerarchica [88]. Il nome deriva dal fatto che viene costruito un albero gerarchico in cui i cluster ad un certo
livello sono uniti tra loro come i cluster a livello più alto.
I comandi utilizzati per l’implementazione dell’algoritmo che esegue il
clustering, sono quelli che si trovano nello “Statistics Toolbox” di Matlab.
La procedura si articola in tre step:
1. Trovare somiglianze e disuguaglianze tra ciascun paio di oggetti della matrice in ingresso
2. Raggruppamento degli oggetti in un albero di clustering binario e
gerarchico
3. Determinare dove tagliare l’albero gerarchico
Vengono di seguito descritti i tre step specificando il comando di Matlab che è stato utilizzato.
¾ Step1: Distanza tra i dati in ingresso
La somiglianza e la disuguaglianza tra i dati in ingresso vengono valutate calcolando la distanza tra tutte le coppie i dati in ingresso. La funzione Matlab che esegue questo comando è la funzione pdist che restituisce in uscita un
vettore in cui ciascun elemento contiene la distanza tra coppie di oggetti. È possibile utilizzare diversi metodi di misurazione delle distanze. In questo caso è stato visto che il tipo di misura che funziona meglio è la distanza euclidea (Fig. 4.38).
Figura 4.38 Valutazione della distanza tra gli oggetti
¾ Step2: Raggruppamento degli oggetti e costruzione dell’albero gerarchico Per determinare dei raggruppamenti tra gli oggetti in ingresso, vengono comparati tutti i valori contenuti nel vettore delle distanze. Coppie di oggetti vicini tra loro vengono messi all’interno di un unico cluster. I cluster che
vengono costituiti sono poi confrontati tra loro e raggruppati in cluster più
grandi finché si genera un albero gerarchico. Il clustering viene realizzato dalla funzione linkage che restituisce in uscita una matrice in cui le prime due
colonne rappresentano gli oggetti o i cluster confrontati e la terza la loro
distanza (Fig. 4.39). Anche in questo caso è possibile utilizzare diversi criteri per la costruzione di cluster. Quello che ha dato i migliori risultati è stato il
Figura 4.39 Raggruppamento degli oggetti in cluster
L’albero gerarchico, chiamato anche dendogramma, può essere poi graficato tramite la funzione dendogram (Fig. 4.40).
Figura 4.40 Esempio di dendogramma
I numeri lungo l’asse orizzontale rappresentano gli indici degli oggetti in ingesso nel caso in cui essi siano meno di 30, oppure nel caso in cui siano di più, come nel caso dei pixel delle immagini, il dendogramma collassa verso il numero minimo di componenti necessario. I link tra gli oggetti sono
rappresentati tramite U rovesciate la cui altezza indica la distanza tra gli oggetti.
¾ Step3: Determinazione dell’altezza del taglio
Per individuare il numero di cluster presenti nella rete è necessario eseguire
un taglio orizzontale del dendogramma e contare quante volte questo viene tagliato (Fig. 4.41). La determinazione dell’altezza del taglio dipenda dalla scala a cui si desidera osservare le correlazione tra i dati.
L’operazione di taglio e la determinazione del numero di cluster sono
eseguite dalla funzione cluster.