1.1 LE 5'-NUCLEOTIDASI
Il metabolismo dei nucleotidi purinici e pirimidinici comprende varie vie biosintetiche e cataboliche che assicurano una concentrazione di nucleosidi trifosfato sufficiente a sostenere i processi vitali della cellula. Specifiche reazioni di fosforilazione e defosforilazione, catalizzate rispettivamente da 5'-nucleotidasi e da chinasi nucleosidiche, creano un equilibrio dinamico che consente un rapido aumento o una rapida riduzione della concentrazione di particolari nucleotidi in risposta a variazioni delle condizioni metaboliche [Bianchi et al., 1986].
Le 5'-nucleotidasi, i componenti catabolici di questi “substrate cycles”, sono una famiglia di enzimi che, catalizzando la defosforilazione di ribo- e deossiribonucleosidi monofosfato, mantengono bilanciati i pools dei ribo- e
deossiribonucleotidi. L'attività delle 5'-nucleotidasi è stata descritta in vari tipi cellulari e in specie filogeneticamente molto diverse (batteri, piante e vertebrati), indice dell'importante ruolo che questi enzimi rivestono nel metabolismo.
Figura 1-A: “SUBSTRATE CYCLE” GENERICO. .
(da Rampazzo et al., 2000b)
Nell'uomo sono state isolate e caratterizzate sette 5'-nucleotidasi con diversa localizzazione subcellulare: una ancorata alla superficie esterna della membrana plasmatica, una localizzata nella matrice mitocondriale e cinque nel citosol. Questi enzimi variano sia nella specificità di substrato che nella distribuzione tissutale; infatti, mentre alcuni sono ubiquitari, altri mostrano un'espressione tessuto-specifica [Bianchi and Spychala, 2003].
Le nucleotidasi intracellulari, molto sensibili alle oscillazioni della concentrazione del substrato, generano nucleosidi che possono essere trasportati all'esterno della cellula, mentre la 5'-nucleotidasi extracellulare, componente di una cascata ecto-nucleotidasica, partecipa alla regolazione dei nucleotidi extracellulari. I nucleosidi prodotti all'esterno
della cellula possono essere internalizzati mediante trasportatori e riutilizzati dalla cellula stessa [Hunsucker et al., 2005].
Bianchi e Spychala hanno recentemente proposto una nomenclatura più semplice per la classificazione di questa famiglia di enzimi: il termine “nucleotidasi” viene indicato con la lettera “N” preceduta dalla lettera minuscola “e”, “c” o “m”, a seconda che si tratti della forma ectoplasmatica, citosolica o mitocondriale, e seguita da un numero romano “I”, “II”, etc. se più forme occupano lo stesso compartimento cellulare; inoltre può essere aggiunto il suffisso “A”, “B”, etc., per distinguere più forme strettamente correlate e con la stessa localizzazione subcellulare [Bianchi and Spychala, 2003].
5 5’’--NNuucclleeoottiiddaassii AAlliiaass SSttrruuttttuurraa DDiissttrriibbuuzziioonnee t tiissssuuttaallee SSuubbssttrraattii Ecto-5’-nucleotidasi (eN) Ecto 5’-NT; NT5; eNT; low Km 5’-NT; CD73
Dimero Ubiquitaria Deossi- e ribonucleosidi monofosfato purinici e pirimidinici 5’-Nucleotidasi citosolica IA (cN-IA) specific AMP 5’-NT Tetramero Muscolo scheletrico, miocardio, encefalo, rene, pancreas AMP, dTMP, dCMP, dGMP, dAMP, dIMP 5’-Nucleotidasi citosolica
IB (cN-IB) homolog, AIRP cN-IA ? Testicoli AMP 5’-Nucleotidasi citosolica II (cN-II) High Km 5’-NT; 5’-purinic NT; specific GMP-IMP NT Tetramero Ubiquitaria
Attività nucleotidasica: IMP, dIMP, GMP, dGMP, XMP;
Attività PHT: Ino, dIno
5’-Nucleotidasi citosolica III (cN-III)
PN-I; P5’-NT, UMPH,
UMPH1 Monomero Eritrociti
Attività nucleotidasica: CMP, UMP, dUMP, dCMP, dTMP; Attività PHT: uridina, citidina e deossicitidina 5’(3’)-deossiribonucleotidasi citosolica (cd-N) dNT-1; PN-II;
UMPH-2 Dimero Ubiquitaria
dUMP, dTMP, 3’-UMP e 2’-3’-UMP; una più bassa attività con
5’-dIMP, 5’-dUMP, 5’-dCMP, 5’-dTMP e 5’dAMP
5’(3’)-deossiribonucleotidasi
mitocondriale (md-N) dNT-II Dimero Ubiquitaria
5’-dUMP; 5’-dTMP; 3’-dTMP; 5’-,3’-,2’-UMP
1.1.1 LA ECTO-5'-NUCLEOTIDASI (eN)
La ecto-5'-nucleotidasi è una proteina glicosilata ancorata alla superficie esterna della membrana plasmatica tramite il legame tra la serina 523 C-terminale, altamente conservata, e un glicosilfosfatidilinositolo (GPI) di membrana [Ogata et al., 1990]. La proteina attiva è un dimero le cui subunità, unite da legami non covalenti [Martinez-Martinez et al., 2000], hanno un peso molecolare apparente che varia tra 60 e 80 kDa, a seconda del tessuto e della specie da cui l'enzima viene isolato.
L'eN ha una ampia specificità di substrato e può idrolizzare vari ribo- e deossiribonucleosidi 5'-monofosfato, ma l'AMP rappresenta il nucleotide idrolizzato con maggior efficienza; i valori della km per il 5'-AMP sono, infatti, nell'ordine del micromolare e il riconoscimento del substrato è stereoselettivo, perché è stato osservato che l’enantiomero levogiro (L-enantiomero) non viene idrolizzato [Zimmermann H., 1992]. Dalla purificazione dell’eN dallo stomaco di pollo è risultato sempre presente il catione metallico Zn2+ con un rapporto di 2 ioni per ogni molecola di enzima [Zimmermann H., 1992]; ciò ha fatto pensare che la ecto-5'-nucleotidasi potesse essere un metallo-enzima. Tale supposizione è stata confermata in seguito alla constatazione che l’utilizzo di chelanti di cationi bivalenti, come l'EDTA, inibisce l’attività enzimatica [Zimmermann H., 1996].
Lo studio della struttura cristallina della 5'-nucleotidasi periplasmatica di Escherichia coli, un omologo di eN, ha permesso di comprendere il meccanismo catalitico della ecto-5'-nucleotidasi umana [Knofel & Strater, 1999, 2001]. L'attività catalitica inizia con l’attacco nucleofilo al gruppo fosfato del substrato da parte di una molecola d'acqua legata ad uno dei due ioni Zn2+; dopo la formazione dell'intermedio fosforilato dell'enzima, si ha il rilascio dell'ortofosfato. Sebbene questi due enzimi abbiano una bassa omologia di sequenza, i residui del sito catalitico, implicati nel legame dei due cationi bivalenti Zn2+, sono conservati nei motivi N-terminale A e B [Knofel & Strater, 2001]. Nella eN umana se l'istidina 92 (His92), appartenente al motivo A, o l'istidina 194 (His194), appartenente al motivo B, vengono mutate in alanina si osserva la perdita dell'attività nucleotidasica [Gutensohn et al., 1995]. E' stato infatti dimostrato che l'His92 gioca un ruolo importante nello stabilizzare il gruppo fosfato del substrato, mentre l'His194 coordina uno dei due ioni Zn2+ presenti nel sito catalitico [Knofel & Strater, 2001].
L'enzima è inibito in modo competitivo da ATP, ADP e dall’analogo nucleotidico adenosina-[α,β-metilene]difosfato (AMPCP), e in modo non competitivo dalle lectine, come la concanavalina A; la massima attività enzimatica si ottiene a pH lievemente alcalini (tra 7 e 8).
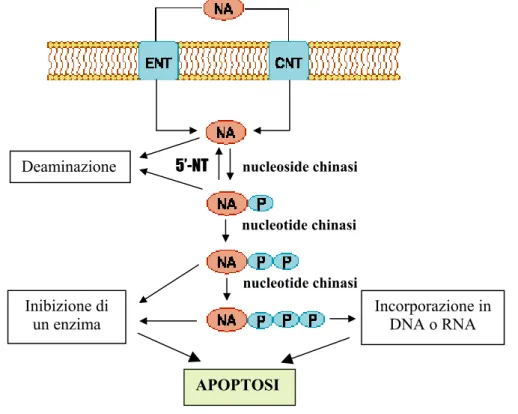
La ecto-5'-nucleotidasi è coinvolta nell'ultimo passaggio della cascata enzimatica che porta alla formazione dei nucleosidi nei distretti extracellulari. Data l'alta affinità per l'AMP, la sua funzione è quella di produrre principalmente adenosina, che può agire localmente attivando i recettori accoppiati a proteine G; questi mediano risposte come il precondizionamento ischemico del miocardio, la vasodilatazione, i fenomeni angiogenetici e l'inibizione della risposta immunitaria. L'attività nucleotidasica dell'eN gioca un ruolo fondamentale anche nel recupero dei nucleotidi, che per poter essere ricaptati dall’ambiente esterno, devono venir defosforilati a nucleosidi (figura 1-B) [Hunsucker et al., 2005].
Figura 1-B: RAPPRESENTAZIONE DELLE PRINCIPALI FUNZIONI DELL’ECTO-5’-NUCLEOTIDASI.
(modificato da Hunsucker et al., 2005)
L'eN è ubiquitaria ma in alcuni tipi cellulari, durante i fenomeni di migrazione e differenziazione cellulare, si osserva una variazione della sua espressione. Questo comportamento dinamico è stato particolarmente studiato sui linfociti. Gli studi condotti su tali cellule hanno dimostrato che l'espressione della nucleotidasi (chiamata anche CD73) è legata allo stadio di maturazione della cellula, perché i linfociti periferici mostrano una attività enzimatica maggiore rispetto ai linfociti del timo e del midollo osseo [Zimmermann H., 1992].
Questo enzima è stato ampiamente studiato anche a livello del sistema nervoso, dove sembra che la ecto-5’-nucleotidasi possa esercitare un effetto trofico sull’estensione del neurite. In culture di cellule nervose il blocco dell’attività dell’eN, ottenuto con l’aggiunta di inibitori competitivi, o l’inibizione della sintesi dell’enzima, ottenuta con l’applicazione di oligonucleotidi antisenso, provoca infatti una riduzione della crescita del neurite indotta dall’NGF (fattore di accrescimento nervoso) [Zimmermann H., 1996].
L'ecto-5'-nucleotidasi è implicata anche in funzioni che non richiedono una attività catalitica, come l'adesione cellulare. Le proprietà adesive di questa proteina sono utilizzate, ad esempio, dai linfociti per aderire all'endotelio vascolare e poter migrare verso il sito della flogosi [Airas et al., 1995]. Nei linfociti T l'eN può agire anche come co-recettore e
partecipare al processo di attivazione di queste cellule [Resta et al.,1998].
È stata isolata anche una forma solubile dell'ecto-5'-nucleotidasi, chiamata eNs; i due enzimi mostrano le stesse caratteristiche cinetiche e sono riconosciuti dagli stessi anticorpi monoclonali, perciò si ritiene che la forma solubile derivi dalla proteina ancorata alla membrana e sia generata dall'idrolisi del legame tra l’enzima e la molecola di GPI ad opera di fosfolipasi endogene [Zimmermann H., 1992].
1.1.2 LE 5'-NUCLEOTIDASI CITOSOLICHE IA E IB (cN-IA, cN-IB)
La cN-IA è un enzima capace di idrolizzare una vasta gamma di substrati nucleotidici, ma ha una maggiore affinità per l'AMP e i deossiribonucleosidi monofosfato dTMP e dCMP. Il gene della cN-IA è localizzato sul cromosoma 1 e la sua espressione è massima nel muscolo scheletrico, nel miocardio e nell’encefalo [Hunsucker et al., 2001]. La proteina, purificata da tessuti provenienti da diversi vertebrati (piccione, topo, coniglio e uomo), è un omotetramero formato da subunità di circa 41 kDa [Sala-Newby et al., 1999; Hunsucker et al., 2001]. Confrontando la sequenza aminoacidica della cN-IA umana con quella di altri vertebrati si può osservare che sia i residui aminoacidici dell’estremità N-terminale che quelli del C-N-terminale mostrano una elevata divergenza, mentre la sequenza tra il residuo aminoacidico 46 e 351 presenta una omologia del 90%-99% e rappresenta la regione contenente i motivi che formano il sito catalitico [Sala-Newby & Newby, 2001].
L’attività enzimatica della cN-IA è modulata allostericamente da molecole effettrici come l’ADP e, in modo minore, il GTP; inoltre, per l’attività catalitica l’enzima richiede la presenza del catione bivalente Mg2+ e un pH lievemente acido (circa 6.5) [Skladanowski & Newby, 1990; Hunsucker et al., 2001]. Il principale ruolo di questa nucleotidasi è la formazione dell’adenosina a partire dall’AMP durante periodi di ipossia e di ischemia, situazioni che creano le condizioni ottimali per l’attività catalitica. La reazione mediata dalla cN-IA è alla base della regolazione dell’omeostasi cellulare dell’ATP; l’adenosina, prodotta quando la velocità di degradazione dell’ATP supera quella di formazione, viene trasportata nello spazio extracellulare dai carrier e, legandosi a specifici recettori, innesca una serie di risposte cellulari volte a ripristinare il corretto livello energetico [Newby, 1984; Newby et al., 1990]. Nel miocardio, ad esempio, l’adenosina, per contrastare gli effetti dell’ischemia, induce la vasodilatazione delle arterie coronarie, riduce la forza di contrazione del cuore, rallenta il ritmo cardiaco e la conduzione atrio-ventricolare; inoltre questo nucleoside contribuisce al precondizionamento ischemico, un breve periodo di
ischemia che protegge il cuore da un successivo e più lungo periodo di carenza di nutrienti e ossigeno [Schrader, 1990; Berne, 1964; Ralevic and Burnstock, 1998; Yokota et al., 1995; Cohen et al., 1995].
Durante l’ischemia l’aumentata produzione di adenosina è, in realtà, legata sia all’inibizione dell’adenosina chinasi che all’incremento dell’attività della cN-IA stimolata da un’alta concentrazione del substrato (AMP) e dell’attivatore (ADP) [Yamazaki et al., 1991]. A concentrazioni normali di ossigeno (normossia) il rapporto tra l’attività dei due enzimi favorisce invece la produzione dell’AMP e, poiché la cN-IA mostra bassi valori di km per i deossiribonucleotidi monofosfato, si pensa che l’enzima, in condizioni fisiologiche, possa giocare un importante ruolo nella regolazione del pool dei deossinucleotidi pirimidinici cellulari [Hunsucker et al., 2005].
Questa nucleotidasi è strettamente correlata ad un altro enzima recentemente scoperto, la cN-IB, con la quale condivide la capacità di idrolizzare l'AMP e l’effetto attivante esercitato dall’ADP. Il gene della cN-IB è localizzato sul cromosoma 2 e la proteina è espressa prevalentemente nei testicoli, dove è stata identificata come fattore antigenico in una malattia autoimmune umana che causa sterilità [Sala-Newby & Newby, 2001]. Purtroppo i dati fino ad oggi disponibili riguardo questo enzima non consentono di conoscere né la completa specificità di substrato, né la struttura dell’enzima.
1.1.3 LA 5’-NUCLEOTIDASI CITOSOLICA II (cN-II)
La cN-II, oggetto del presente studio, sarà discussa dettagliatamente nel prossimo capitolo.
1.1.4 LA 5’-NUCLEOTIDASI CITOSOLICA III (cN-III)
Questa nucleotidasi è stata oggetto di importanti studi che hanno dimostrato una correlazione positiva tra il gene della cN-III e lo sviluppo di anemia emolitica. La sequenza genica che codifica per questo enzima è localizzata sul cromosoma 7 e contiene 11 esoni [Marinaki et al., 2001; Kanno et al., 2004]. Il trascritto primario, in seguito a splicing alternativo dell’esone 2 e/o dell’esone R (recentemente identificato e localizzato tra l’esone 2 e 3), dà origine a tre mRNA di diversa lunghezza e, tra questi, quello più corto codifica per una nucleotidasi attiva di 286 aminoacidi [Kanno et al., 2004; Marinaki et al., 2001]. Questa proteina è stata clonata e studiata dettagliatamente e le caratteristiche dell’enzima
sono risultate le stesse della cN-III purificata [Rees et al., 2003]. Questa nucleotidasi è l’unico membro della famiglia ad agire come monomero [Amici et al., 1997; Marinaki et al., 2001].
La cN-III idrolizza esclusivamente nucleotidi pirimidinici (per questo è stata chiamata anche pN-I) e il suo substrato migliore è il CMP seguito dall’UMP. Il pH ottimale per la reazione è di 7.5 e, come le altre nucleotidasi citosoliche, è dipendente dal catione bivalente Mg2+ per l’attività catalitica [Amici & Magni, 2002]. Questo enzima ha anche la capacità di trasferire il gruppo fosfato dal substrato ad un nucleoside accettore, come l’uridina e la citidina [Amici et al., 1997], ma ancora non è stato chiarito se l’attività fosfotransferasica, meno efficiente di quella mostrata dalla 5’-nucleotidasi citosolica II [Pesi et al., 1994], possa svolgere un ruolo anche in vivo.
Nonostante la cN-III sia presente in vari tessuti, è stata studiata prevalentemente negli eritrociti dove la sua concentrazione è massima. Durante la maturazione dei globuli rossi, un processo accompagnato da importanti modificazioni morfologiche e biochimiche, la cN-III è implicata nell’idrolisi dei ribonucleotidi pirimidinici derivati dalla degradazione dell’RNA. Il ruolo svolto dalla nucleotidasi in questo tipo di cellule è di fondamentale importanza, perché individui omozigoti per una mutazione del gene della cN-III sono affetti da anemia emolitica, una patologia caratterizzata da una prematura lisi delle emazie e dall’accumulo negli eritrociti di grandi quantità di citidina e uridina monofosfato [Valentine et al., 1974; Rees et al., 2003].
Il sequenziamento parziale dell’enzima e la conseguente clonazione del gene hanno rivelato che la cN-III è identica alla p36, una proteina indotta dall’interferon α (IFNα) e legata alla formazione di corpi di inclusione nelle cellule endoteliali e nelle cellule mononucleate del sangue periferico di pazienti affetti da una malattia autoimmune, il lupus eritematoso sistemico, e dalla sindrome dell’immunodeficienza acquisita (AIDS) [Amici et al., 2000].
1.1.5 LA 5’(3’)-DEOSSIRIBONUCLEOTIDASI CITOSOLICA (cd-N)
La cd-N umana, che è stata purificata per la prima volta dalla placenta, presenta una distribuzione tissutale ubiquitaria, ma con una maggiore espressione nel pancreas, nel muscolo scheletrico e nel miocardio. La forma attiva dell’enzima è un omodimero costituito da subunità di 23.4 kDa e il suo gene, contenente 5 esoni e 4 introni, è localizzato sul cromosoma 17 [Hoglund & Reichard, 1990; Rampazzo et al., 2000a; Rampazzo et al.,
2002 ]. La nucleotidasi predilige come substrati i nucleotidi contenenti la base pirimidinica uracile o timina e mostra una elevata attività nucleotidasica con i 2’- e i 3’-nucleosidi monofosfato, senza avere una spiccata preferenza né per i ribo- né per i deossiribonucleotidi; i migliori substrati per la cd-N umana sono, infatti, il dUMP, il 3’-dTMP, il 3’-UMP e il 2’-UMP seguiti da tutti i 5’-dNMPs eccetto il dCMP che non viene defosforilato [Hoglund & Reichard, 1990].
I pools dei dNTP, utilizzati dalla cellula per la sintesi del DNA e la sua riparazione, sono sottoposti ad un continuo controllo affinché non si crei né un eccessivo aumento della loro concentrazione né uno squilibrio tra un pool e gli altri, perché tale condizione porterebbe ad una riduzione della fedeltà di replicazione del DNA e quindi ad un aumento dell’incidenza di mutazioni e aberrazioni cromosomiche. I dati ottenuti da esperimenti condotti su cellule che sovraesprimono questa nucleotidasi sembrano coinvolgere la cd-N e la timidina chinasi 1 (TK1) nella regolazione dei livelli dei deossinucleotidi, affidando alla nucleotidasi il ruolo di proteggere la cellula da un aumento dei nucleotidi pirimidinici e in particolare dai dUTP e i dTTP [Gazziola et al., 2001].
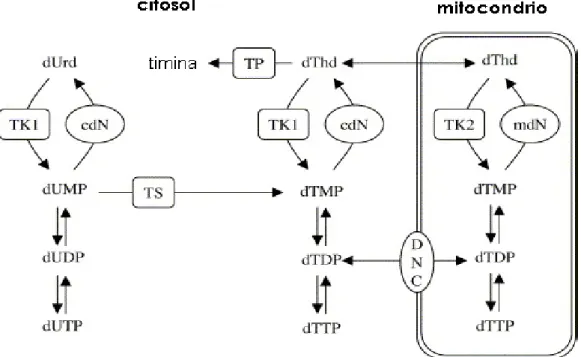
1.1.6 LA 5’(3’)-DEOSSIRIBONUCLEOTIDASI MITOCONDRIALE (md-N) La md-N è strettamente correlata ad un’altra nucleotidasi, la deossiribonucleotidasi citosolica (cd-N); dall’analisi della loro struttura primaria emerge una omologia di sequenza pari al 52% (se si esclude la sequenza leader mitocondriale della md-N ) e i loro geni, che si trovano entrambi sul cromosoma 17, mostrano la stessa organizzazione in 5 esoni e 4 introni. Queste correlazioni hanno fatto ipotizzare che i due geni possano essere il risultato di un fenomeno di duplicazione genica [Rampazzo et al., 2000b].
Pochi anni fa è stata determinata la struttura cristallina della md-N, un omodimero formato da subunità di 26 kDa in ciascuna delle quali è possibile rintracciare due diversi domini: un grande dominio simile al motivo α/β di Rossmann e un piccolo dominio costituito da quattro α-eliche. Il sito attivo dell’enzima, accessibile al solvente, è localizzato nell’interfaccia tra i due domini, dove alcuni residui aminoacidici, conservati tra le nucleotidasi intracellulari, coordinano uno ione Mg2+ richiesto per la catalisi [Rinaldo-Matthis et al., 2002].
La mdN ha una specificità di substrato più ristretta rispetto alla cd-N, perché è in grado di defosforilare solo i deossiribonucleotidi della timina e dell’uracile; pertanto si ritiene che questo enzima possa svolgere un importante ruolo nella regolazione dei pools
mitocondriali del dTTP e del dUTP, utilizzati durante la replicazione del DNA dell’organello. I dNTPs, presenti nel mitocondrio, derivano da due diverse vie: dai deossinucleotidi (dNDP) del citosol introdotti da un trasportatore nel mitocondrio o dai deossinucleosidi trasportati da un carrier nell’organello e fosforilati da una specifica chinasi mitocondriale [Rampazzo et al., 2004]. L’importanza di mantenere i pools dei dNTP entro un certo intervallo di concentrazione è dimostrata dal fatto che una elevata quantità di deossinucleotidi può causare fenomeni di mutagenesi durante la sintesi del DNA mitocondriale. Inoltre è stato osservato che il gene della md-N mappa in una regione coinvolta nella sindrome di Smith-Magenis, una grave patologia ereditaria la cui eziologia potrebbe dipendere da un alterato metabolismo nucleotidico a livello del mitocondrio [Rampazzo et al., 2000b].
In conclusione si ritiene che, così come la coppia cdN-timidina chinasi 1 regola la concentrazione dei dTTP e i dUTP nel citosol, l’enzima md-N insieme alla timidina chinasi 2 (TK2) regoli la concentrazione di questi stessi deossinucleotidi nel mitocondrio (Figura 1-C). Il ruolo della md-N è quindi quello di contrastare l’attività della TK2 defosforilando il dTMP e proteggendo il mitocondrio da un aumento della concentrazione di questo deossinucleotide [Rampazzo et al., 2004].
Figura 1-C: SCHEMA DELLA CONNESSIONE TRA I POOLS DEI DNTP CONTROLLATI DALLA 5’(3’)-DEOSSIRIBONUCLEOTIDASI CITOSOLICA (CDN) E DALLA 5’(3’)-DEOSSIRIBONUCLEOTIDASI MITOCONDRIALE (MDN). TK1: timidina chinasi 1; TK2: timidina chinasi 2; TS: timidilato sintetasi; TP: timidina fosforilasi; DNC: carrier per deossinucleotidi (modificato da Hunsucker et al., 2005).
1.2 LA 5’-NUCLEOTIDASI CITOSOLICA II (cN-II)
1.2.1 DISTRIBUZIONE TISSUTALE e SUBCELLULARE
L’esistenza di una 5’-nucleotidasi, che idrolizza preferibilmente nucleotidi 6-idrossipurinici come l’IMP e/o il GMP, è stata dimostrata per la prima volta da Itoh e coll. (1967, 1968), che hanno isolato l’enzima dal fegato di alcuni vertebrati. Negli anni successivi questa proteina è stata purificata da vari tessuti e da specie diverse e attualmente risulta che la cN-II sia presente nei mammiferi (ratto, maiale, bovino, uomo), negli uccelli (pollo, oca), nella rana, in tre specie di pesce e nell’invertebrato Artemia [Itoh 1993].
I primi tentativi volti a valutare l’attività della 5’-nucleotidasi citosolica II non fornirono buoni risultati, in quanto i metodi di dosaggio utilizzati non eliminavano l’azione delle fosfatasi non specifiche e delle altre nucleotidasi che andavano ad alterare i risultati. Questa complicazione fu superata con l’utilizzo di un antisiero specifico per la cN-II, che ha permesso di eliminare l’interferenza degli enzimi contaminanti e misurare l’attività dell’enzima mediante immunotitolazione [Itoh and Yamada, 1991].
Grazie a questa tecnica è stato possibile osservare che negli uccelli la massima attività della nucleotidasi si registra a livello del fegato, dove questa è almeno uno o due ordini di grandezza maggiore rispetto a quella degli altri tessuti e molto più alta rispetto a quella misurata nel fegato dei mammiferi. In quest’ultimi si è osservata invece una elevata attività nei testicoli e nella milza e una bassa attività nel muscolo scheletrico e negli eritrociti.
Nell’uomo il gene della cN-II è localizzato sul cromosoma 10 [Galmarini et al., 2003] e il suo mRNA è espresso in modo ubiquitario. Dall’analisi di vari tipi cellulari la più alta attività è stata riscontrata nei fibroblasti, nei granulociti e nei linfoblasti T e B, piuttosto che negli eritrociti e nei linfociti periferici [Itoh and Yamada, 1991].
In generale è stato dimostrato che questa nucleotidasi ha una attività maggiore nei tessuti che mostrano una attiva sintesi del DNA o una elevata velocità di turnover degli acidi nucleici e dei loro precursori; tale affermazione è stata confermata dal fatto che, rimuovendo una porzione di fegato (epatectomia parziale) le cellule restanti iniziano a proliferare per ricostruire il tessuto epatico perduto e in esse si è osservato un incremento dell’attività dell’enzima [Itoh 1993]. Una ulteriore conferma è giunta dagli esperimenti eseguiti su cellule prelevate dal carcinoma del colon umano, nelle quali l’attività della cN-II è risultata significativamente più alta rispetto a quella misurata nelle cellule apparentemente sane del tessuto peritumorale [Tozzi et al., 1991].
Grazie all’antisiero specifico per la nucleotidasi è stato possibile determinare anche la sua distribuzione subcellulare: dopo colorazione immunoistochimica è emerso che la cN-II è localizzata esclusivamente nella matrice citoplasmatica, in quanto il nucleo e gli altri organelli appaiono negativi alla colorazione [Itoh 1993].
1.2.2 CARATTERISTICHE STRUTTURALI
L’elettroforesi su gel di poliacrilammide in presenza di sodio dodecil solfato (elettroforesi SDS/PAGE) dell’enzima purificato da vari tessuti di vertebrati (pollo, ratto, maiale e uomo) e dall’embrione dell’invertebrato Artemia ha mostrato una banda, corrispondente alla stima della massa molecolare della subunità proteica, che varia tra 42 kDa per l’enzima purificato da Artemia [Pinto et al., 1987] e 69 kDa per quello purificato dal tessuto polmonare di maiale [Itoh and Yamada, 1990]. La tecnica della gel filtrazione, utilizzata per stimare la massa molecolare apparente della proteina, ha mostrato dei valori che variano tra 165 kDa per la nucleotidasi dell’invertebrato [Pinto et al., 1987] e 265 kDa per quella del polmone di maiale [Itoh and Yamada, 1990]. Queste analisi, eseguite sempre in presenza di inibitori delle proteasi, hanno fornito dei risultati che suggeriscono una struttura omotetramerica della 5’-nucleotidasi citosolica II [Itoh 1993]. Sebbene l’isolazione e la purificazione della cN-II da sorgenti di natura diversa abbia più volte confermato la struttura tetramerica dell’enzima, la relazione tra l’attività catalitica e il grado di oligomerizzazione della cN-II appare poco chiara e controversa. Spychala e coll., ad esempio, hanno proposto che l’ATP, il principale attivatore dell’enzima, possa indurre una associazione delle subunità, mentre il Pi, inibitore dell’enzima, sia responsabile della loro dissociazione [Spychala et al., 1999].
Esperimenti eseguiti sulla cN-II purificata dal timo di vitello hanno messo in luce la presenza di due diverse forme dell’enzima (A e B), che sull’elettroforesi SDS/PAGE migrano al peso molecolare di 59 kDa e 54 kDa. Poiché le due forme sono presenti durante tutti i passaggi della purificazione e la loro distribuzione è stabile per parecchi mesi, le due proteine potrebbero rappresentare isoenzimi della 5’-nucleotidasi citosolica. Lo studio delle loro proprietà regolatrici (cfr. paragrafo 1.2.5) ha indotto a pensare che le forme A e B possano essere generate da una modificazione post-traduzionale [Pesi et al., 1998].
In seguito all’identificazione della sequenza genica codificante per la cN-II umana [Oka et al., 1994], è stato possibile isolare il corrispondente cDNA bovino e ottenere dalla sua espressione l’enzima ricombinante, che presenta le stesse proprietà cinetiche dell’enzima
nativo. Le sequenze nucleotidiche codificanti per l’enzima bovino e per quello umano mostrano un grado di omologia di circa il 94%, mentre le sequenze aminoacidiche possiedono una omologia del 99.5%; la nucleotidasi bovina differisce infatti da quella umana solo per due sostituzioni aminoacidiche conservative (Treonina 2 al posto di una serina e Valina 335 al posto di una isoleucina) e per l’assenza di un glutammato C-terminale. Queste divergenze non comportano differenze significative né tra le proprietà cinetiche dei due enzimi, né tra la loro organizzazione strutturale; inoltre l’espressione del cDNA bovino inserito in un vettore plasmidico consente di ottenere una grande quantità di enzima su cui è possibile eseguire studi funzionali e strutturali [Allegrini et al., 1997].
L’enzima ricombinante bovino si è rivelato utile anche per dimostrare che le forme A e B della nucleotidasi, diverse per proprietà regolatorie e elettroforetiche, sono generate in vitro, e probabilmente anche in vivo, da un taglio proteolitico a livello dell’arginina 526 (R526) riconosciuta come sito bersaglio da una proteasi non ancora identificata [Allegrini et al., 2004].
1.2.3 LA cN-II E LA SUPERFAMIGLIA DELLE ALOACIDO DEALOGENASI
La superfamiglia delle aloacido dealogenasi. Nel 1994 gli studi condotti da Koonin e Tatusov svelarono l’appartenenza delle aloacido dealogenasi batteriche ad una vasta famiglia di enzimi con diversa specificità di substrato; tutte le proteine di questa superfamiglia esibivano una singolare omologia di sequenza: contenevano dei motivi aminoacidici altamente conservati coinvolti nella catalisi. La presenza di queste sequenze conservate viene ancora utilizzata per definire la appartenenza di un enzima alla superfamiglia delle aloacido dealogenasi (HAD).
Attualmente questo gruppo di enzimi è formato da più di 3000 proteine, ubiquitarie nei procarioti e negli eucarioti. All’interno di uno stesso organismo sono in genere presenti vari enzimi appartenenti a questa famiglia (nell’uomo, ad esempio, sono stati individuati circa 58 componenti) che sono coinvolti nel metabolismo primario e secondario, nel trasporto di membrana, nella traduzione del segnale e nella riparazione degli acidi nucleici. Tuttavia gran parte dei componenti di questo gruppo non hanno una funzione conosciuta [Allen and Dunaway-Mariano, 2004], perciò solo i membri della superfamiglia delle HAD con funzione nota sono stati ulteriormente divisi in 5 sottofamiglie [Lahiri et al., 2004]:
• Aloalcano dealogenasi (idrolizza il legame C-Cl)
• Fosfato monoesterasi (idrolizza il legame P-OC)
• ATPasi (idrolizza il legame P-OP)
• Fosfomutasi (taglia il legame P-OC e esegue un trasferimento intramolecolare del fosfato)
La maggior parte delle attività catalitiche conosciute sono quindi coinvolte nel trasferimento di un gruppo fosfato [Aravind and Koonin, 1998; Aravind et al., 1998] e le attività fosfoidrolasiche più diffuse sono quelle catalizzate dalle ATPasi e dalle fosfatasi, mentre quelle meno frequenti sono quelle delle fosfomutasi e della fosfoacetaldeide idrolasi [Allen and Dunaway-Mariano, 2004].
Molte fosfotrensferasi e fosfatasi utilizzano come meccanismo di reazione una catalisi nucleofila che implica la formazione di un intermedio enzima-fosfato, nel quale il gruppo fosfato, derivante dal substrato, è legato covalentemente alla catena laterale di un aminoacido (serina, istidina, cisteina oppure aspartato) localizzato nel sito attivo. Tutti gli enzimi della superfamiglia delle HAD utilizzano questa catalisi, ma sono caratterizzati dal possedere un residuo di aspartato che compie l’attacco nucleofilo al substrato formando l’intermedio enzimatico fosfo-aspartato. L’alta energia contenuta dall’intermedio può essere utilizzata per guidare il trasferimento del fosfato all’acqua, il principale accettore del gruppo fosfato [Allen and Dunaway-Mariano, 2004].
Tutte le fosfotransferasi e le P-ATPasi della superfamiglia delle HAD sono Mg2+ -dipendenti ed il cofattore è mantenuto in posizione da uno dei motivi conservati. La funzione del magnesio sembra essere quella di legare il gruppo fosfato del substrato e schermare le sue cariche negative; inoltre è stato proposto che la coordinazione del substrato effettuata dallo ione metallico possa polarizzare il legame P-O e aumentare l’elettrofilicità del fosfato facilitando l’attacco nucleofilo [Ridder and Dijkstra, 1999 ; Wang et al., 2001; Morais et al., 2000].
Nella fosfoacetaldeide idrolasi la rimozione dell’Mg2+ provoca la denaturazione dell’enzima, che potrebbe essere dovuta alla repulsione elettrostatica tra i tre aminoacidi carbossilati interessati nella coordinazione dello ione; per questo motivo è stato ipotizzato che il magnesio possa essere richiesto anche per la dimerizzazione dell’enzima [Morais et al., 2000].
I quattro motivi aminoacidici conservati. Le proteine che sono state introdotte in questa superfamiglia, benché possano svolgere funzioni molto diverse e mostrino una scarsa omologia di sequenza (< 10%), hanno in comune quattro motivi aminoacidici (I-VI) altamente conservati che, a causa del ripiegamento secondario e terziario del polipeptide, si
ritrovano vicini e formano il sito attivo. Per tale motivo tutti gli enzimi condividono la stessa struttura tridimensionale e attuano uno stesso generico meccanismo di reazione. L’allineamento delle sequenze primarie di tutti i membri della superfamiglia delle HAD permette di osservare le sequenze conservate che questi enzimi hanno in comune (Figura 1-D).
Il motivo I (DXDXT/V) contiene un residuo di aspartato, conservato in tutti i componenti della superfamiglia, e un residuo di treonina altamente conservato [Aravind et al., 1998]. Il primo aspartato è il residuo che esegue l’attacco nucleofilo, quindi svolge un ruolo indispensabile e la sua sostituzione con un qualunque altro aminoacido provoca la completa inattivazione dell’enzima [Liu et al., 1995; Collet et al., 1998; Morais et al., 2000]; la treonina invece sembra implicata nel corretto orientamento dell’aspartato nucleofilo. Anche il secondo aspartato, meno conservato, è implicato nella catalisi; la struttura cristallina della fosfoserina fosfatasi ha indicato che questo aspartato partecipa alla coordinazione dello ione Mg2+ ed essendo protonato forma un legame a idrogeno con un atomo di ossigeno del gruppo fosfato [Wang et al., 2001].
In altri enzimi la terza posizione di questo motivo è occupata da aminoacidi diversi, come la tirosina nella L-2-aloacido dealogenasi e la treonina nelle P-ATPasi. La struttura cristallina della dealogenasi e gli studi di mutagenesi sito-diretta hanno indicato che la tirosina ha la funzione di attrarre l’alogenuro uscente [Hisano et al., 1996]. Questa osservazione ha fatto supporre che nelle varie famiglie il terzo residuo di questo motivo possa svolgere la funzione di facilitare il rilascio del primo prodotto e quindi aiutare la formazione dell’intermedio. Alla luce di queste considerazioni la presenza di un aspartato nella famiglia fosfatasi/mutasi appare indispensabile e legata al fatto che, essendo un acido debole, può donare un protone al gruppo idrossilico uscente e favorire la formazione dell’intermedio; infine nella famiglia delle ATPasi la presenza della treonina è giustificata dal fatto che, in questo caso, il gruppo uscente (ADP) è stabile anche se non viene protonato [Collet et al., 1998].
Il motivo II, separato dal primo da una sequenza aminoacidica di lunghezza variabile, è rappresentato da un residuo di serina o treonina. In questo caso è evidente l’importanza della presenza di un gruppo idrossilico, che ha la funzione di stabilizzare il substrato formando un legame a idrogeno con l’ossigeno del gruppo fosfato del substrato o, nella L-2-aloacido dealogenasi, con il gruppo carbossilico del substrato [Wang et al., 2001; Morais et al., 2000]. Inoltre l’allineamento delle sequenze mostra che questo motivo, come il motivo I, è preceduto da circa 4-5 residui idrofobici.
Il motivo III contiene una lisina conservata, che forma un ponte salino con l’aspartato nucleofilo e stabilizza le cariche negative del substrato e/o dell’intermedio [Ridder and Dijkstra, 1999]. Tale affermazione è stata confermata in seguito alla realizzazione delle strutture cristalline di alcuni componenti della superfamiglia [Wang et al., 2001; Morais et al., 2000].
Il motivo IV, separato dal precedente da una sequenza aminoacidica di lunghezza variabile, è formato da due residui di aspartato più vicini al C-terminale, che hanno la funzione di coordinare il catione bivalente Mg2+ nel sito attivo. Nelle dealogenasi, che non sono enzimi Mg2+-dipendenti, il primo aspartato è sostituito con una serina che interagisce con l’aspartato nucleofilo; il secondo residuo, altamente conservato, è invece responsabile dell’attivazione della molecola d’acqua che idrolizza l’intermedio covalente [Morais et al., 2000].
Struttura tridimensionale: il Core domain e il Cap domain. Conoscendo la sequenza aminoacidica di una proteina è possibile ipotizzare la sua struttura secondaria; quella prevista per i membri della superfamiglia delle HAD mostra che i motivi identificati con l’allineamento delle sequenze sono compatibili con gli elementi strutturali che sono stati determinati sperimentalmente per la dealogenasi e per alcuni degli altri membri. I residui conservati sembrano essere giustapposti nello spazio e le loro posizioni suggeriscono un diretto intervento nella catalisi [Aravind et al., 1998].
Attualmente è conosciuta la struttura tridimensionale di almeno uno dei componente delle 5 sottofamiglie. La struttura cristallina rivela che ognuno di questi enzimi è formato da un α/β core domain altamente conservato e nel quale è localizzato il sito attivo formato da quattro loops che contengono i motivi sovracitati [Lahiri et al., 2004]. Questo dominio, simile al motivo di Rossmann, è formato da sei β-foglietti paralleli circondati da sei α-eliche; le funzioni principali di questa struttura sono quelle di legare, orientare e attivare il gruppo fosforico del substrato e il gruppo che esegue l’attacco nucleofilo [Lu et al., 2005].
Gli enzimi della superfamiglia delle HAD vengono raggruppati in tre sottofamiglie (I-III) in base alla presenza e alla localizzazione di un secondo dominio, denominato cap domain, che agisce come un cappuccio sopra il sito attivo del core domain [Morais et al., 2000; Zhang et al., 2002]. La sottofamiglia I possiede un piccolo cap domain formato da più α-eliche localizzato tra i loops 1 e 2 del core domain; la sottofamiglia II ha un grande dominio a β-sandwich inserito tra i loops 2 e 3 [Shin et al., 2003]; la sottofamiglia III è priva del cap domain (Figura 1-E).
Figura 1-E: LE TRE SOTTOFAMIGLIE DELLA SUPERFAMIGLIA DELLE HAD: a) sottofamiglia I b) sottofamiglia II c) sottofamiglia III (da Allen and Dunaway-Mariano, 2004).
La funzione del cap domain è quella di riconoscere il substrato e chiudere il sito attivo al solvente. Nella fosfoacetaldeide idrolasi [Morais et al., 2000; Zhang et al., 2002], nella β-fosfoglucomutasi [Lahiri et al., 2002] e nella fosfoserina fosfatasi [Wang et al., 2002], componenti della sottofamiglia I, è stato osservato che il cap domain, collegato al core domain da due segmenti peptidici solvatati, si muove come un corpo rigido legato ad un perno. Gli studi cristallografici a raggi-X di questi tre enzimi hanno infatti rivelato la presenza di due stati conformazionali, funzionalmente molto importanti, che differiscono nelle posizioni del cap domain e del core domain. Nella conformazione “cap-chiuso” il cap domain si trova in stretto contatto con il sito attivo del core domain, nella conformazione “cap-aperto” la distanza tra i due domini permette l’ingresso del solvente nel sito attivo facilitando il legame con il substrato ma anche il rilascio del prodotto.
Gli enzimi della sottofamiglia II possono essere a loro volta distinti in due gruppi (tipo A e tipo B) sulla base di alcune differenze presenti a livello delle strutture secondarie del cap domain. Un recente studio eseguito su un componente della sottofamiglia II tipo B, ha dimostrato che anche per questo enzima si verifica un movimento dei domini per raggiungere una conformazione chiusa, suggerendo una analoga funzione a quella proposta per la sottofamiglia I. Nelle prime due sottofamiglie il substrato viene quindi incapsulato tra i due domini, infatti, gli enzimi che fanno parte di questi due gruppi, riconoscono dei substrati di piccole dimensioni; al contrario i membri della sottofamiglia III, essendo privi di questo dominio accessorio, hanno il sito attivo aperto al solvente e possono agire su substrati di maggiori dimensioni [Allen and Dunaway-Mariano, 2004; Lu et al., 2005].
elica-giro-elica (elica2-elica-giro-elica3); il segmento compreso tra le due eliche è chiamato loop 5 (o loop della specificità di substrato) e comprende dei residui che nella conformazione chiusa entrano nel sito attivo e partecipano al legame del substrato o alla catalisi. Nel passaggio tra gli stati funzionali “cap-aperto” e “cap-chiuso” il loop 5 mantiene la sua conformazione ma funge da elemento di giunzione tra il cap e il core domain; il sito catalitico è quindi composto dai quattro loops del core domain e dal loop 5 del cap domain. [Lahiri et al., 2004]. La sottofamiglia II sembra presentare delle differenze rispetto a questo gruppo, perché Lu e coll. hanno recentemente individuato la presenza di due loops conservati, che contengono dei residui implicati nel legame del substrato e che entrano nel sito attivo [Lu et al., 2005].
All’interno della sottofamiglia I una caratteristica comune del loop 5 è la presenza di un residuo conservato di glicina, localizzato verso l’estremità N-terminale della forcina tra l’elica 2 e 3 del motivo strutturale del cap domain (Figura 1-F). La conservazione di questa glicina, l’aminoacido con il minor ingombro sterico, è probabilmente legata alla posizione occupata da questo residuo perché il ripiegamento che si viene a creare ha bisogno di aminoacidi che consentono libertà di movimento; anche i residui che si trovano a monte e a valle della glicina, all’interno di una data sottofamiglia, sembrano essere abbastanza conservati. Gli studi cinetici e cristallografici hanno sottolineato il ruolo essenziale della glicina per l’efficienza del cap domain perché questo residuo è responsabile della conformazione e della flessibilità del loop 5, necessarie per il corretto funzionamento dell’attività catalitica [Lahiri et al., 2004].
Figura 1-F: ALLINEAMENTO DELLA SEQUENZA AMINOACIDICA DEL MOTIVO ELICA-GIRO
-ELICA DEL CAP DOMAIN DI ALCUNI MEMBRI DELLA SUPERFAMIGLIA DELLE HAD.
Phn: fosfonatasi; BPGM: β-fosfoglucomutasi; LipP: epossido idrolasi; PserP: fosfoserina fosfatasi; dNP: deossiribo-nucleotidasi (da Lahiri et al., 2004).
Recentemente Rinaldo-Matthis e coll. hanno ottenuto la struttura cristallina della 5’(3’)-deossiribonucleotidasi mitocondriale e hanno potuto dimostrare la relazione di questo enzima con la superfamiglia delle HAD. Nella struttura primaria sono stati infatti individuati i motivi aminoacidici conservati (I-IV) coinvolti nel meccanismo catalitico e anche il motivo S, contenuto nel loop 5, responsabile della specificità di substrato. In
quest’ultimo motivo è stata anche rintracciata la glicina conservata ed è stato proposto che essa possa svolgere una funzione di perno e conferire una certa flessibilità a questa regione permettendo al substrato di entrare nel sito attivo. L’allineamento delle sequenze primarie della md-N e delle altre nucleotidasi intracellulari suggerisce la presenza di una glicina in ognuno di questi enzimi [Rinaldo-Matthis et al., 2002].
La II: un nuovo membro della superfamiglia delle HAD. La scoperta che la cN-II, durante la catalisi, si fosforila sul primo aspartato (D52) della sequenza DMDYT, ha fatto pensare che la nucleotidasi appartenesse alla superfamiglia delle aloacido dealogenasi, in quanto una sequenza simile (motivo I) è presente in tutti i membri della superfamiglia [Allegrini et al., 2001]. L’allineamento delle sequenze primarie della cN-II e delle altre nucleotidasi intracellulari con quelle dei componenti della superfamiglia delle HAD ha rivelato la presenza dei quattro motivi in ognuna di queste nucleotidasi.
Nel 2002 la cristallizzazione della 5’(3’)-deossiribonucleotidasi mitocondriale ha rafforzato le ipotesi formulate, perché, come negli altri membri della superfamiglia, le quattro sequenze consenso si organizzano su anse che si ritrovano nel sito attivo e ogni monomero dell’enzima è formato da due domini: uno simile al motivo di Rossmann e l’altro più piccolo costituito da quattro α-eliche [Rinaldo-Matthis et al., 2002].
Sebbene non sia stato ancora possibile cristallizzare la cN-II a causa della sua scarsa solubilità, gli esperimenti di mutagenesi sito-diretta sono stati utili per indagare sul ruolo dei residui conservati. Le modificazioni dei parametri cinetici causate da mutazioni conservative e non conservative suggeriscono che questi aminoacidi rivestano ruoli importanti e specifici a livello del sito attivo. In generale i risultati ottenuti hanno confermato che gli aminoacidi conservati svolgono funzioni simili a quelle svolte negli altri enzimi della superfamiglia; ciò significa che la cN-II presenta un meccanismo catalitico molto simile a quello degli altri membri. Unendo i dati ottenuti da questi esperimenti e le informazioni emerse dai precedenti studi eseguiti sulla fosfoserina fosfatasi, la ATP-asi e la mdN è stato costruito un modello ipotetico del sito attivo della cN-II (Figura 1-G), in cui sono state riportate le probabili disposizioni degli aminoacidi e le loro interazioni con il Mg2+ e il substrato [Allegrini et al., 2004].
I due residui di aspartato del motivo I (DMDYT) sono entrambi indispensabili per l’attività dell’enzima perché una qualunque mutazione di questi aminoacidi provoca la totale perdita dell’attività. Il residuo D52 è responsabile della formazione dell’intermedio, mentre il residuo D54 è coinvolto nella stabilizzazione dell’estere e nella coordinazione dello ione Mg2+. La treonina 56 (T56) ha la funzione di orientare l’aspartato nucleofilo
mediante un legame a idrogeno. Il motivo II è costituito dal solo aminoacido treonina 249, che stabilizza il substrato grazie ad un legame a idrogeno con un atomo di ossigeno del fosfato; il motivo III rappresentato dalla lisina 292 (K292) forma un ponte salino con l’aspartato nucleofilo al fine di correggere il suo orientamento, mentre i due aspartati (D351 e D 356) del motivo IV sono direttamente responsabili della coordinazione dello ione Mg2+ [Allegrini et al., 2004].
Motivo IV
Motivo I Figura 1-G: Modello del sito attivo della cN-II con i residui aminoacidici dei quattro motivi conservati implicati nel meccanismo catalitico (da Allegrini et al., 2004).
Motivo IV
Motivo II Motivo I Motivo I
Motivo III
Per quanto riguarda la struttura tridimensionale attualmente si ritiene che la cN-II appartenga alla sottofamiglia I, perché la regione aminoacidica compresa tra il motivo I e II appare molto più ampia e ricca di residui idrofobici rispetto a quella compresa tra il motivo II e III; le subunità della cN-II sarebbero quindi formate da un α/β core domain e da un cap domain ed il sito attivo risulterebbe localizzato in una tasca idrofobica posta nell’interfaccia tra i due domini.
1.2.4 MECCANISMO DI REAZIONE
I principali substrati della cN-II sono i nucleosidi monofosfato IMP e GMP e i loro deossiderivati. Questa nucleotidasi, come la cN-III, oltre ad avere una attività fosfatasica, possiede anche la capacità di catalizzare una reazione di fosfotransferasi.
Durante l’idrolisi di un nucleoside monofosfato, l’enzima si fosforila e rilascia come primo prodotto, il nucleoside (attività nucleotidasica); il fosfato legato all’enzima può essere idrolizzato da una molecola d’acqua (attività fosfatasica o idrolizzante) oppure può
essere trasferito ad un opportuno nucleoside accettore (attività fosfotransferasica, PHT), (Figura 1-H).
I primi ricercatori che ipotizzarono questo meccanismo di reazione, che procede attraverso la formazione di un intermedio enzima-fosfato, proposero tale modello basandosi solo sui dati cinetici da loro osservati [Worku & Newby, 1982]. L’esistenza dell’intermedio fosforilato è stata dimostrata direttamente molti anni dopo, utilizzando un substrato radioattivo, il [32P]IMP, che può essere idrolizzato dalla cN-II. In questo saggio dopo una breve incubazione della miscela di reazione, l’aggiunta di una soluzione di SDS blocca l’intermedio e la proteina fosforilata, dopo corsa elettroforetica, può essere messa in evidenza su lastra autoradiografica [Baiocchi et al., 1996].
H
2O
(d)N
1P
i+ E
(d)
N
1MP + E
E
•P
(d) N
2MP + E
(d) N
2Figura 1-H: MECCANISMO DI REAZIONE DELLA 5’-NUCLEOTIDASI CITOSOLICA II (CN-II).
(d)N1MP: (deossi)nucleoside 5’-monofosfato 1, donatore del fosfato; (d)N1: (deossi)nucleoside 1, primo prodotto della reazione; (d)N2: (deossi)nucleoside 2, accettore del fosfato; Pi: fosfato inorganico; (d)N2MP: (deossi)nucleoside 5’-monofosfato 2, prodotto della reazione fosfotransferasica.
Per l’attività catalitica è necessaria la presenza dei cationi bivalenti Mg2+, cofattore fisiologico dell’enzima, ma la stessa funzione può essere svolta anche da Co2+, Mn2+e Ni2+, seppur in modo meno efficiente; altri cationi metallici come il Ca2+, lo Zn2+, il Cd2+ o il Pb2+ rendono invece l’enzima inattivo [Itoh, 1993].
Grazie alla duplice attività mostrata dalla cN-II, questa nucleotidasi è stata definita un “ENZIMA BIFUNZIONALE”. Nella reazione fosfotransferasica i principali accettori di fosfato
sono i substrati naturali inosina e idrossinosina; poiché l’enzima si è dimostrato capace di fosforilare anche degli analoghi purinici, utilizzati nella terapia antitumorale e antivirale, è stato proposto che la cN-II possa avere un ruolo fondamentale nel processo di attivazione/deattivazione di alcuni pro-farmaci (cfr. capitolo 1.3 pag. 30). In assenza di un nucleoside disponibile e appropriato l’enzima si comporta però come una fosfatasi; pertanto la somma dell’attività fosfatasica e fosfotransferasica corrisponde all’attività nucleotidasica, che rappresenta l’attività complessiva.
L’attività nucleotidasica può essere stimata misurando la quantità di nucleoside prodotto e rilasciato dall’enzima in presenza di un nucleoside accettore di fosfato; la PHT invece viene stimata misurando la quantità di nucleoside monofosfato formato dal nucleoside accettore; infine l’attività fosfatasica può essere calcolata sia in modo indiretto, sottraendo la PHT dall’attività complessiva, che in modo diretto, misurando la concentrazione di fosfato rilasciato [Banditelli et al., 1996]. Se la base purinica del nucleotide, utilizzato come donatore di fosfato, è diversa da quella del nucleoside accettore, è possibile seguire contemporaneamente l’attività nucleotidasica e quella fosfotransferasica nella stessa miscela di saggio. Questo metodo ha permesso, ad esempio, di studiare gli effetti esercitati dagli attivatori e dagli inibitori sulla velocità delle due reazioni [Allegrini et al., 1993].
Nella reazione catalizzata dalla cN-II la tappa limitante è rappresentata dall’idrolisi dell’intermedio fosforilato, in quanto è stato osservato che l’aggiunta di un opportuno accettore del fosfato causa l’aumento della velocità della reazione nucleotidasica. Poiché il trasferimento del fosfato ad un nucleoside accettore avviene più rapidamente rispetto alla reazione di idrolisi, è stato anche ipotizzato che il sito attivo dell’enzima possa avere una natura parzialmente idrofobica che favorisce l’ingresso di molecole organiche meno polari dell’acqua [Pesi et al., 1994; Banditelli et al., 1996].
Le prime informazioni sui residui aminoacidici coinvolti nel sito attivo sono state ottenute saggiando l’effetto inibitorio di alcuni composti. L’inattivazione della cN-II ad opera del reagente K di Wood-ward (WRK), capace di modificare i residui di aspartato e glutammato, ha fatto ipotizzare il coinvolgimento di un residuo carbossilico nell’attività catalitica. Mediante ulteriori indagini è stato dimostrato che l’aspartato 52 (Asp-52) è il residuo che viene fosforilato durante la reazione enzimatica ed esperimenti di mutagenesi sito-diretta hanno confermato il ruolo fondamentale di questo aminoacido nella catalisi. L’Asp-52 è il primo aspartato del motivo DMDYT, altamente conservato tra le diverse specie e condiviso dagli altri componenti della famiglia delle 5’-nucleotidasi. Questa scoperta ha permesso di considerare la possibile appartenenza della cN-II ad una grande
famiglia di enzimi, la superfamiglia delle aloacido dealogenasi (HAD). Nonostante la bassa omologia di sequenza tra la cN-II e i membri della superfamiglia delle HAD, questi enzimi condividono una attività fosfotransferasica-fosfatasica, una dipendenza dal catione bivalente Mg2+, un meccanismo di reazione che procede attraverso la formazione di un intermedio enzima-fosfato e la presenza, vicino all’N-terminale, del motivo DXDX(T/V) fiancheggiato da residui idrofobici che, durante la catalisi, viene fosforilato sul primo aspartato [Allegrini et al., 2001].
L’attività fosfotransferasica della cN-II su substrati naturali, attualmente è stata dimostrata solo in vitro, quindi resta da chiarire se l’enzima in condizioni fisiologiche abbia, oltre ad un ruolo catabolico, anche un ruolo anabolico. Per tale scopo sarebbe utile individuare gli eventuali residui aminoacidici che rendono la cN-II e la cN-III, a differenza delle altre nucleotidasi, capaci di catalizzare il trasferimento del fosfato su una molecola accettrice.
1.2.5 REGOLAZIONE
L’attività della cN-II è controllata da un complesso meccanismo di regolazione comprendente vari fattori che possono influenzare l’attività enzimatica totale oppure il rapporto 5’N/PHT, che indica quanto l’attività fosfotransferasica incida sull’attività nucleotidasica. La totale comprensione dei meccanismi regolatori di questo enzima potrebbe chiarire gli aspetti non ancora accertati riguardo il ruolo fisiologico della cN-II e aprire una strada verso la progettazione di farmaci più efficaci.
I dati fino ad oggi disponibili indicano che l’attività della cN-II può essere modulata da molecole effettrici, come l’ATP, l’ADP e il 2,3-bisfosfoglicerato (BPG), e inibitrici, come il fosfato inorganico (Pi); la maggior parte delle conoscenze riguardo questi modulatori allosterici derivano da studi cinetici, perciò non si conosce quali siano le modificazioni strutturali a cui la proteina è soggetta e quali siano i residui aminoacidici coinvolti.
Il principale attivatore della cN-II è l’ATP, che mostra una K50 di circa 2 mM, ovvero un valore inferiore alla concentrazione cellulare presente in condizioni fisiologiche (4.5 mM). L’effetto esercitato dall’ATP si manifesta fondamentalmente con un incremento della Vmax sia della attività nucleotidasica che della attività fosfotransferasica; nonostante l’attivatore favorisca maggiormente quest’ultima reazione, l’analisi dell’efficienza catalitica ha indicato che in presenza di ATP l’enzima agisce prevalentemente come una idrolasi [Pesi et al., 1994].
Lo stesso effetto attivante dell’ATP può essere ottenuto aggiungendo nella miscela di reazione alte concentrazioni di sali, come l’NaCl, il KCl o LiCl oppure addizionando alcuni acidi carbossilici come l’acetato, il malato, il fumarato o il succinato [Itoh, 1993]. L’azione attivatrice e stabilizzatrice del KCl, ottenuta con concentrazioni maggiori di 0.3 M, può essere attribuita alla forza ionica esercitata dal sale; inoltre in presenza dell’ATP è stato notato che il KCl non provoca alcuna variazione dell’attività catalitica [Tozzi et al., 1991; Pesi et al., 1994; Spychala et al., 1999].
L’inibitore per eccellenza della cN-II è il fosfato inorganico (Pi), che esplica la sua azione modulatrice a concentrazioni fisiologiche (5 mM). L’inibizione esercitata dal Pi sulla 5’-nucleotidasi si manifesta con un incremento del valore delle Km e, in particolare, è stata osservata una più marcata riduzione dell’affinità nei confronti dell’IMP che non dell’inosina. Studi cinetici hanno dimostrato che i due nucleotidi adenilici, ADP e ATP, sono in grado di rimuovere l’inibizione esercitata dal fosfato sull’enzima, ma con una diversa efficienza; è stato infatti calcolato che livelli fisiologici di ATP riescono a rimuovere l’85% dell’inibizione provocata da una concentrazione di Pi pari a 5 mM, mentre la stessa concentrazione di ADP riesce ad eliminare solo il 40% dell’inibizione. Con ciò possiamo concludere che il ruolo dell’ADP è simile a quello dell’ATP, ma il potere attivante di quest’ultimo risulta molto più forte [Pesi et al., 1994].
In condizioni fisiologiche l’enzima si trova probabilmente sotto il controllo dell’ATP, come attivatore, e del Pi, come inibitore; l’analisi dell’efficienza catalitica della cN-II in presenza di 4.5 mM di ATP e 5 mM di Pi ha indicato che l’enzima agirebbe quasi esclusivamente come fosfotransferasi, se non fosse per l’alta Km dell’inosina (1 mM) che rappresenta il fattore limitante. Questa osservazione è stata confermata con lo studio delle variazioni delle due attività enzimatiche in funzione della carica energetica. A valori fisiologici di carica energetica e a concentrazioni millimolari di fosfato la possibilità che la cN-II operi in vivo una fosforilazione di nucleosidi sembra dipendere solo dalla disponibilità di un opportuno accettore di fosfato. [Pesi et al., 1994].
Un altro attivatore della cN-II è il 2,3-bisfosfoglicerato (BPG). La K50 di questo composto è stata stimata intorno a 1 mM, quindi si ritiene che il BPG svolga un ruolo fisiologico come modulatore solo negli eritrociti dove raggiunge una concentrazione sufficientemente elevata. Ulteriori studi cinetici, messi a punto per svelare le proprietà regolatrici della cN-II, hanno permesso di scoprire l’esistenza di un effetto sinergico tra l’ADP e il BPG; quando questi due metaboliti sono presenti insieme nella miscela di saggio le loro costanti di attivazione si riducono, dimostrando che i due attivatori vanno
presumibilmente ad occupare siti diversi dell’enzima. Un parametro cinetico che questi due composti riescono a modificare significativamente è la Vmax dell’attività PHT: quando i due effettori sono presenti separatamente la Vmax risultata aumentata di 2-3 volte, quando sono presenti contemporaneamente viene aumentata di 5 volte [Pesi et al., 1996]. L’osservazione che anche il BPG facilita il trasferimento del fosfato ad un nucleoside accettore piuttosto che all’acqua, suggerisce un possibile ruolo di questo composto nella regolazione della fosforilazione di analoghi purinici.
L’attività enzimatica non è soggetta solo ad un controllo di tipo allosterico, ma viene influenzata anche dall’ambiente di reazione. Le proprietà cinetiche della cN-II, come quelle di altri enzimi, sono infatti dipendenti dal grado di acidità della soluzione; è stato però sorprendente scoprire che le due attività possedute dall’enzima abbiano pH ottimali diversi: l’attività fosfatasica richiede un pH lievemente acido (6.5), mentre l’attività fosfotransferasica ha bisogno di un pH più vicino a quello fisiologico (7.2). Questi risultati indicano che il trasferimento del fosfato ad un accettore sarebbe favorito, oltre che dalla carica energetica, anche dal pH cellulare [Banditelli et al., 1996].
Altri parametri importanti che possono influenzare l’attività della cN-II sono le condizioni ossido-riduttive dell’ambiente di reazione. L’aggiunta dell’agente riducente ditiotreitolo (DTT) alla miscela di saggio, si è dimostrata necessaria per ottenere una completa funzionalità dell’enzima, anche quando la proteina è stata appena purificata. Per confermare il ruolo dell’agente riducente sono stati eseguiti degli esperimenti anche in ambiente ossidante, ovvero in presenza di 5,5’-ditiobis-2-acido nitrobenzoico (DTNB) [Baiocchi et al., 1996]; in tali condizioni la cN-II perde attività, ma l’aggiunta del DTT è sufficiente a far sì che l’enzima possa recuperare l’attività persa. Questi dati suggeriscono che nel meccanismo catalitico siano coinvolti, in modo diretto o indiretto, dei residui di cisteina i cui gruppi sulfidrilici (gruppi –SH) devono essere presenti nello stato ridotto [Allegrini et al., 2004].
Dopo la scoperta dell’esistenza di due forme (A e B) della cN-II purificata dal timo di vitello, Pesi e coll. hanno identificato e caratterizzato queste due proteine, che si differenziano, non solo per proprietà elettroforetiche e cromatografiche, ma anche per caratteristiche regolatrici. La forma A di peso molecolare maggiore (59 KDa) è attivata dal BPG, verso il quale possiede una elevata affinità, e dall’ATP o dall’ADP che competono per lo stesso sito di legame. La forma B ha un peso molecolare di 54 Kda, possiede un sito di legame specifico per ogni effettore (ATP, ADP e BPG) ed è attivata in modo sinergico dal BPG e dall’ADP [Pesi et al., 1998].
La conferma dell’esistenza di queste due forme è stata ottenuta qualche anno dopo grazie all’utilizzo dell’enzima ricombinante bovino. Quando la proteina ricombinante è stata conservata a 4 °C per un periodo di tempo abbastanza lungo oppure quando appena purificata è stata incubata overnight a 25 °C con una proteasi (trombina), è stato possibile ottenere le stesse due forme dell’enzima. Poiché la quantità della forma B aumenta a discapito di quella ad alto peso molecolare, è stato proposto che un evento proteolitico intervenga a rimuovere una piccola porzione dalla sequenza peptidica [Allegrini et al., 2004]. Le precedenti conoscenze riguardo la dipendenza della cN-II dalle condizioni ossido-riduttive dell’ambiente di reazione, hanno suggerito ai ricercatori di testare la sensibilità di queste due forme all’agente riducente DTT [Allegrini et al., 2004]. I risultati di questa indagine hanno dimostrato che mentre la forma B di 54 KDa è completamente attiva anche in assenza del DDT, la forma A è molto sensibile al composto, necessario affinché l’enzima possa essere totalmente attivo.
Infine la caratterizzazione strutturale della forma A ha condotto alla scoperta dell’esistenza di un ponte disolfuro che si forma per ossidazione spontanea tra le cisteine 175 e 547 e che il DTT è in grado di rompere; evidentemente questi due residui, lontani nella sequenza primaria, durante il ripiegamento terziario del polipeptide vengono a trovarsi vicini ed esposti sulla superficie in modo da essere sensibili alle condizioni ossido-riduttive dell’ambiente. L’analisi strutturale della forma B dell’enzima ha invece dimostrato che il taglio proteolitico della trombina elimina un frammento di peptide all’estremità C-terminale che va dal residuo aminoacidico 526 al residuo aminoacidico 560 [Allegrini et al., 2004]. L’osservazione che questa forma non possiede la cisteina 547 coinvolta nella formazione del ponte disolfuro, può spiegare il motivo per cui le due proteine mostrano una sensibilità diversa verso il DTT.
Tutte queste considerazioni hanno spinto ad introdurre la regione carbossiterminale della proteina nel meccanismo di regolazione della cN-II. La presenza simultanea delle due forme dell’enzima potrebbe, ad esempio, rendere la cN-II meno suscettibile alle variazioni fisiologiche della carica energetica, ma soprattutto meno sensibile alle fluttuazioni dello stato redox della cellula [Allegrini et al., 2004].
La coda C-terminale era già stata sospettata di essere coinvolta nella modulazione dell’attività enzimatica. Nel 1999 Spychala e coll. avevano verificato che l’eliminazione degli ultimi 13 aminoacidi, ovvero solo residui di acido aspartico e glutammico, riduceva l’efficienza catalitica. Inoltre gli esperimenti di gel filtrazione mostravano che l’ATP stimolava l’associazione delle subunità a formare polimeri (non solo tetrametri ma anche
ottametri), mentre il Pi esercitava l’effetto opposto e quindi induceva la dissociazione degli aggregati. Dalle loro analisi avevano potuto concludere che la capacità di costruire polimeri di grandi dimensioni era legata alla presenza della coda C-terminale [Spychala et al., 1999]. Queste considerazioni sulla forma attiva dell’enzima sono ancora oggetto di discussioni e solo degli studi cristallografici potrebbero risolvere definitivamente questo interrogativo.
1.2.6 RUOLI FISIOLOGICI
L’elevata conservazione filogenetica della cN-II, la sua distribuzione ubiquitaria e il suo complesso meccanismo di regolazione sono indice della estrema importanza che questo enzima riveste nel metabolismo dei nucleotidi purinici. La funzione più accreditata della cN-II è il controllo della concentrazione intracellulare dell’IMP, sintetizzato ex novo oppure prodotto in seguito all’aumentata degradazione dell’ATP. Grazie all’azione catabolica dell’enzima, in presenza di un’alta carica energetica l’IMP in eccesso viene idrolizzato ad inosina, la quale viene scissa in ribosio-1-fosfato e ipoxantina e quest’ultima convertita in acido urico (Figura 1-I).
Negli animali uricotelici come gli uccelli, un aumento dell’apporto proteico provoca un incremento dell’attività della cN-II a livello del fegato, perché l’eccesso di azoto derivante dalla degradazione degli aminoacidi viene eliminato sotto forma di acido urico passando attraverso la formazione dell’IMP [Itoh et al., 1972; 1974; 1992]. Nell’uomo una dieta iperproteica causa un aumento dell’escrezione di acido urico; quindi sembra plausibile che anche nei mammiferi la cN-II defosforili l’IMP in eccesso, incrementando la produzione del prodotto finale della degradazione delle purine [Bien et al., 1953].
Quando si verifica una caduta della carica energetica cellulare l’azione combinata della AMP deaminasi e della adenilato chinasi cerca di mantenere alto il rapporto ATP/ADP. Dall’azione della AMP deaminasi viene prodotto IMP, come mostrato in Figura 1-E. In queste circostanze l’alta concentrazione di fosfato e la ridotta reperibilità dell’ATP rendono la cN-II poco attiva e incapace di idrolizzare l’IMP, evitando così la perdita del nucleo purinico sotto forma di inosina o ipoxantina diffusibili. Nel caso in cui la caduta della carica energetica sia moderata è probabile che l’enzima defosforili l’IMP, perché la disponibilità dell’attivatore ATP e lo scarso accumulo dell’inibitore Pi rendono l’enzima ancora attivo [Itoh, 1993].
PRPP
ATP
13Xao
IMP
ADP
5’ N
6 4 Ino 1AS
2GMP
XMP
IMP
AMP
3
5’ N
5’ N
Pi NH3 8 4 4
9 4 PPi Pi
Guo
INO
ADO
7
11 PRPP Pi NH3 5
R-1-P R-1-P
Gua
Hyp
10 12
Xn
1 Adenilosuccinato sintetasi 2 Adenilsuccinato liasi 3 AMP deaminasi 4 5’-nucleotidasi citosolica II 5 Purino nucleoside fosforilasi 6 Adenilato chinasi 7 Adenosina deaminasi 9 I rasi e fosforilasi 13 Nucleoside-difosfato chinasi 8 Adenosina chinasi poxantina guanina fosforibosiltransfe 10 Xantina ossidasi 11 Nucleosid 12 Guanasi10
Acido Urico
Figura 1-I: VIA METABOLICA DI SINTESI, RECUPERO E DEGRADAZIONE DELL’IMP.
PRPP: 5-fosforibosil-1-pirofosfato; AS: adenilosuccinato; Ado: adenosina; INO: inosina; Hyp: ipoxantina; XMP: xantosina monofosfato; Guo: guanosina; Gua: guanina; Xn: xantina.
Una bassa ma consistente attività della cN-II è stata rilevata anche negli eritrociti dove è presente il 2,3-bisfosfoglicerato, attivatore dell’enzima ma anche importante effettore allosterico dell’emoglobina. Quando la pressione parziale di ossigeno è bassa e il pH è acido, il BPG si lega alla forma deossigenata dell’emoglobina e non può attivare la cN-II, provocando un accumulo di IMP negli eritrociti [Berman et al., 1988]. Al contrario quando l’ambiente è neutro e la pressione parziale dell’ossigeno è alta, il BPG si associa all’enzima che, stimolato dall’attivatore, riesce a defosforilare l’IMP producendo inosina; quest’ultima e il prodotto della sua degradazione, l’ipoxantina, possono essere rilasciati all’esterno. Alla luce di queste osservazioni è stato proposto che gli eritrociti, grazie all’azione della cN-II, possano essere implicati nel trasporto dell’ipoxantina (accumulata nel citosol sotto forma di IMP) dai tessuti anossici a quelli ben ossigenati.
Nei tessuti con una attiva sintesi del DNA o con una rapida velocità di turnover degli acidi nucleici, come i testicoli e la milza ma anche i linfoblasti e le cellule tumorali, si registra un’alta attività della cN-II, che suggerisce un possibile ruolo anabolico dell’enzima in aggiunta a quello catabolico già accertato. Grazie all’attività fosfotransferasica, la cN-II potrebbe trasferire il fosfato dall’IMP alla deossi-inosina e produrre dIMP, che può essere successivamente convertito in dAMP e dGMP [Tozzi et al., 1991]. Questa reazione inserirebbe l’enzima nel processo di sintesi dei dNTPs purinici, utilizzati per la sintesi degli acidi nucleici nelle cellule proliferanti.
Recentemente è stato osservato che in vitro l’attività fosfotransferasica della cN-II potrebbe essere coinvolta anche nella via di recupero della xantosina, che conduce alla formazione dei nucleotidi della guanina. In questa ipotetica via anabolica la cN-II trasferisce il fosfato dall’IMP alla xantosina (Xao) per produrre XMP e, in presenza di glutammina, quest’ultima viene convertita in GMP. La fosforilazione della Xao, che è fortemente dipendente da ATP e 2,3-bisfosfoglicerato, attivatori allosterici della cN-II, e dal catione bivalente Mg2+, consentirebbe di evitare la conversione NAD+-dipendente dell’IMP a XMP catalizzata dall’enzima IMP deidrogenasi. Come avviene per altri nucleotidi purinici, il recupero della xantosina è particolarmente attivo nel periodo di riperfusione perché durante l’anossia l’accumulo di IMP, derivante dal catabolismo dell’ATP, fornisce il substrato per l’attività fosfotransferasica [Barsotti et al., 2005].
1.3 INTERESSI TERAPEUTICI DELLA cN-II
I difetti del metabolismo dei nucleotidi purinici e pirimidinici provocano la comparsa, nell’individuo affetto da tale disfunzione, di un grave quadro clinico che interessa principalmente il sistema nervoso. In alcune sindromi neurologiche con una eziologia non completamente chiara, è stata osservata anche una alterata attività 5’-nucleotidasica che potrebbe essere responsabile di alcuni dei sintomi.
Gli studi sulla cN-II hanno degli obiettivi terapeutici che non si fermano alla ricerca di una cura per le gravi patologie genetiche, ma si prefiggono anche lo scopo di migliorare la terapia antivirale e antitumorale. La 5’-nucleotidasi sembra infatti associata sia alla resistenza nei confronti dei farmaci utilizzati per queste patologie, ma anche ad una loro attivazione inappropriata, responsabile di un effetto tossico nelle cellule sane.
1.3.1 SINDROMI NEUROLOGICHE
La sindrome di Lesch-Nyhan è una malattia genetica causata da una mutazione X-linked recessiva che provoca la mancata espressione dell’enzima ipoxantina-guanina fosforibosil transferasi (HGPRT), coinvolto nel recupero del nucleo purinico.
Questo errore congenito del metabolismo ha conseguenze devastanti sul comportamento del soggetto e sulla funzionalità di alcuni apparati. I principali sintomi neurologici sono il comportamento autistico, il ritardo mentale, l’aggressività e la tendenza all’automutilazione; inoltre si osserva un ritardo nello sviluppo motorio e successivamente la comparsa di movimenti incontrollabili, spasmi involontari e movimenti distonici. Una delle conseguenze biochimiche dell’assenza dell’enzima HGPRT è l’iperproduzione di acido urico (iperuricemia), che si verifica già dopo i primi mesi di vita e determina la formazione di cristalli di urato, che si depositano soprattutto a livello dei reni e delle articolazioni provocando delle serie infiammazioni [Lesch and Nyhan, 1964]. Poiché i nuclei purinici non possono essere recuperati, si ha un accumulo del fosforibosil pirofosfato (PRPP), co-substrato dell’HGPRT, e un aumento della sintesi de novo dei nucleotidi purinici.
Gli eritrociti e i fibroblasti dei soggetti affetti da questa sindrome mostrano anche un incremento della attività della cN-II, che potrebbe essere collegata con lo sviluppo dei danni neurologici. Il 5’-amminoimidazolo-4-carbossammide-1-ribofuranosil monofosfato